本文主要是介绍Linux系统中已知SRR号如何从NCBI上下载SRA数据到服务器中,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
高通量组学数据目前已经成为生物研究的重要板块,对于一些文章中出现的数据的挖掘尤其是人体数据的再利用也成为探究科学问题的重要前沿组成。通常情况下文章的高通量数据需要上传到NCBI的SRA(Sequence Read Archive)供大家下载学习,而我们也可以通过多种方法对数据进行下载再挖掘。在此介绍一种下载NCBI SRA数据的最佳方法。
首先,我们在下面的网址中进入SRA Toolkit下载的官网。Downloading SRA Toolkit · ncbi/sra-tools Wiki · GitHub ![]() https://github.com/ncbi/sra-tools/wiki/01.-Downloading-SRA-Toolkit
https://github.com/ncbi/sra-tools/wiki/01.-Downloading-SRA-Toolkit
其次选择点击"CentOS Linux 64 bit architecture"下载Linux版本的软件压缩包。
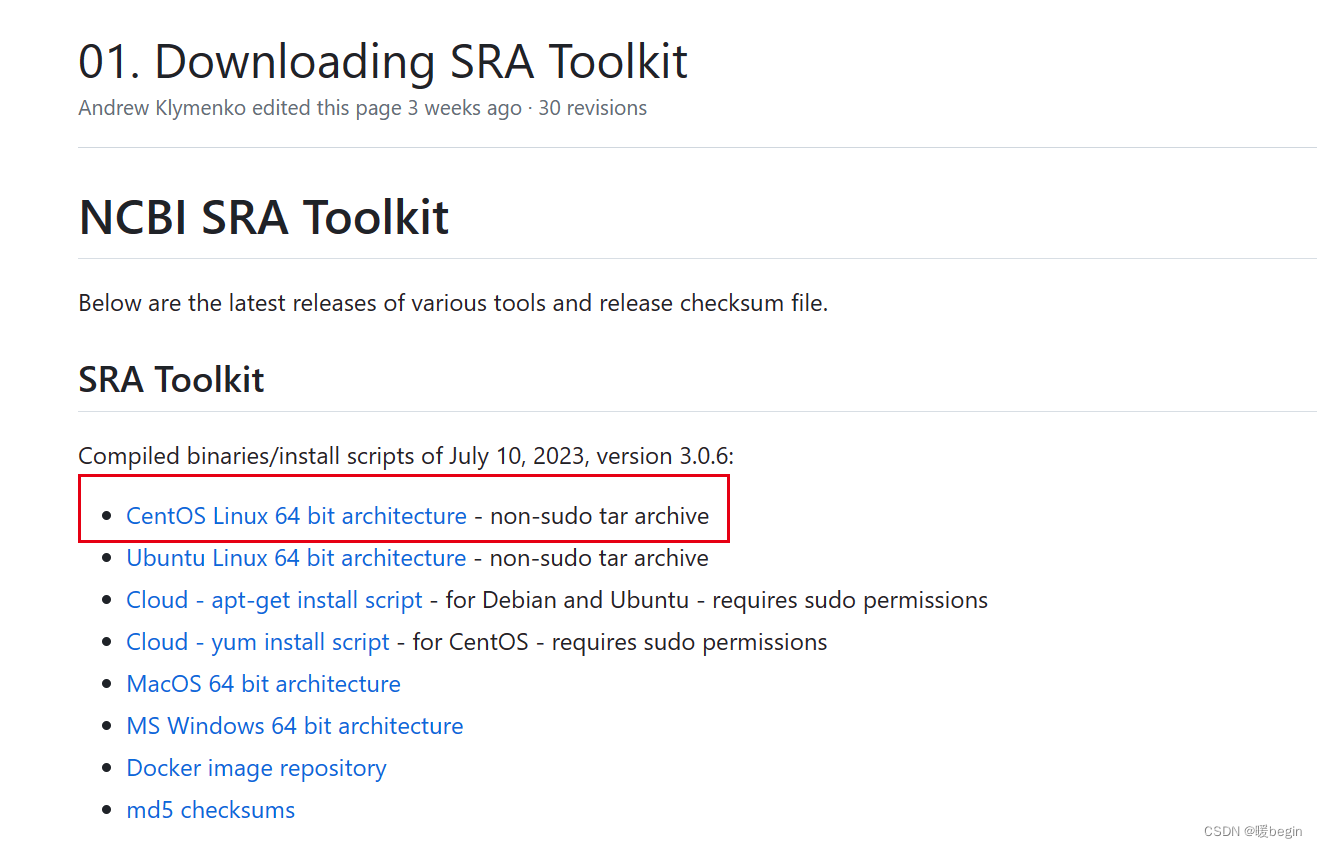
将压缩包下载完成以后,通过Xshell上传到服务器的指定文件夹,我个人一般喜欢建一个software的文件夹用于安装各类软件工具。
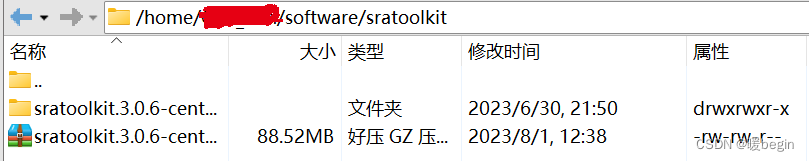
通过“tar -zxvf ”指令对压缩包进行解压。
tar -zxvf sratoolkit.3.0.6-centos_linux64.tar.gz
然后在PATH中加入fastq-dump命令,此时需要编辑Shell配置文件(例如bash的配置文件为~/.bashrc或~/.bash_profile),例如,在bash中使用以下命令打开~/.bashrc:
vi ~/.bashrc
在配置文件中添加以下行,将sra-toolkit的bin目录路径加入到PATH中:
export PATH=$PATH:/home/Guo_Jian/software/sratoolkit/sratoolkit.3.0.6-centos_linux64/bin
按“Esc”再“:wq”保存文件并关闭编辑器。然后为使修改生效,可以重新启动终端,或者在当前终端中运行以下命令:
source ~/.bashrc
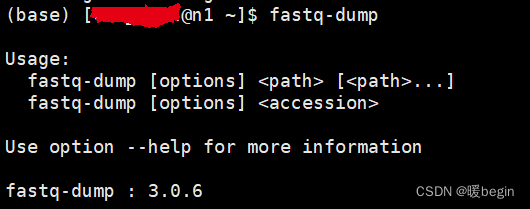
最后可以尝试在终端中运行fastq-dump命令,看看是否成功启动。如果成功,将显示fastq-dump的帮助信息。如果仍然出现问题,请再次检查PATH设置是否正确,并确保在PATH中包含了sra-toolkit的bin目录。如下所示即为安装成功,随后我们便可以开始数据的下载了。
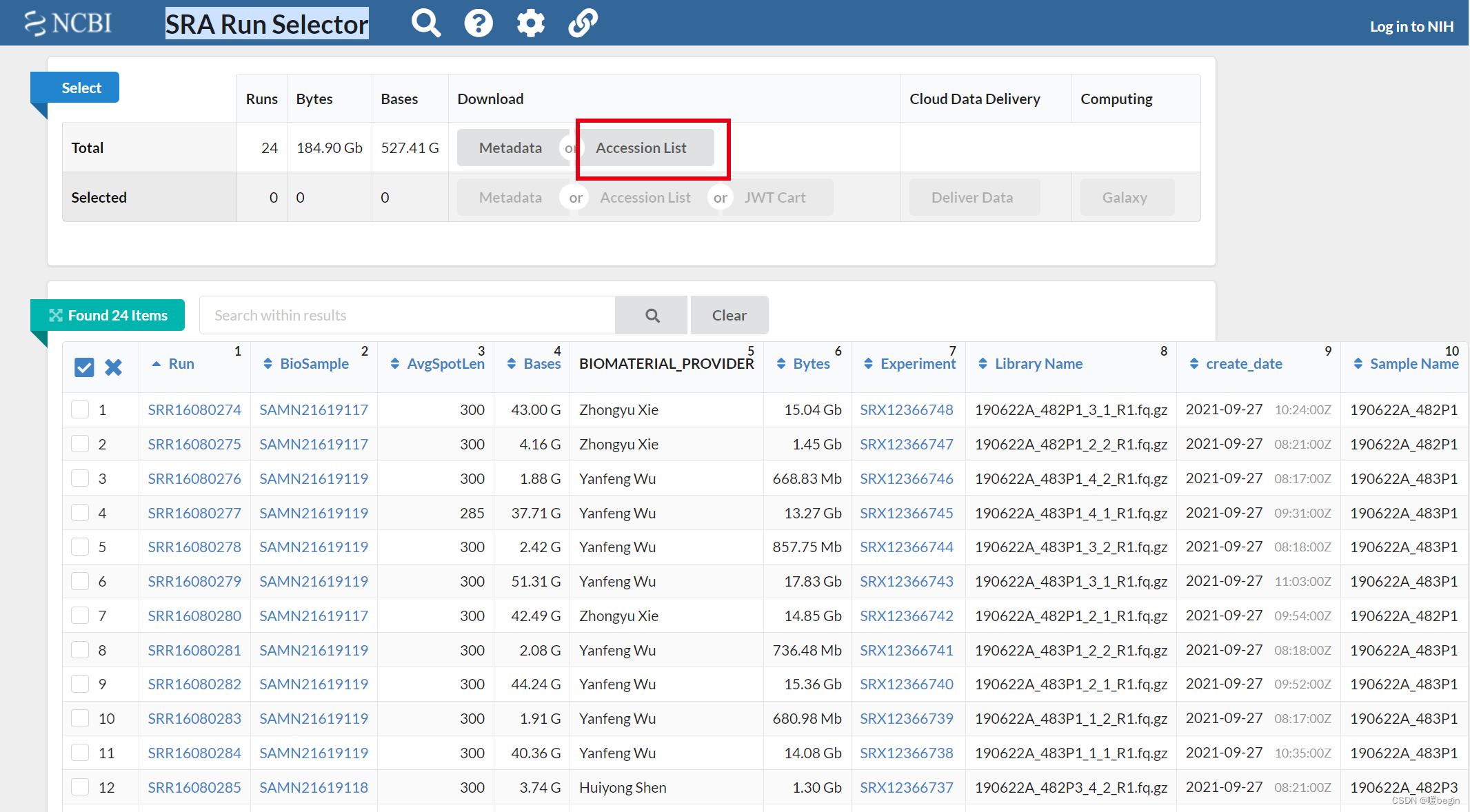
我们在NCBI上找到我们需要的数据的SRA Run Selector界面,点击"Accession List "获得我们所需要的下载的SRR序号List,其文件命名通用为“SRR_Acc_List .txt”:
 可使用如下命令在后台进行批量下载:
可使用如下命令在后台进行批量下载:
nohup prefetch -O . $(<SRR_Acc_List.txt) &下载结束后,可使用如下命令进行批量解压:
for f in *.sra
do
nohup fastq-dump --split-3 $f &
done如此,数据下载和转换就完成了。
这篇关于Linux系统中已知SRR号如何从NCBI上下载SRA数据到服务器中的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







