本文主要是介绍R语言基因功能富集分析气泡图,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
文章目录
- 前言
- 一、基因本体论介绍
- 二、数据获得
- 三、气泡图绘制
- 四、参考
前言
大家好✨,这里是bio🦖。有一位粉丝询问我如何绘制基因功能富集分析图,简单了解过后发现这是gene ontology(基因本体论)有关的知识,之前从未了解过。于是想写篇博客记录一下新知识以及第一次教粉丝绘图。
看完本篇博客,你将学习到:
1. 什么是基因本体论
2. 如何绘制基因功能富集分析图
一、基因本体论介绍
基因本体论 (Gene Ontology, GO)是生物信息学主要的项目,旨在是统一所有物种的基因和基因产物属性的代表。具体而言,这个项目的目的是(1)维持和发展在控制下基因及基因产物属性的词汇;(2)注释基因及基因产物属性,同化或传播注释数据;(3)提供工具,方便访问项目提供的数据的各个方面,并使用GO实现实验数据的功能解释[1]。
GO包含生物学的三个方面[1]:
细胞组分(cellular component):细胞的组成部分或细胞外环境
分子功能(molecular function):基因产物在分子水平的活性,如结合或催化活性
生物过程(biological process):有明确开始和结束的操作或一组分子事件,与整合的生命单位的功能有关。
哔哩哔哩有个讲解视频,个人觉得很不错,感兴趣的读者可以去看看哔哩哔哩讲解视频
二、数据获得
数据是粉丝提供的,感兴趣的读者可以下载:
百度网盘下载链接
提取码:svwx

Category:种类
CategoryID:种类id
GO:GO编码
Description:描述
PARENT_GO:上一级的GO(这个可能没理解对)
LogP:P值的负对数
Enrichment:富集
Z-score:
GeneInHitList:基因在自己列表中的数量
GeneInGOAndHitList:基因在自己列表中且在GO中的数量
Z-score 的意思没有查到,如果有读者知道,可以告知一下,谢谢!
三、气泡图绘制
因为它的数据只需要简单的处理就可以使用了,所以话不多说直接上代码:
library(openxlsx)
library(ggplot2)setwd('YOUR_WORKWAY')# import data
data <- read.xlsx('1021_csdn.xlsx', sheet = 2)# data processing## calculate gene ratio
data$GeneRatio <- data$`#GeneInGOAndHitList` / data$`#GeneInHitList`## transfer negative PV to positive
data$positive_Pvalue <- -(data$LogP)draw_data <- data.frame(Description = data$Description, LogP = data$positive_Pvalue,GeneRatio = data$GeneRatio,Enrichment = data$Enrichment)# visualize data
ggplot(draw_data, aes(x=GeneRatio, y=Description))+geom_point(aes(size=Enrichment, color=LogP))+scale_colour_continuous(name="LogP", low='orange', high='red')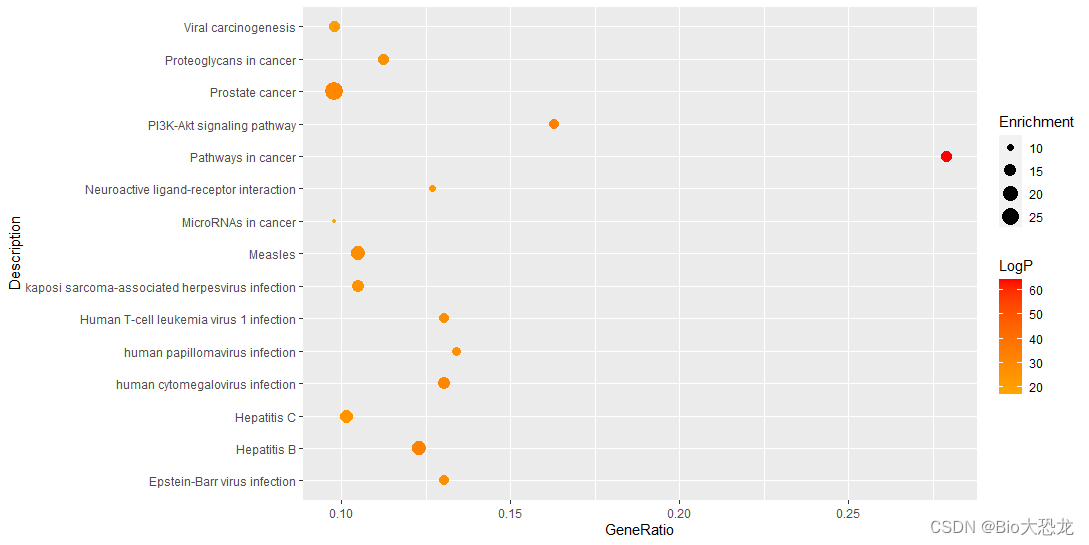
绘制出的散点图有部分点偏移了,可以对数据进行缩放(不等缩放,类似于y=ax+b)
library(openxlsx)
library(ggplot2)setwd('YOUR_WORKWAY')# import data
data <- read.xlsx('1021_csdn.xlsx', sheet = 2)# data processing## calculate gene ratio
data$GeneRatio <- data$`#GeneInGOAndHitList` / data$`#GeneInHitList`## transfer negative PV to positive
data$positive_Pvalue <- -(data$LogP)## scale the gene ratio
data$percentage <- ceiling(rep(1,15) / data$GeneRatio)
data$GeneRatio <- data$percentage * data$GeneRatiodraw_data <- data.frame(Description = data$Description, LogP = data$positive_Pvalue,GeneRatio = data$GeneRatio,percentage = data$percentage * 10)# visualize data
ggplot(draw_data, aes(x=GeneRatio, y=Description))+geom_point(aes(size=percentage, color=LogP))+scale_colour_continuous(name="LogP", low='pink', high='red')+labs(size="10*percentage")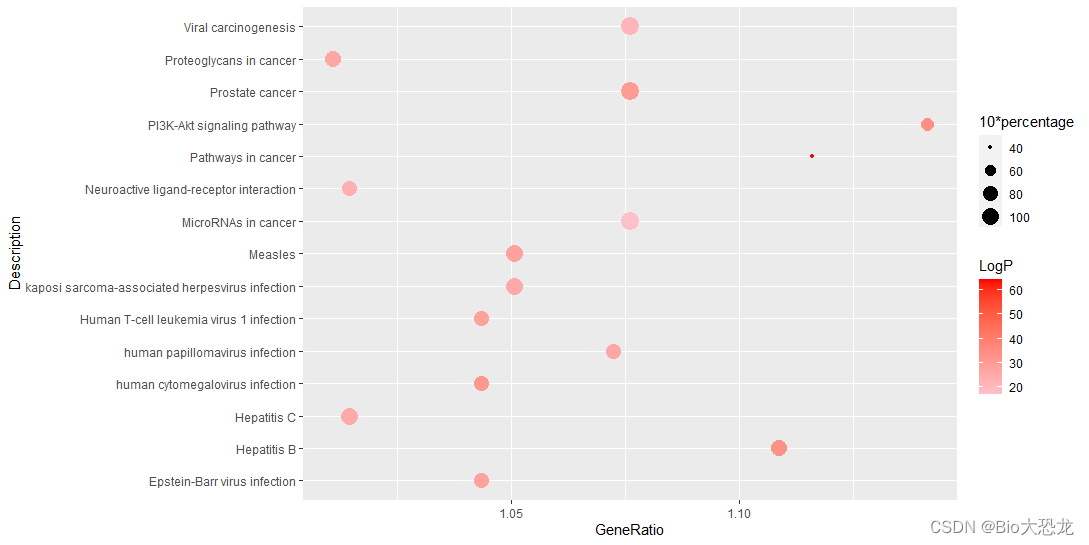
四、参考
[1]. wikipedia Gene Ontology
[2]. 哔哩哔哩视频讲解
这篇关于R语言基因功能富集分析气泡图的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







