本文主要是介绍超实用!用ChimeraX对AlphaFold 结果可视化(下): 蛋白互作界面分析,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
❝之前给大家介绍了AlphaFold预测结果可视化软件Chimera( 点击此处阅读)以及如何利用ChimeraX软件美化蛋白结构( 点击此处阅读);利用AlphaFold预测蛋白互作后我们最想知道的是互作位点和界面,本期我们将介绍如何使用ChimeraX分析蛋白互作界面。
❞
蛋白质的三级结构和四级结构的作用力主要有「疏水作用、离子键、氢键和范德华力」等,利用ChimeraX可以分析这些分子间的作用力。
01:分析互作位点
在ChimeraX中分析蛋白结构间作用力的方法有三种,包括「Clashes」(分子间因为距离太近导致的不利相互作用)、「Contacts」(分子间的各种极性和非极性互作)和「H-Bonds」(氢键分析)。可以在Tools>>Structure Analysis中选择。

三种分析流程基本相似,在此以分析氢键为例进行介绍。点击「H-Boods」后弹出如下界面。红框①内的距离和角度等参数软件已设置好,一般选择默认参数,不需要更改。默认的氢键颜色为黑色,点击Color可以更改显示氢键的颜色。红框②内可以选择显示蛋白结构间、蛋白内部等各种想要的氢键。

红框③选择后可以在Log界面看到具体的互作位点,为我们后续标注位点提供便利。
分析完成后就可以看到氢键和含有氢键的位点。
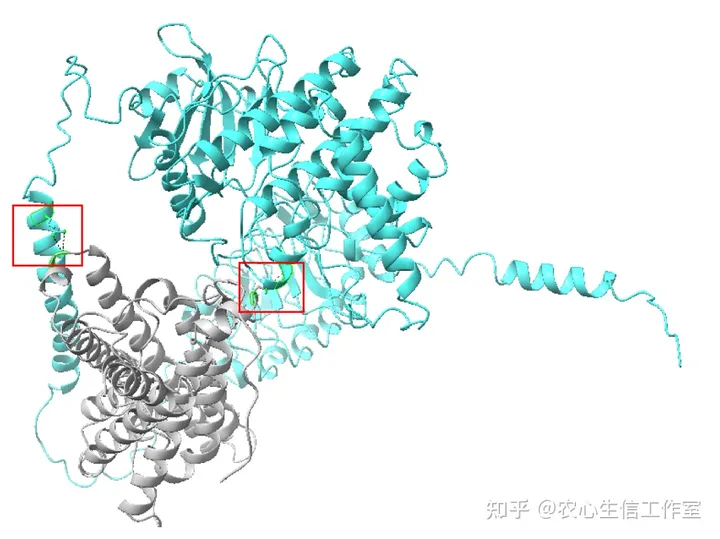

02:查看并标注互作位点
当分析出互作位点后需要对其进行观察和标注,根据Log界面的信息我们选中互作的位点,然后依次选择Graphics>>View selected就可以查看氢键位点的细节图。

知道氢键位点后可以对这个位点进行标注,依次选择Actions>>Label>>Residues就可以选择想要的标签,这里的标签颜色也是可以更改的,如果对标签位置不满意也可移动标签。

03:结果导出与保存
当我们分析完结果后便可以导出我们想要的图片,点击保存可保存图片为「tif」、「jpg」等常规格式,这里建议大家也将分析后的结果存储为「cxs」格式,用于后续分析或修改。

以上是关于chimera的常规用法介绍,chimera还有很多可以开发的用法满足蛋白可视化中很多需求,大家可以在使用中点击Help了解具体用法。
//
本期的介绍就到这里,至此「用ChimeraX对AlphaFold 结果可视化」系列教程已完结。喜欢的朋友请多多点赞、收藏加转发!!如果大家在阅读的过程中有任何问题,欢迎在农心生信工作室后台留言,或加入 农心生信工作室学习交流群 提问,我们会努力为大家答疑解惑。
这篇关于超实用!用ChimeraX对AlphaFold 结果可视化(下): 蛋白互作界面分析的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







