本文主要是介绍代码:DESeq2包做转录组RNAseq差异分析,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
教程来自:R语言DESeq2包做转录组RNAseq差异表达分析的一个简单小例子_哔哩哔哩_bilibili
代码:
getwd()
counts<-read.csv("lsm/counts.csv",row.names = 1)head(counts)
dim(mycounts)
counts_1<-counts[rowSums(counts) != 0,]
dim(counts_1)
group<-read.csv("lsm/group.csv",stringsAsFactors = T)
group
colnames(counts_1) == group$id'''
如果没有安装DESeq2,使用如下命令进行安装,
把两个井号去掉
'''
#install.packages("BiocManager")
#BiocManager::install("DESeq2")
library(DESeq2)dds <- DESeqDataSetFromMatrix(countData=counts_1, colData=group, design=~dex)dds <- DESeq(dds)
res <- results(dds)head(res)
class(res)
res_1<-data.frame(res)
class(res_1)
head(res_1)
dim(res_1)
library(dplyr)
res_1 %>% mutate(group = case_when(log2FoldChange >= 1 & pvalue <= 0.05 ~ "UP",log2FoldChange <= -1 & pvalue <= 0.05 ~ "DOWN",TRUE ~ "NOT_CHANGE")) -> res_2table(res_2$group)write.csv(res_2,file="rnaseq/diff_expr_result.csv",quote = F)制作count.csv时应注意:
1.第一列选取gene_id,不然会有重复项。
2.数据选择read counts,注意不是FPKM。
3.counts列表里的read counts需要为整数。因此在excel种用ROUND函数四舍五入取整。
4.第一列不能有表头
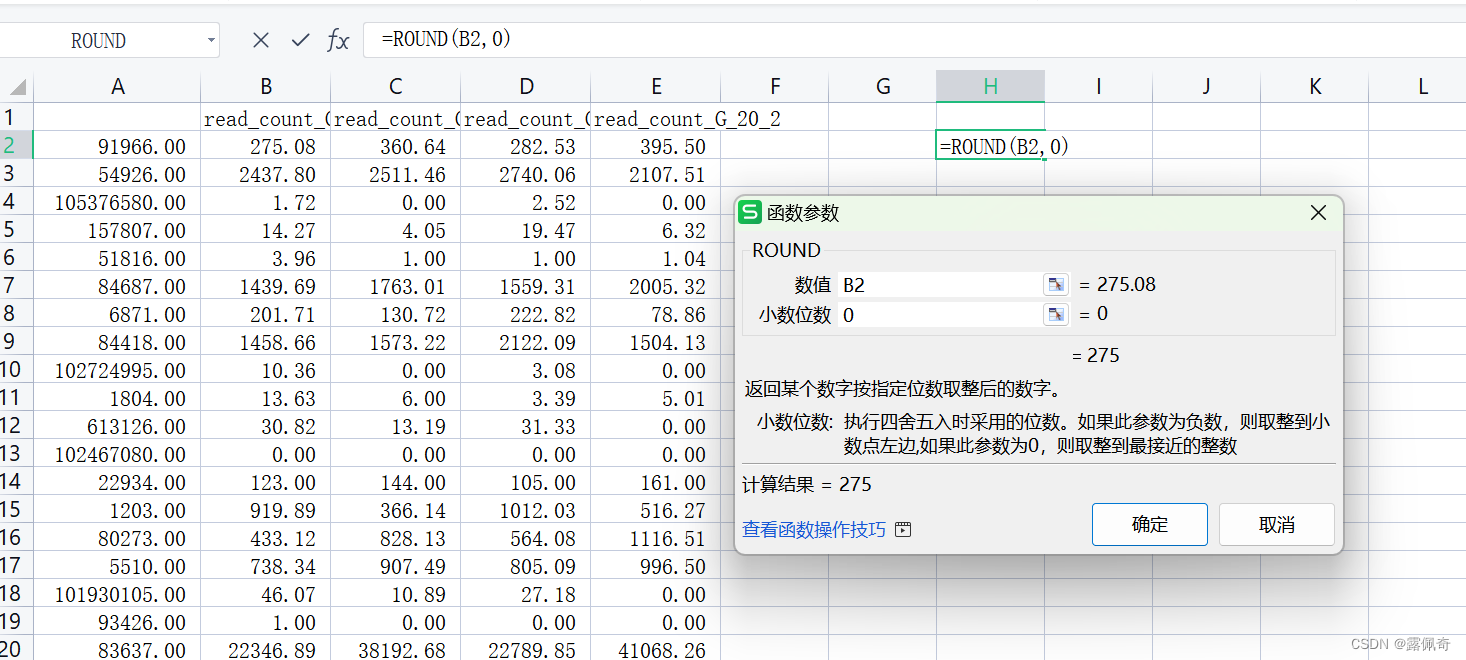
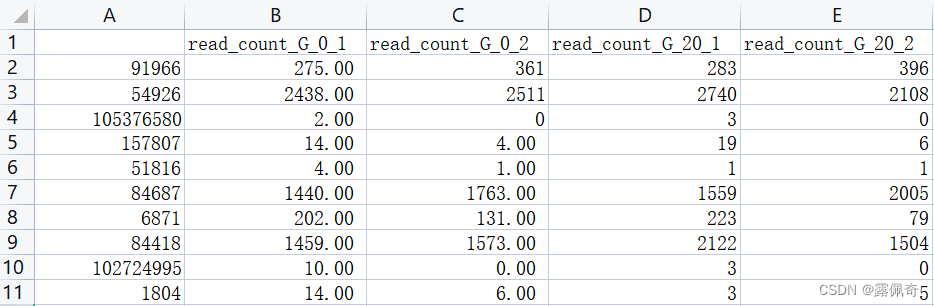

制作group.vsc
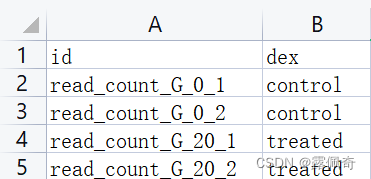
踩坑:
1.报错:Error: unexpected ')' in " )"
跟括号没关系,其实是代码忘加逗号。
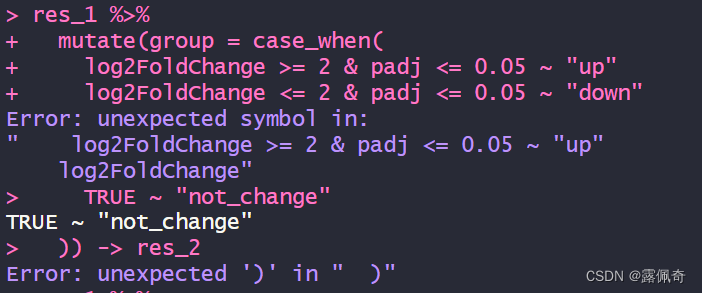
修改后:
res_1 %>%mutate(group = case_when(log2FoldChange >= 2 & padj <= 0.05 ~ "up",log2FoldChange <= 2 & padj <= 0.05 ~ "down",TRUE ~ "not_change")) -> res_2遇到跑不通的多跑几次

这篇关于代码:DESeq2包做转录组RNAseq差异分析的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!





