本文主要是介绍Seurat -- Normalize Data,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
文章目录
- brief
- 为什么要做normalization
- 实例
- Normalization之前的预处理
- NormalizeData --> LogNormalize
- 这种方法有是什么缺点呢
- SCTransform
- 仔细去看看这个函数
- 所以这个函数到底干了什么
- 这种normalization方法优秀在哪里
- 正确使用scTransform
brief
- seurat提供的教学里面包含了
Standard pre-processing workflow,workflow包括QC,normalization,scale data ,detection of highly variable features。 - 其中 normalization就有蛮多方法的,seurat自己就提供了两种,一种是"LogNormalization",另一种是目前正在大力推荐的"SCTransform",而且每一种方法参数众多,我觉得有必要对其进行仔细的探究,所以需要分开学习和记录。
- 还有 detection of highly variable features以及scale data,也是方法和参数众多,我觉得有必要对其进行仔细的探究,所以需要分开学习和记录
- 概要图以及系列博文可以参见链接。
这里主要记录了Normalize Data的学习过程,包括LogNormalization 和 SCTransform两种normalization方法的参数以及对数据的影响,以及对下游数据处理流程的影响等。
为什么要做normalization
scRNA-seq中细胞与细胞之间的异质性可能是生物学上的,也可能是技术上造成的。其中每个细胞检测到的分子数,测序深度对细胞之间的变异度贡献很大,需要进行矫正。



实例
Normalization之前的预处理
library(dplyr)
library(Seurat)
library(patchwork)rm(list=ls())# 使用read10X读取output of the cellranger pipeline from 10X,包括barcodes,genes,matrix.mtx三个文件
pbmc.data <- Read10X(data.dir = "D:/djs/pbmc3k_filtered_gene_bc_matrices/filtered_gene_bc_matrices/hg19")
# 使用 CreateSeuratObject函数构造seurat对象
pbmc <- CreateSeuratObject(counts = pbmc.data, project = "pbmc3k",min.cells = 3, min.features = 200,names.delim = "-",names.field = 1)pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^MT-")pbmc <- subset(pbmc, subset = nFeature_RNA > 200 & nFeature_RNA < 2500 & percent.mt < 5)
NormalizeData --> LogNormalize

-
object
上述构建的seurat object -
normalization.method
-
LogNormalize: Feature counts for each cell are divided by the total counts for that cell and multiplied by the scale.factor. This is then natural-log transformed using log1p.For counts per million (CPM) set scale.factor = 1e6
-
CLR: Applies a centered log ratio transformation
-
RC: Relative counts. Feature counts for each cell are divided by the total counts for that cell and multiplied by the scale.factor. No log-transformation is applied.
-
-
scale.factor
Sets the scale factor for cell-level normalization。For counts per million (CPM) set scale.factor = 1e6 -
margin
If performing CLR normalization, normalize across features (1) or cells (2) -
verbose
display progress bar for normalization procedure
# 下面就可以进行LogNormalize
pbmc <- NormalizeData(pbmc, normalization.method = "LogNormalize", scale.factor = 10000)# 其中NormalizeData函数提供了三个normalization.method:
# LogNormalize,CLR,RC,从描述可以看出来差异,那具体差在哪儿呢?
首先RC这种normalization.method我们就不考虑了,和LogNormalize比较你可以发现LogNormalize 之前做的就是RC然后做了log转化。log转化让方差稳定而且非正态的数据近似于正态分布了。
最主要要比较的是CLR和LogNormalize,CLR称为中心对数转化,具体原理和算法需要技术文档帮助,这里不写了。
# install.packages("compositions")
# library(compositions)
# library(reshape2)pbmc_logN <- NormalizeData(pbmc, normalization.method = "LogNormalize", scale.factor = 10000)
pbmc_CLR <- NormalizeData(pbmc, normalization.method = "CLR",margin = 1)opar <- par(no.readonly=TRUE)
par(mfrow=c(1,2))
hist(pbmc_logN[["RNA"]]@data["MT-ND4",],main = "LogNormalize",xlab = "")
hist(pbmc_CLR[["RNA"]]@data["MT-ND4",],main = "CLR",xlab = "")
par(opar)# log转化让方差稳定而且非正态的数据近似于正态分布了,其中LogNormalize方法貌似更胜一筹
# 其中library(compositions)中有clr函数,利用clr手动处理和
# NormalizeData(pbmc, normalization.method = "CLR",margin = 1) 结果不一样

这种方法有是什么缺点呢

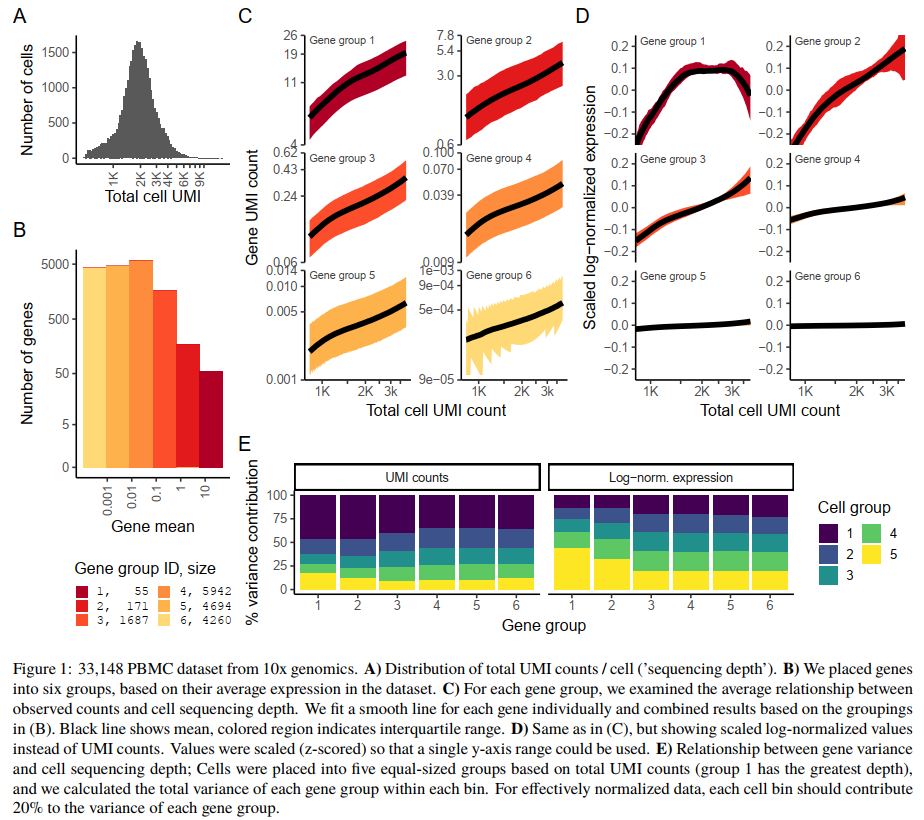
SCTransform
我目前在seurat看到的技术文档版本是V2,链接如下:
https://satijalab.org/seurat/articles/sctransform_v2_vignette.html
# install glmGamPoi,可以提高scTransform的运算速度
if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager")
BiocManager::install("glmGamPoi")# install sctransform >= 0.3.3
install.packages("sctransform")library(sctransform)
# invoke sctransform - requires Seurat>=4.1
pbmc_scT <- SCTransform(pbmc, vst.flavor = "v2")
# 上面那一步就把normalization做完了str(pbmc_scT)
# 现在的seurate object数据组织结构在asssys slot下多了一个SCT slot,这里面的东西和 RNA slot平级
# 如果你用LogNormalization 数据会放在 assays$RNA@data@x 下面
# 现在放在了 assays$SCT@data@x 下面

pbmc_logN <- NormalizeData(pbmc, normalization.method = "LogNormalize", scale.factor = 10000)
pbmc_scT <- SCTransform(pbmc, vst.flavor = "v2")opar <- par(no.readonly=TRUE)
par(mfrow=c(1,2))
hist(pbmc_logN[["RNA"]]@data["MT-ND4",],main = "LogNormalize",xlab = "")
hist(pbmc_scT[["SCT"]]@data["MT-ND4",],main = "SCTransform",xlab = "")
par(opar)

仔细去看看这个函数
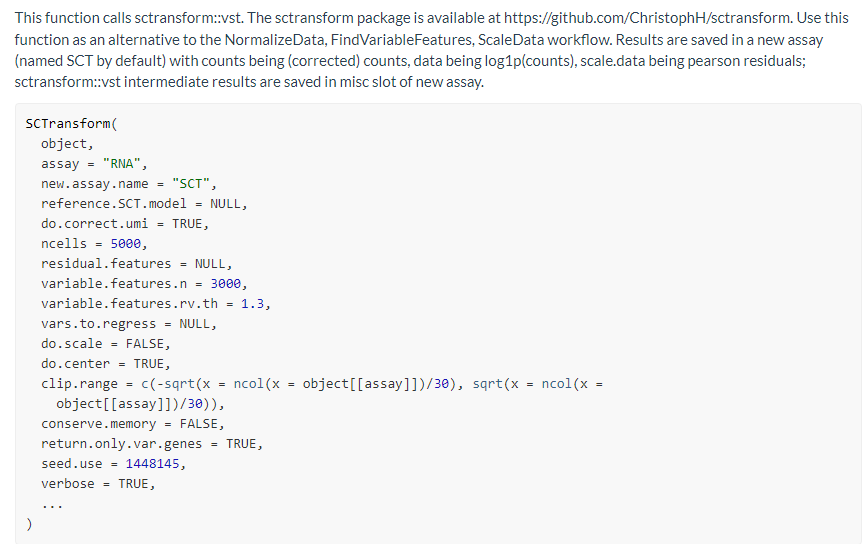
-
assay
Name of assay to pull the count data from; default is ‘RNA’ -
do.correct.umi
Place corrected UMI matrix in assay counts slot; default is TRUE
我读到这里才发现该函数还会对原始的counts数据进行矫正,然后放到assays@SCT$counts@x下面 -
ncells
Number of subsampling cells used to build NB regression; default is 5000 -
residual.features
Genes to calculate residual features for; default is NULL (all genes). If specified, will be set to VariableFeatures of the returned object. -
variable.features.n
Use this many features as variable features after ranking by residual variance; default is 3000. Only applied if residual.features is not set. -
variable.features.rv.th
Instead of setting a fixed number of variable features, use this residual variance cutoff; this is only used when variable.features.n is set to NULL; default is 1.3. Only applied if residual.features is not set. -
vars.to.regress
Variables to regress out in a second non-regularized linear regression. For example, percent.mito. Default is NULL。这里需要注意的是指定哪些基因或者变量让其不参与回归模的估计。比如出现线粒体基因细胞周期基因等明显影响细胞聚类结果的时候,应该剔除回归模型的估计。 -
do.scale
Whether to scale residuals to have unit variance; default is FALSE -
do.center
Whether to center residuals to have mean zero; default is TRUE -
clip.range
Range to clip the residuals to; default is c(-sqrt(n/30), sqrt(n/30)), where n is the number of cells -
return.only.var.genes
If set to TRUE the scale.data matrices in output assay are subset to contain only the variable genes; default is TRUE -
seed.use
Set a random seed. By default, sets the seed to 1448145. Setting NULL will not set a seed. -
verbose
Whether to print messages and progress bars
所以这个函数到底干了什么
这个函数发表的文章链接。



- 个人理解哈,第一步用基因的表达量作为因变量,整个细胞的total UMI counts作为解释变量做回归分析
- 第二部估计回归模型的参数以及矫正参数
- 根据回归模型的参数进行数据转化,UMI数 ==> Pearson residual
个人理解哈,他在做用基因的表达量作为因变量,整个细胞的total UMI counts作为解释变量做回归分析的时候得到了测序深度对基因变异度的解释程度,基因的总变异 - 测序深度能解释的变异 = Pearson residual(基因的生物学变异 + 误差 )。所以用Pearson residual做下游分析相对来说更准确些。
============================================================================
这种normalization方法优秀在哪里
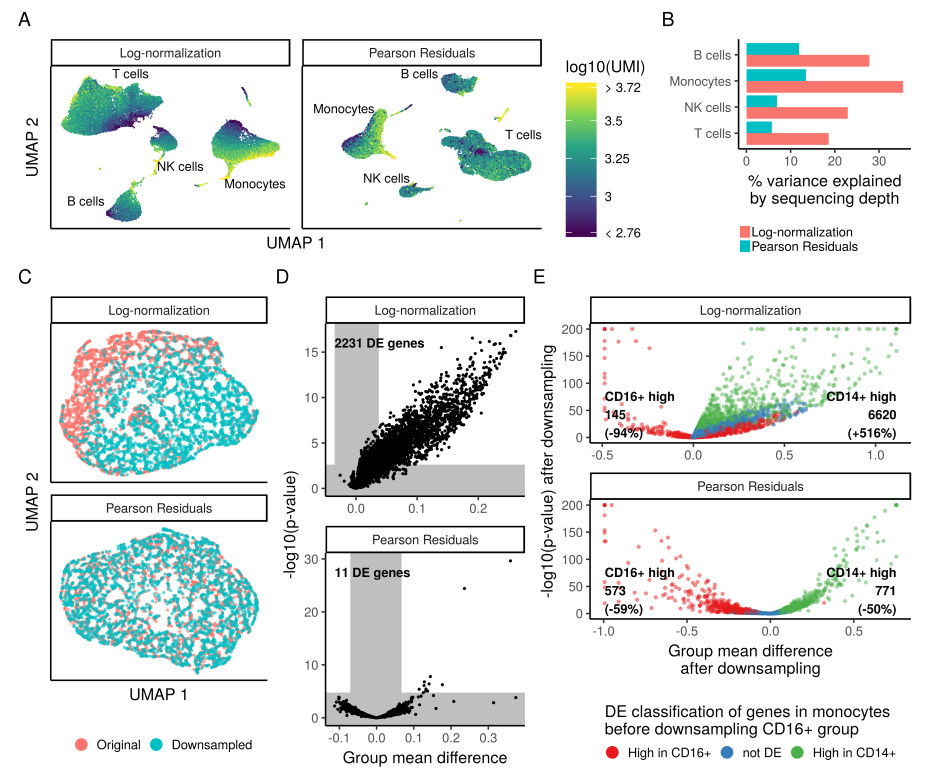

正确使用scTransform
比如细胞周期以及线粒体基因可能会对细胞间的变异贡献度很大,可能会导致scTransform函数构建回归模型的时候错误估计模型参数,我们应该让这些基因不参与scTransform函数构建回归模型。
- 估计细胞周期对细胞变异的影响
# BiocManager::install("ensembldb")
# BiocManager::install("AnnotationHub")
library(ensembldb)
library(AnnotationHub)
library(RCurl)cell_cycle_genes <- read.csv("I:/bioinformatics/cell_cycle_Home_sapies.csv.txt",sep = ",",header = T)# We will use the AnnotationHub R package to query Ensembl using the ensembldb R package
# Connect to AnnotationHub
ah <- AnnotationHub()
# Access the Ensembl database for organism
ahDb <- query(ah, pattern = c("Homo sapiens", "EnsDb"), ignore.case = TRUE)# Acquire the latest annotation files
id <- ahDb %>%mcols() %>%rownames() %>%tail(n = 1)# Download the appropriate Ensembldb database
edb <- ah[[id]]# Extract gene-level information from database
annotations <- genes(edb, return.type = "data.frame")# Select annotations of interest
annotations <- annotations %>%dplyr::select(gene_id, gene_name, seq_name, gene_biotype, description)# Get gene names for Ensembl IDs for each gene
cell_cycle_markers <- dplyr::left_join(cell_cycle_genes, annotations, by = c("geneID" = "gene_id"))
write.csv(cell_cycle_markers,file = "I:/bioinformatics/cell_cycle_Home_sapies_annotation.csv")# Acquire the S phase genes
s_genes <- cell_cycle_markers %>%dplyr::filter(phase == "S") %>%pull("gene_name")# Acquire the G2M phase genes
g2m_genes <- cell_cycle_markers %>%dplyr::filter(phase == "G2/M") %>%pull("gene_name")# Perform cell cycle scoring
pbmc <- CellCycleScoring(pbmc,g2m.features = g2m_genes,s.features = s_genes)# Perform PCA and color by cell cycle phase
pbmc <- NormalizeData(pbmc, normalization.method = "LogNormalize", scale.factor = 10000)
pbmc <- FindVariableFeatures(pbmc,assay = "RNA" ,selection.method = "vst", nfeatures = 2000)all.genes <- rownames(pbmc)
pbmc <- ScaleData(pbmc, features = all.genes,assay = "RNA")
pbmc <- RunPCA(pbmc)# Visualize the PCA, grouping by cell cycle phase
DimPlot(pbmc,reduction = "pca",group.by= "Phase")

这里可以看到细胞周期对细胞聚类的影响很小。如果影响很大,你如何在scTransform中剔除呢?
pbmc <- SCTransform(pbmc, vars.to.regress = c("Phase"))
- 估计线粒体基因对细胞变异的影响
rm(list=ls())
# 使用read10X读取output of the cellranger pipeline from 10X,包括barcodes,genes,matrix.mtx三个文件
pbmc.data <- Read10X(data.dir = "D:/djs/pbmc3k_filtered_gene_bc_matrices/filtered_gene_bc_matrices/hg19")
# 使用 CreateSeuratObject函数构造seurat对象
pbmc <- CreateSeuratObject(counts = pbmc.data, project = "pbmc3k",min.cells = 3, min.features = 200,names.delim = "-",names.field = 1)# 计算 a percentage of cell reads originating from the mitochondrial genes
pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^MT-")
# 计算 complexity of the RNA species
pbmc@meta.data$log10GenesPerUMI <- log10(pbmc$nFeature_RNA) / log10(pbmc$nCount_RNA)pbmc <- subset(pbmc, subset = nFeature_RNA > 200 & nFeature_RNA < 2500 & percent.mt < 5)
pbmc <- NormalizeData(pbmc, normalization.method = "LogNormalize", scale.factor = 10000)
pbmc <- FindVariableFeatures(pbmc,assay = "RNA" ,selection.method = "vst", nfeatures = 2000)all.genes <- rownames(pbmc)
pbmc <- ScaleData(pbmc, features = all.genes,assay = "RNA")
pbmc <- RunPCA(pbmc)
summary(pbmc@meta.data$percent.mt)
pbmc@meta.data$mitoFr <- cut(pbmc@meta.data$percent.mt, breaks=c(-Inf, 1.520, 2.011, 2.591, Inf), labels=c("Low","Medium","Medium high", "High"))
DimPlot(pbmc,reduction = "pca",group.by= "mitoFr")

这里可以看到线粒体基因对细胞聚类的影响很小。如果影响很大,你如何在scTransform中剔除呢?
pbmc <- SCTransform(pbmc, vars.to.regress = c("percent.mt"))
这篇关于Seurat -- Normalize Data的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!




