本文主要是介绍MIT-6874-Deep Learning in the Life Sciences Week5,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
Lecture 6: Regulatory genomicsGene regulation, chromatin accessibility, DNA regulatory code
- AI在调控组学中应用
- 1 生物学基础:调控构建块
- 1.1 基因调控:Cell diversity, Epigenomics, Regulators(TFs), Motifs, Disease role
- 1.2 Probing gene regulation: TFs/histones: ChIP-seq, Accessibility: DNase/ATAC-seq
- 2 调控组学和基序发现的传统方法
- 2.1 基于Enrichment-based基序发现:EM算法、Gibbs Sampling
- 2.2 基于实验的方法:PBMs,SELEX. Comparative genomics:Evolutionary conservation
- 在这里插入图片描述
- 3 卷积神经网络在转录调控中的基础应用
- 3.1 低维特征:主要想法:pixels<->DNA letters. Patches/filters<->Motifs. Higher<->combinations
- 3.2 高维特征:学习卷积核<-> Motif discovery. Applying them<-> Motif matches
- 4 CNNS/RNNs在调控组学中的实际应用:各种架构
- 4.1 DeepBind: learn motifs, use in(shallow) fully-connected layer, mutations impact
- 4.2 DeepSea: Train model directly on mutational impact prediction
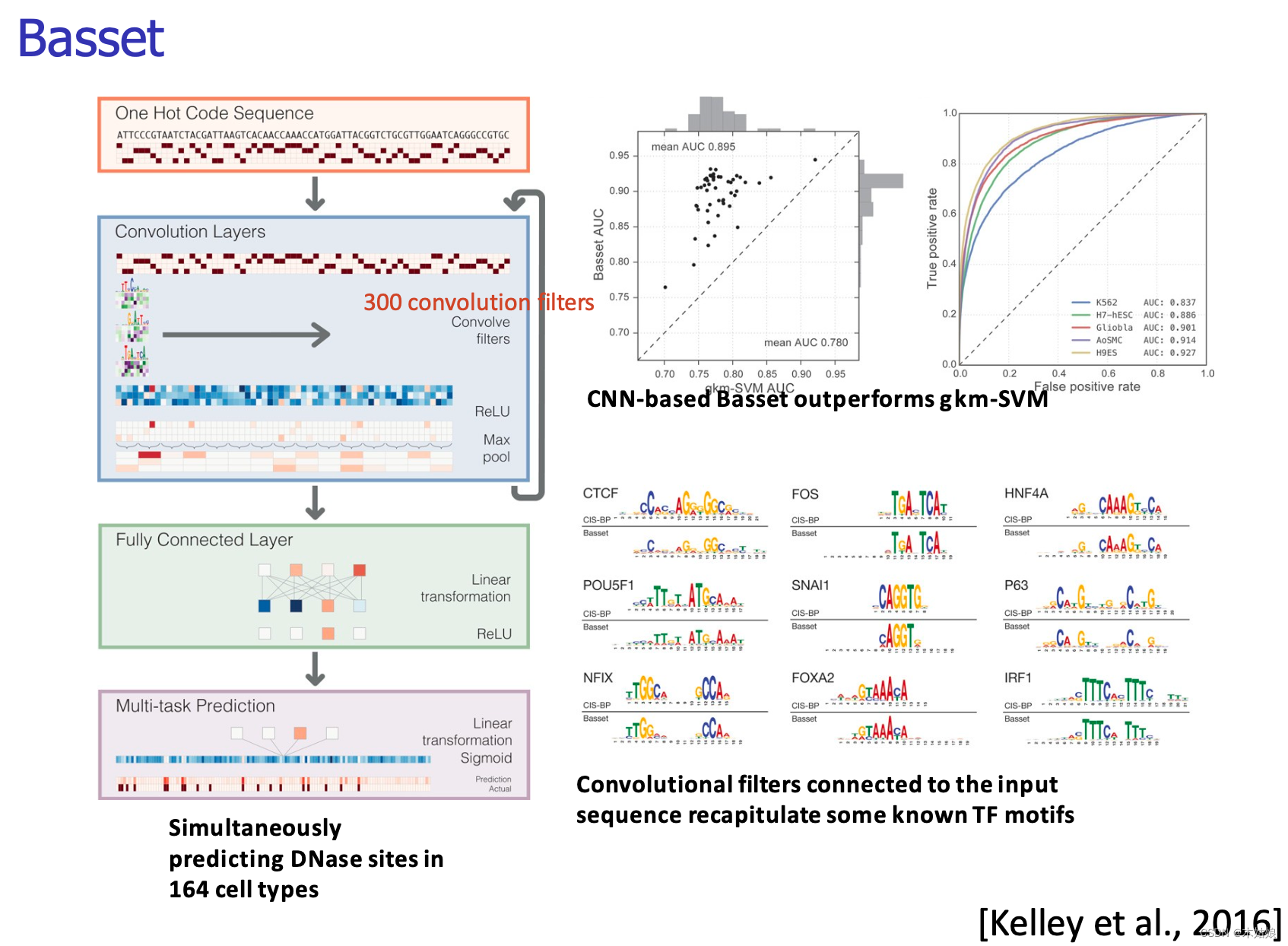
- 4.3 Baseet: Multi-task DNase prediction in 164 cell types, reuse/learn motifs
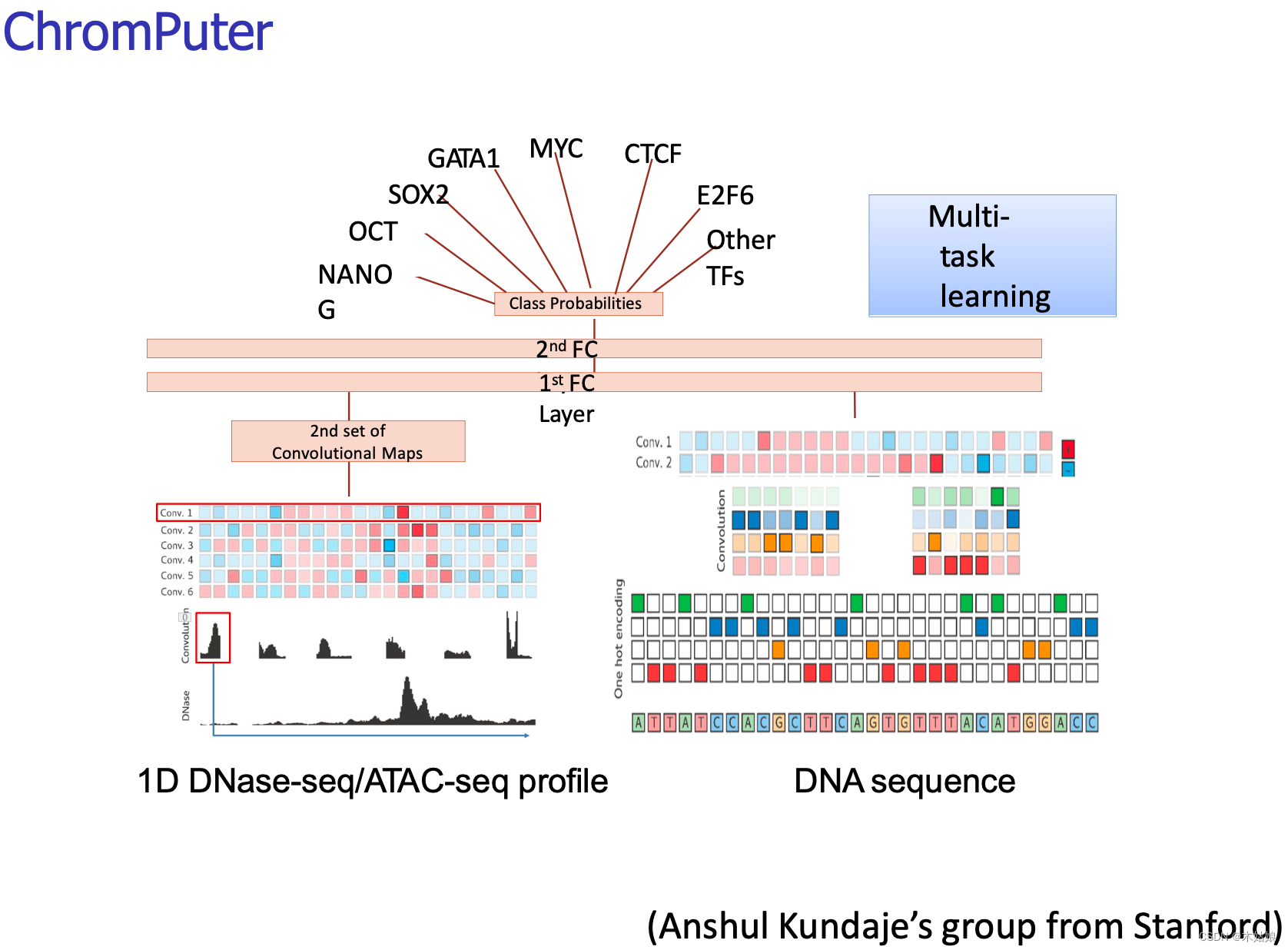
- 4.4 ChromPuter: Multi-task prediction of different TFs, reuse partner motifs
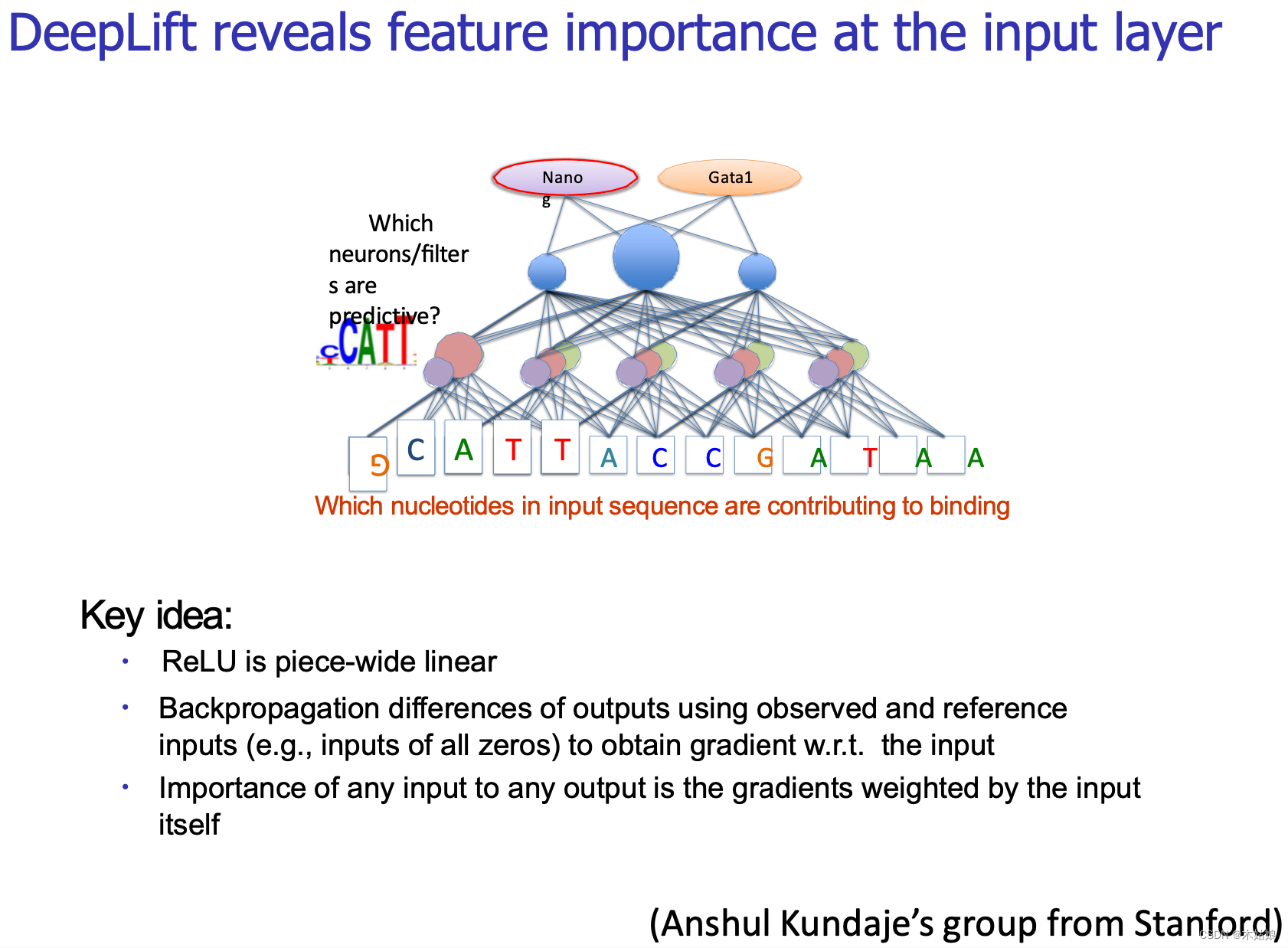
- 4.5 DeepLIFT: Model interpretation based on neuron activation properties
- 5 讲座:斯坦福Anshul Kundaje,Deep Learning for Reg. Genomics
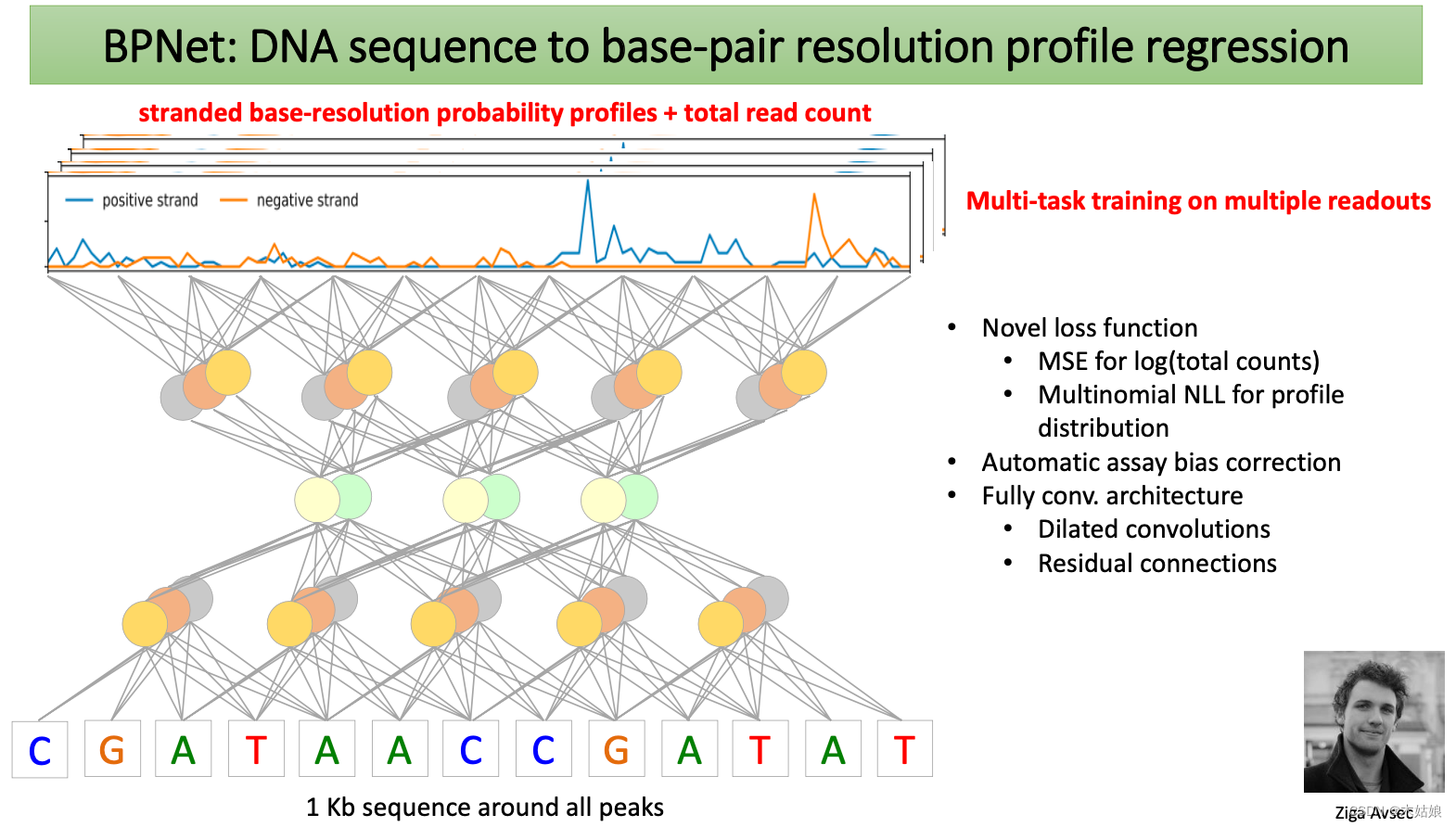
- BPNet: DNA sequence to base-pair resolution profile regression
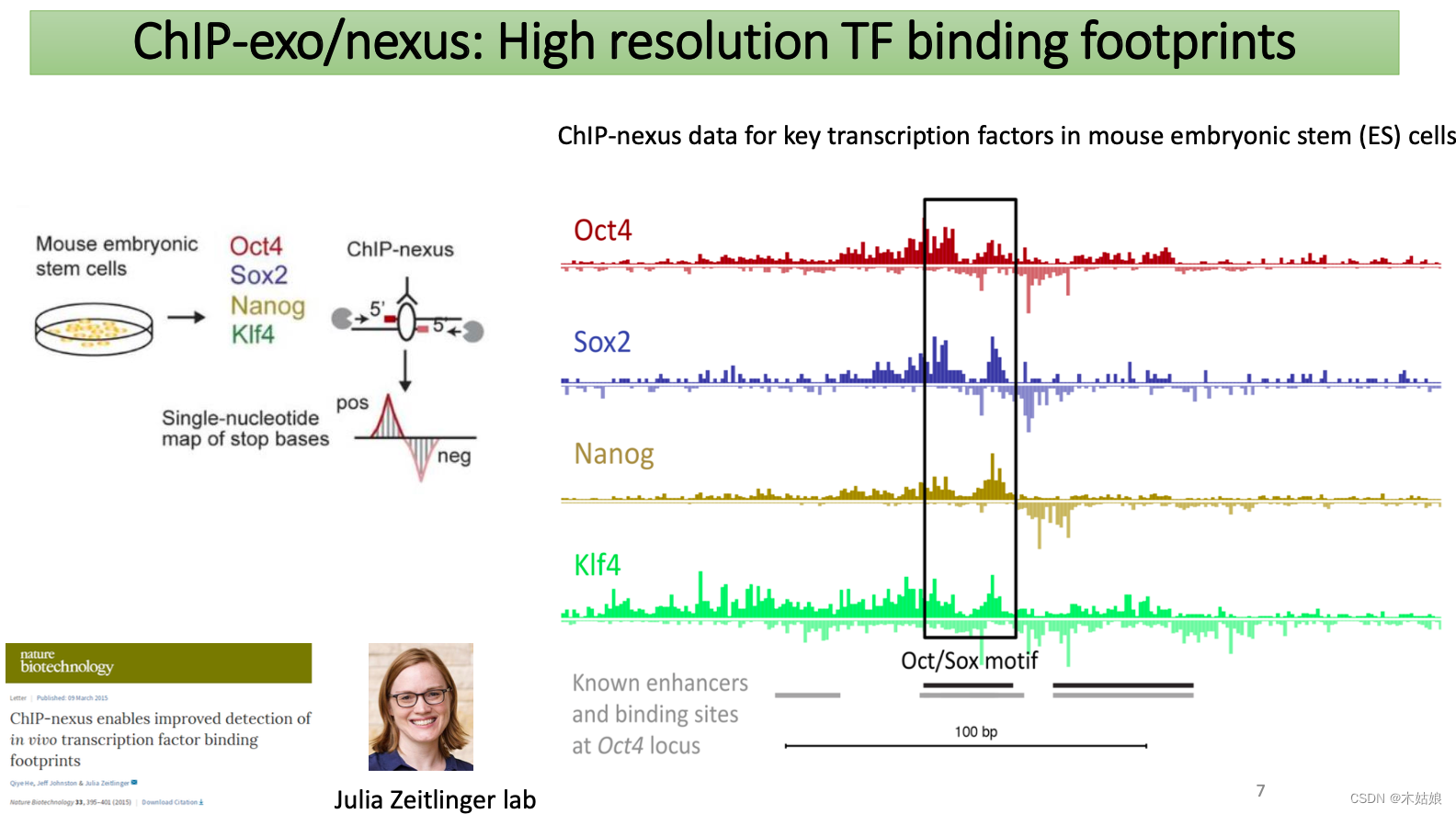
- ChIP-exo/nexus: High resolution TF binding footprints
AI在调控组学中应用
1 生物学基础:调控构建块
1.1 基因调控:Cell diversity, Epigenomics, Regulators(TFs), Motifs, Disease role
基因通过调控,表达不同的细胞类型,从而在人体内产生不同的器官。
DNA packaging的意义:DNA 的高度螺旋化,实现染色质装配的目的
dna通过双螺旋成双链后,与组蛋白结合构成核小体,在通过不断的螺旋和卷曲形成超螺旋体,最后再不短聚集形成染色体/染色质。如果不行成这种高度密集的结构的话所需要的空间就很大,也阻碍了细胞内的各种代谢反应

(DNA压缩的主要步骤是什么啊? - Ryan的回答 - 知乎
https://www.zhihu.com/question/381570484/answer/1103039550)
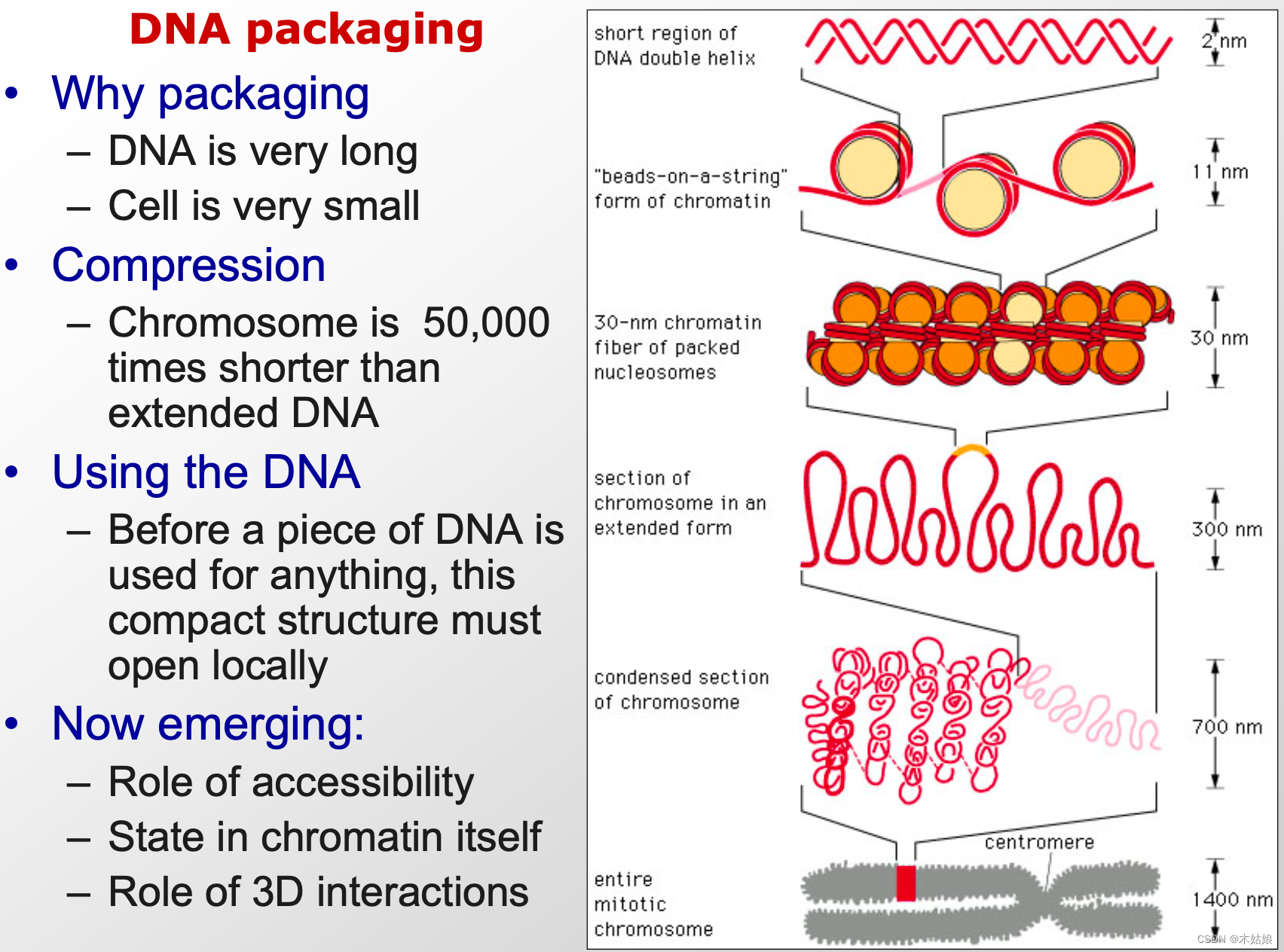
核小体的装配是染色体装配的第一步,DNA包装成核小体,大约压缩了7倍.
染色质以核小体作为基本结构逐步进行包装压缩,经30nm染色质纤维、超螺旋环、最后压缩包装成染色体,总共经过四级包装.
● DNA到核小体
● 从核小体到螺线管(solenoid)
● 从螺线管到超螺线管(supersolenoid)
● 从超螺线管到染色体
DNA在细胞内压缩是非常重要的。DNA内的约147个碱基对loop形成核小体(nucleosome),每个核小体由四个 组蛋白 组成,大概50个核小体组成一个染色质丝(chromatin fiber)
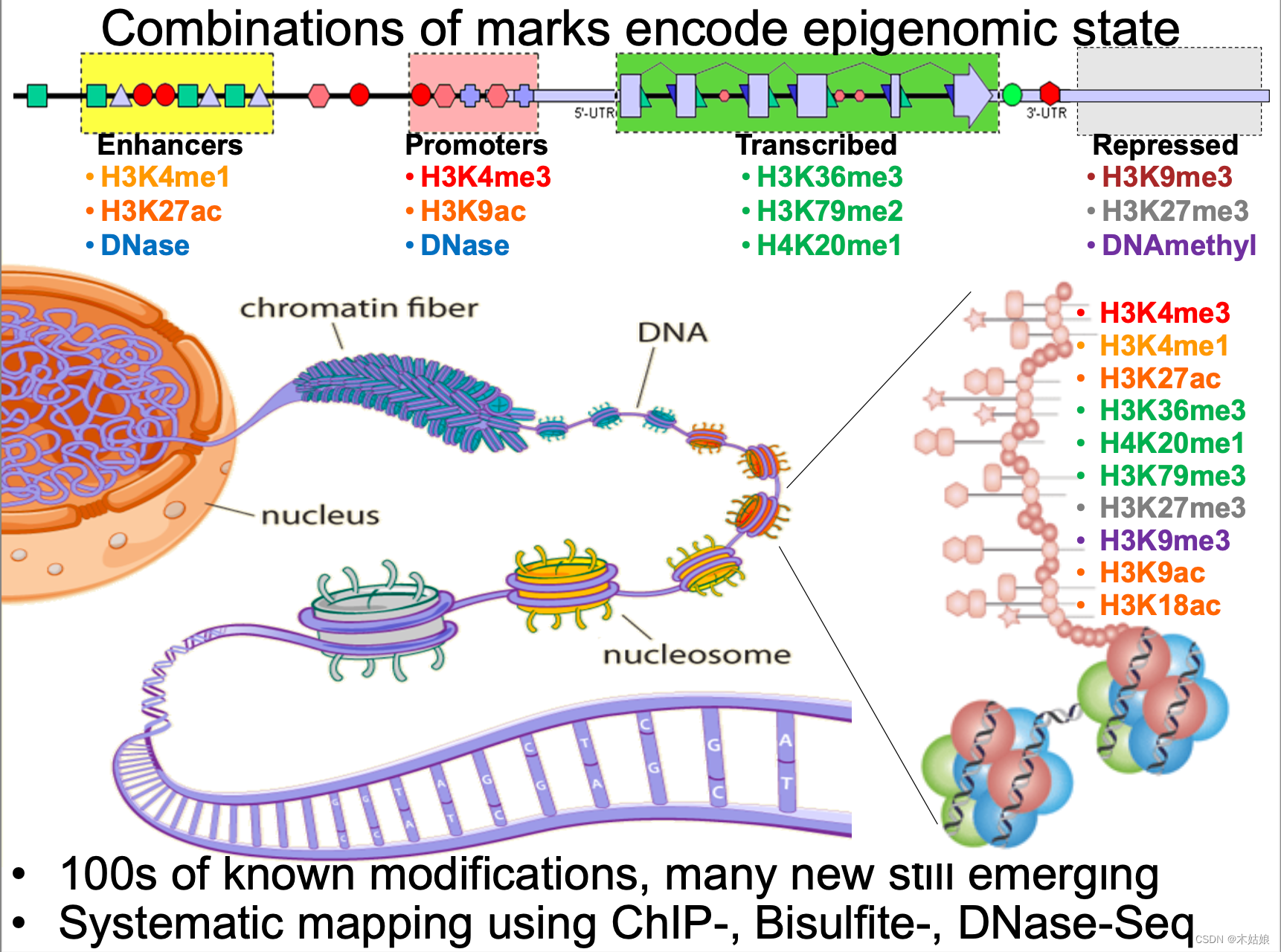
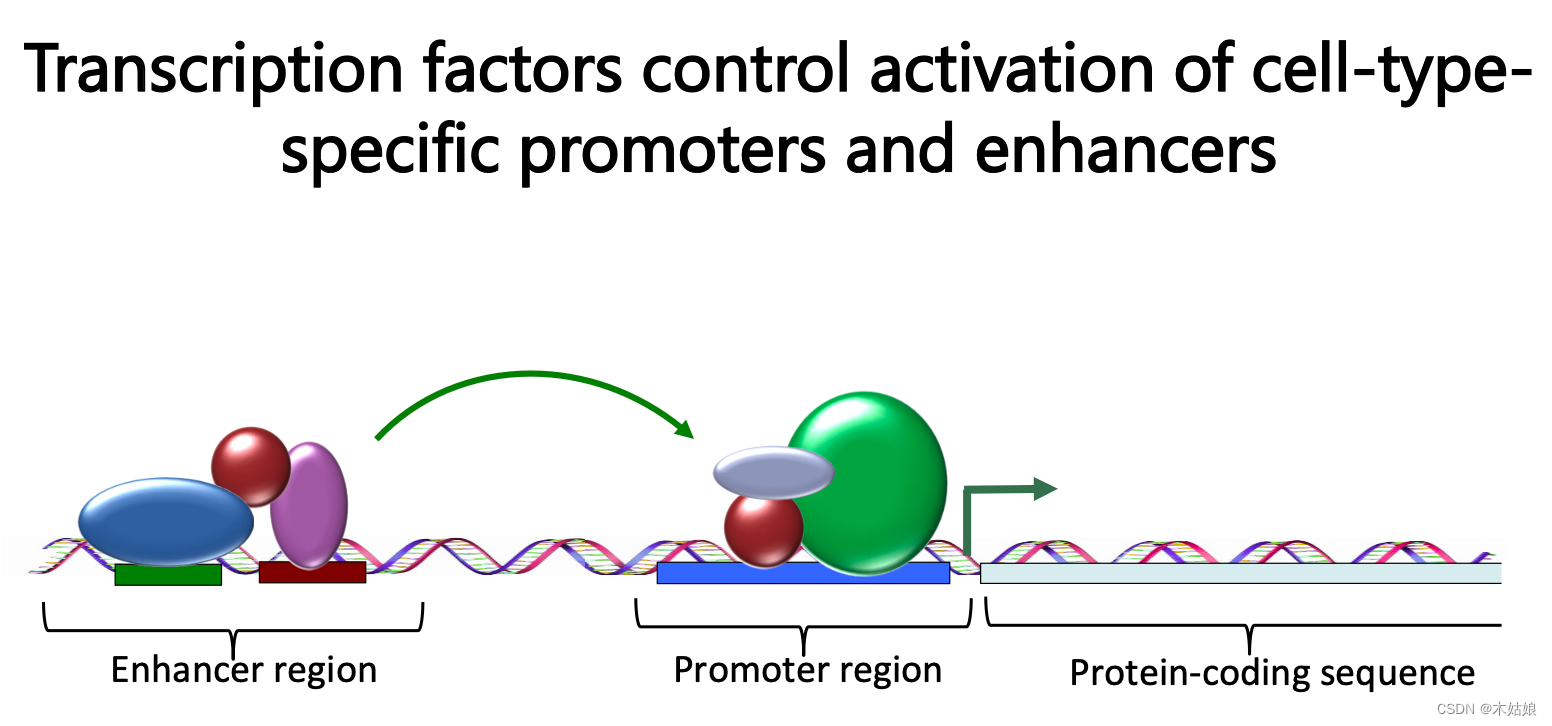
表观基因组学–体内每个细胞类型的编程方法,通过调整压缩特定的DNA序列启动子区域的特征。
启动子:RNA聚合酶可以结合,并转录DNA的部分。细胞表达开始启动的区域
转录因子利用DNA结合域识别基因组中的特定DNA序列
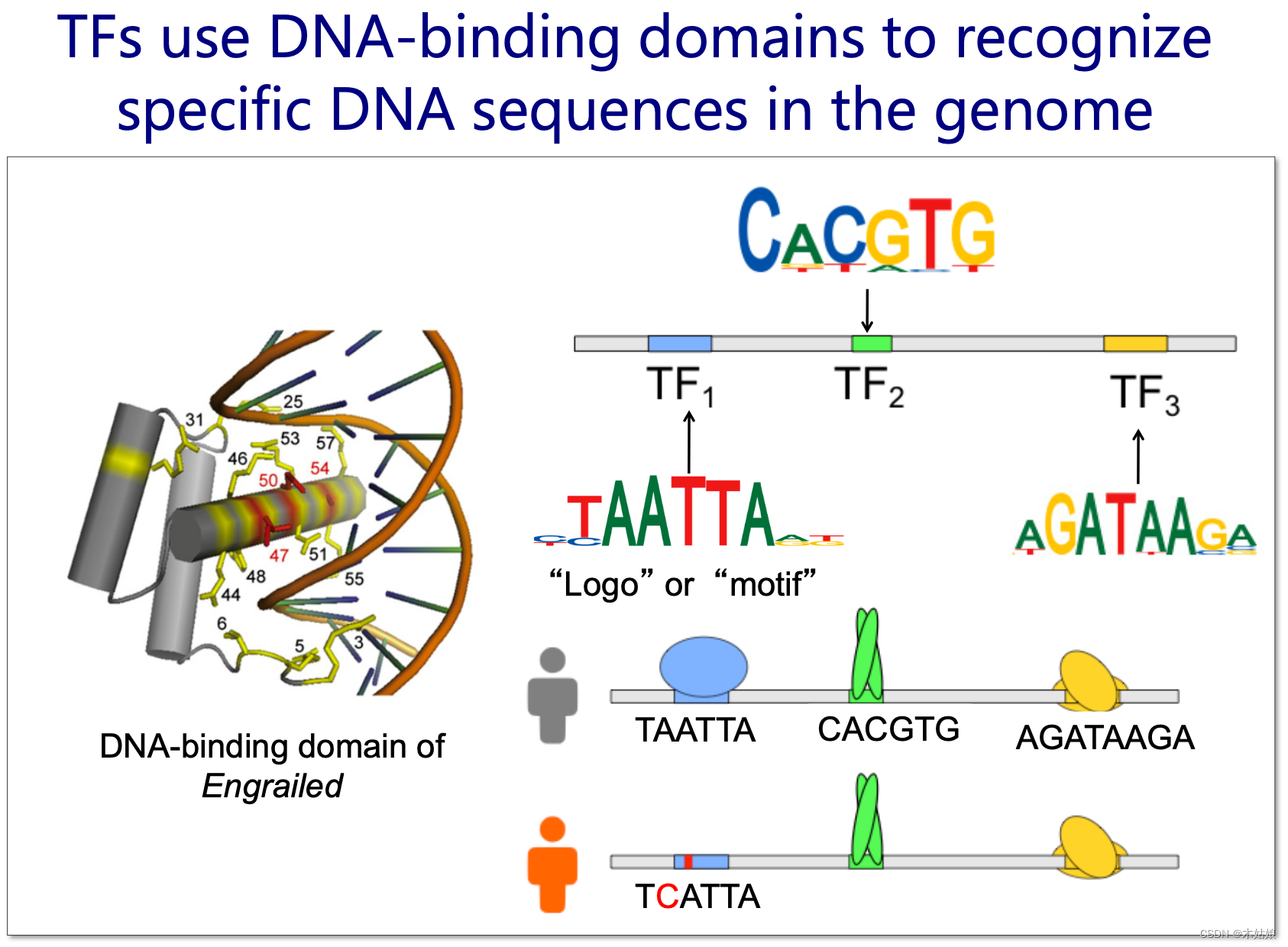
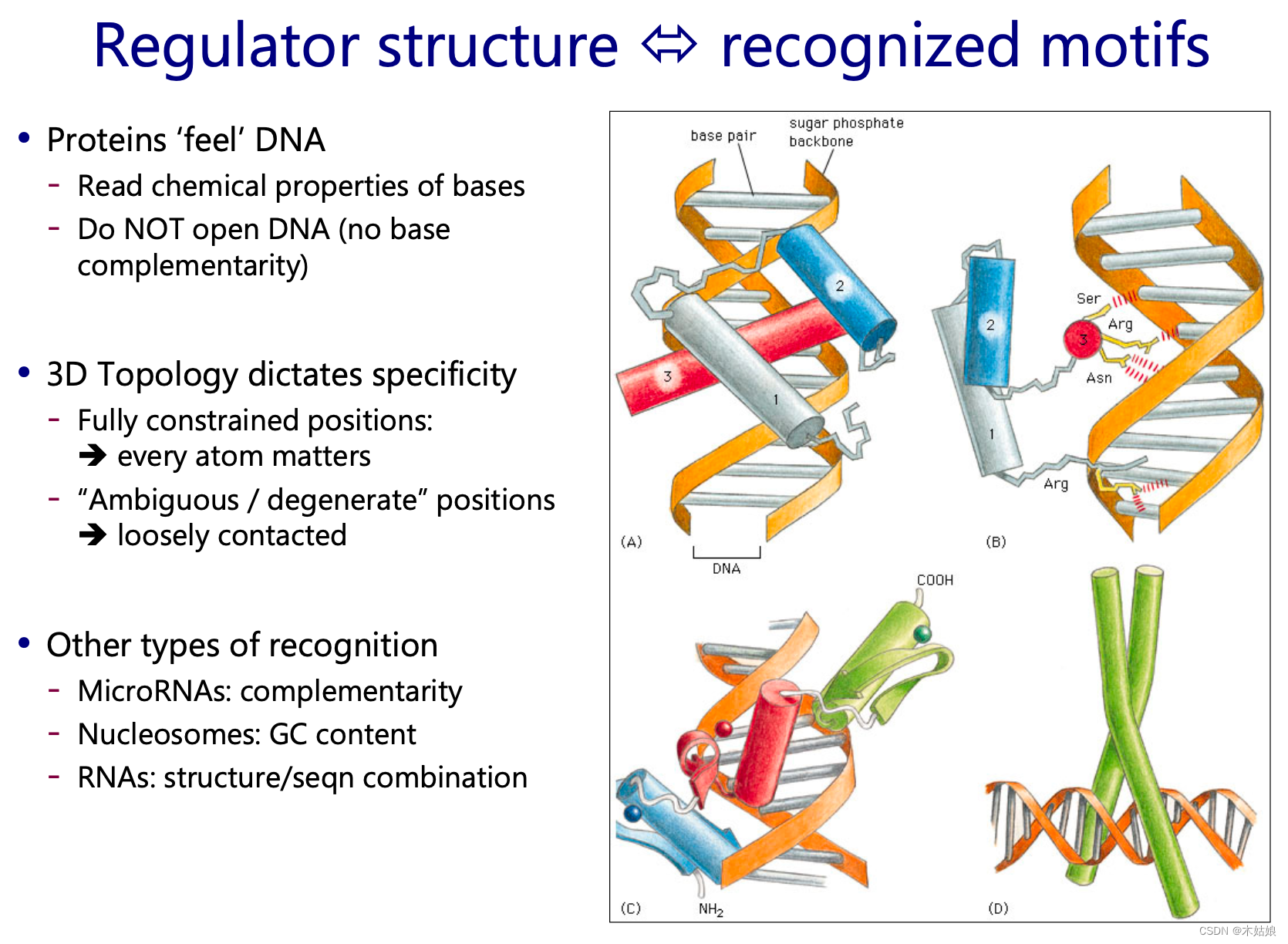
每个蛋白质都有着不同的结构,蛋白质之间的结合没有像DNA,RNA之间一样的互补性,因此他们需要通过binding motifs
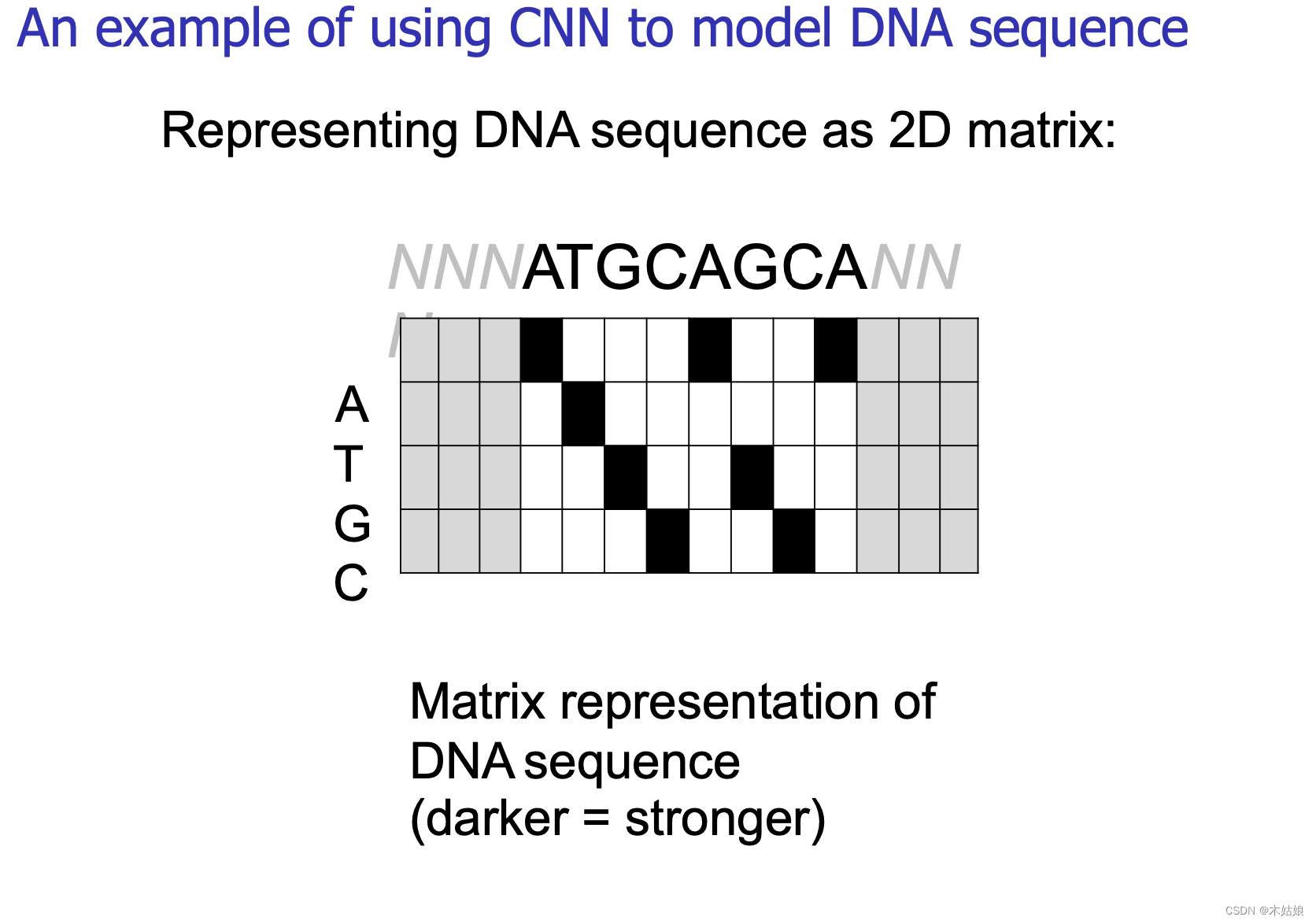
DNA 的表示方法:把所有结合点堆起来,然后建立矩阵,表达DNA的特异性。字母的高度表示信息量

1.2 Probing gene regulation: TFs/histones: ChIP-seq, Accessibility: DNase/ATAC-seq
- DNA转录:组蛋白:ChIP-seq
- DNA可及性(可访问性):一文读懂染色质可及性及ATAC-seq https://zhuanlan.zhihu.com/p/166500744
2 调控组学和基序发现的传统方法
2.1 基于Enrichment-based基序发现:EM算法、Gibbs Sampling
2.2 基于实验的方法:PBMs,SELEX. Comparative genomics:Evolutionary conservation
3 卷积神经网络在转录调控中的基础应用
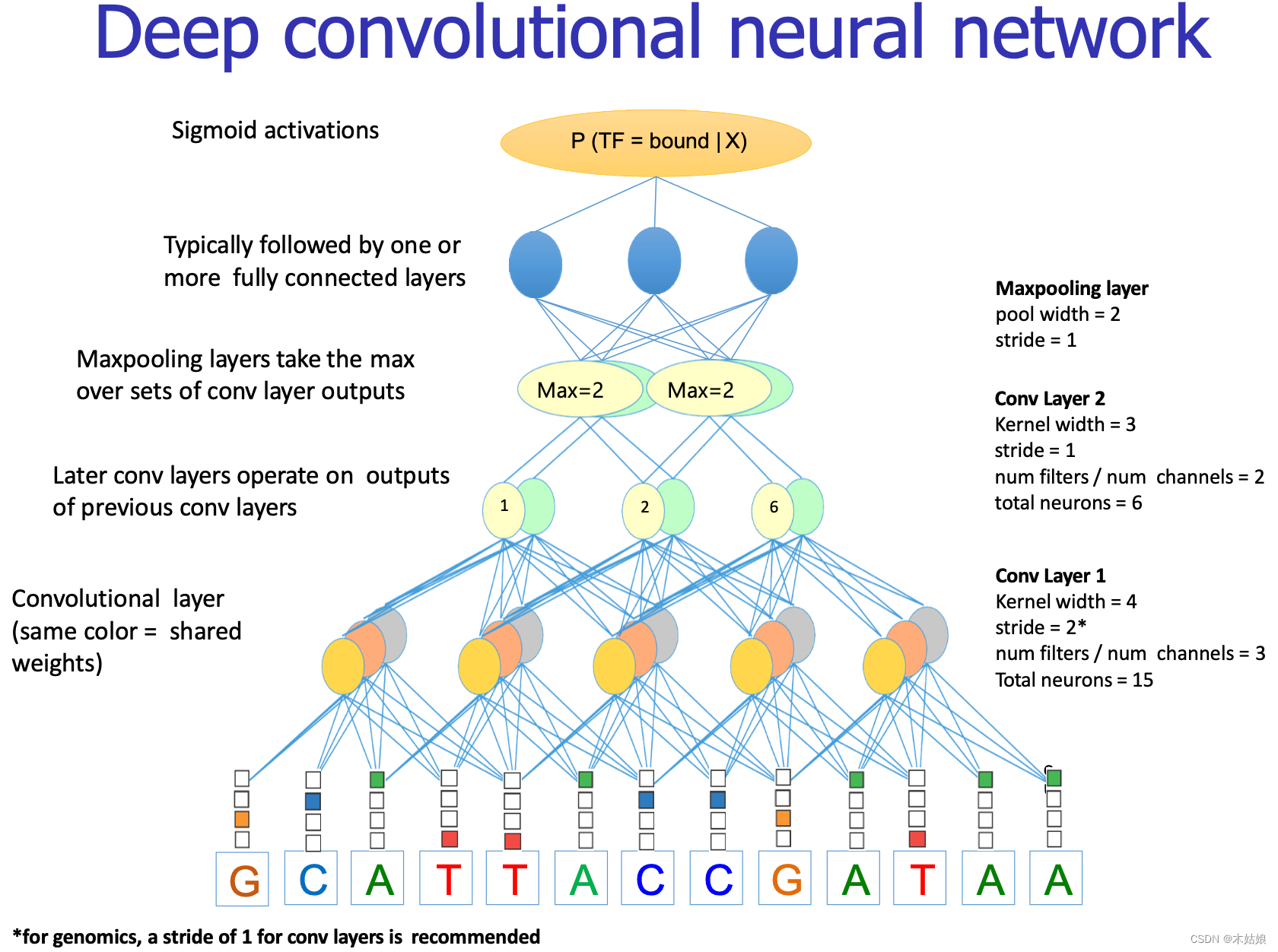
3.1 低维特征:主要想法:pixels<->DNA letters. Patches/filters<->Motifs. Higher<->combinations
将DNA序列表示为一个二维矩阵
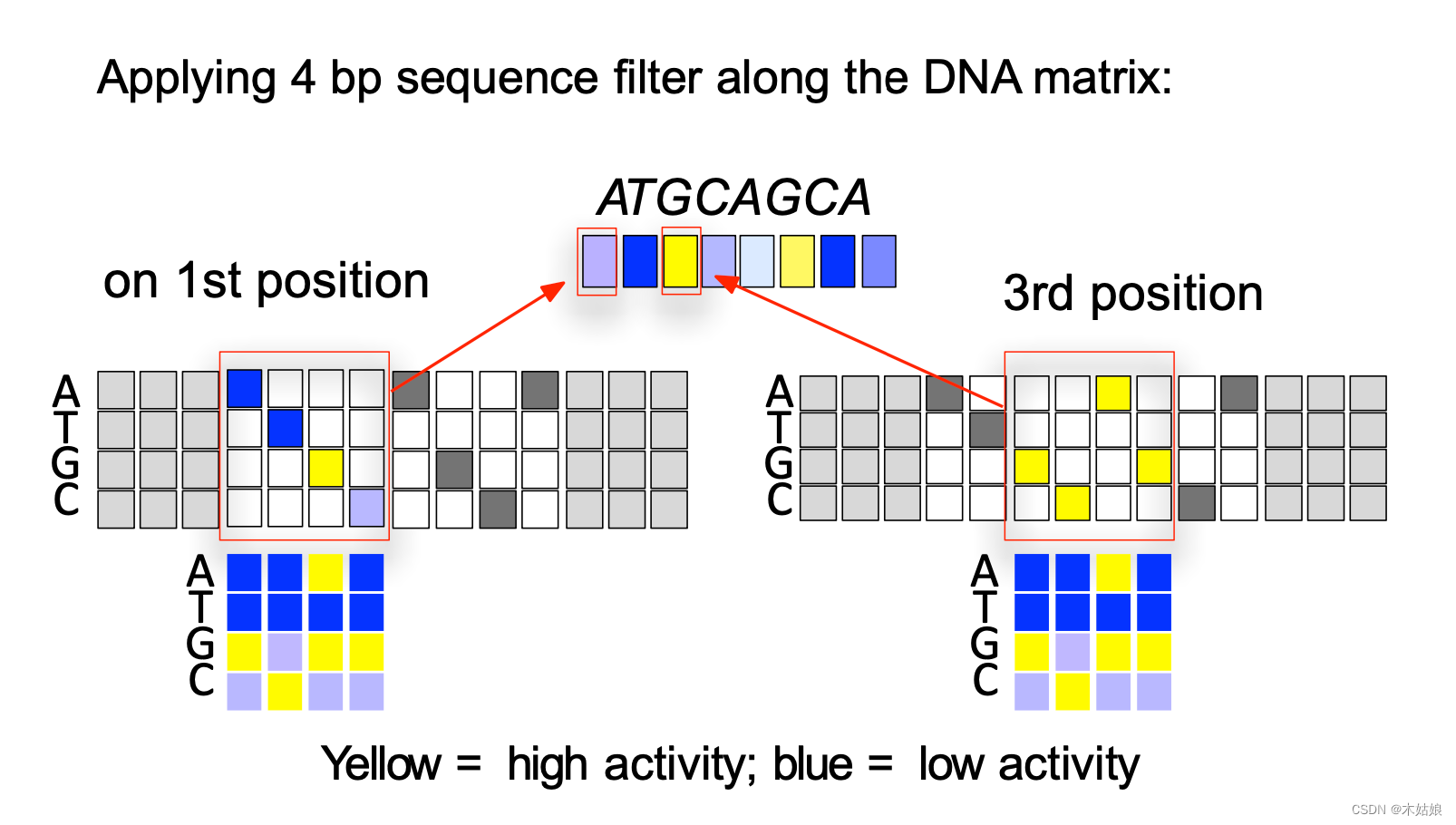
修正 = 忽略低于某些阈值的信号。
池化 = 按max或average汇总每个通道。
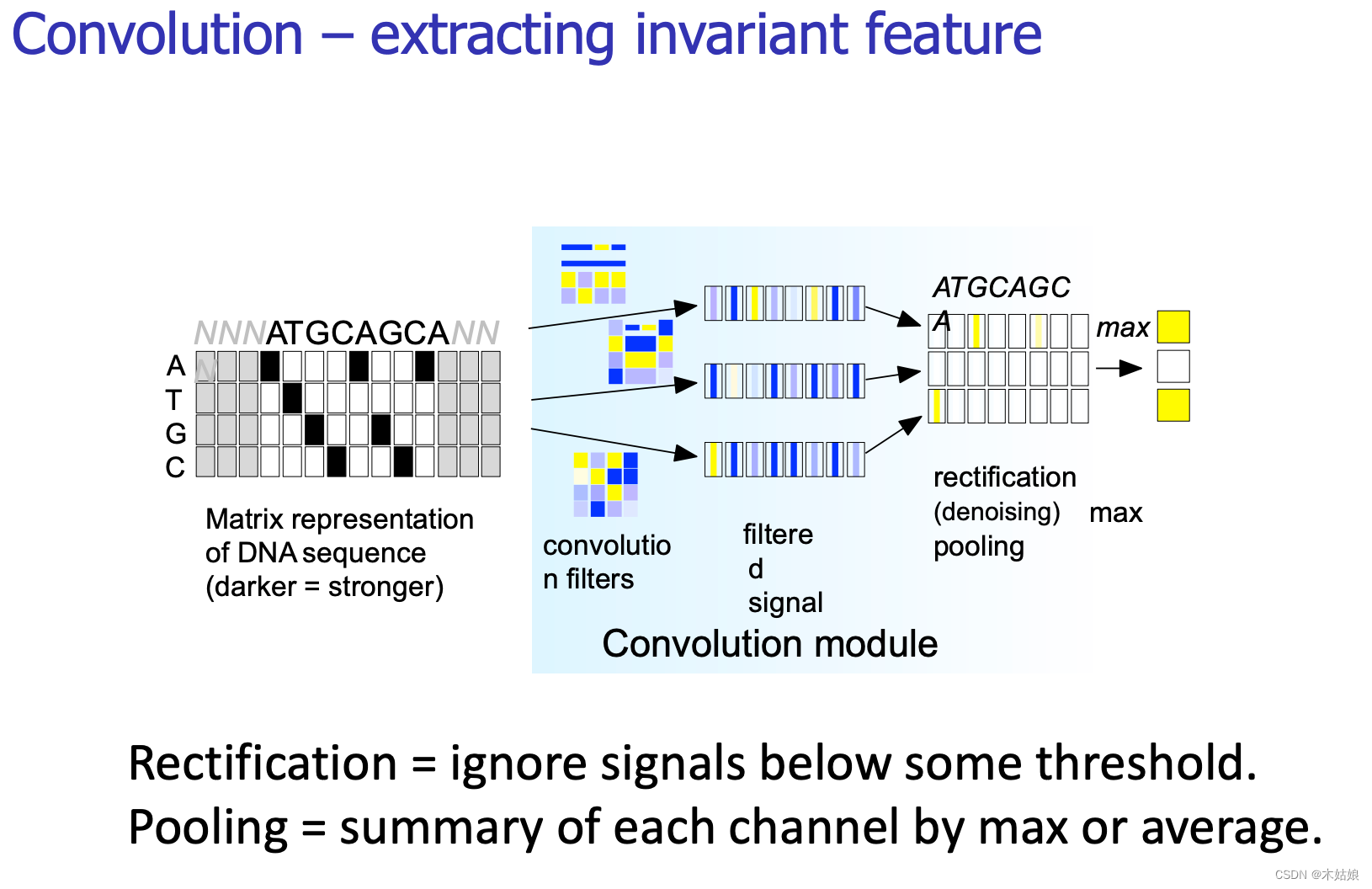
将提取到的特征输入到网络中进行预测
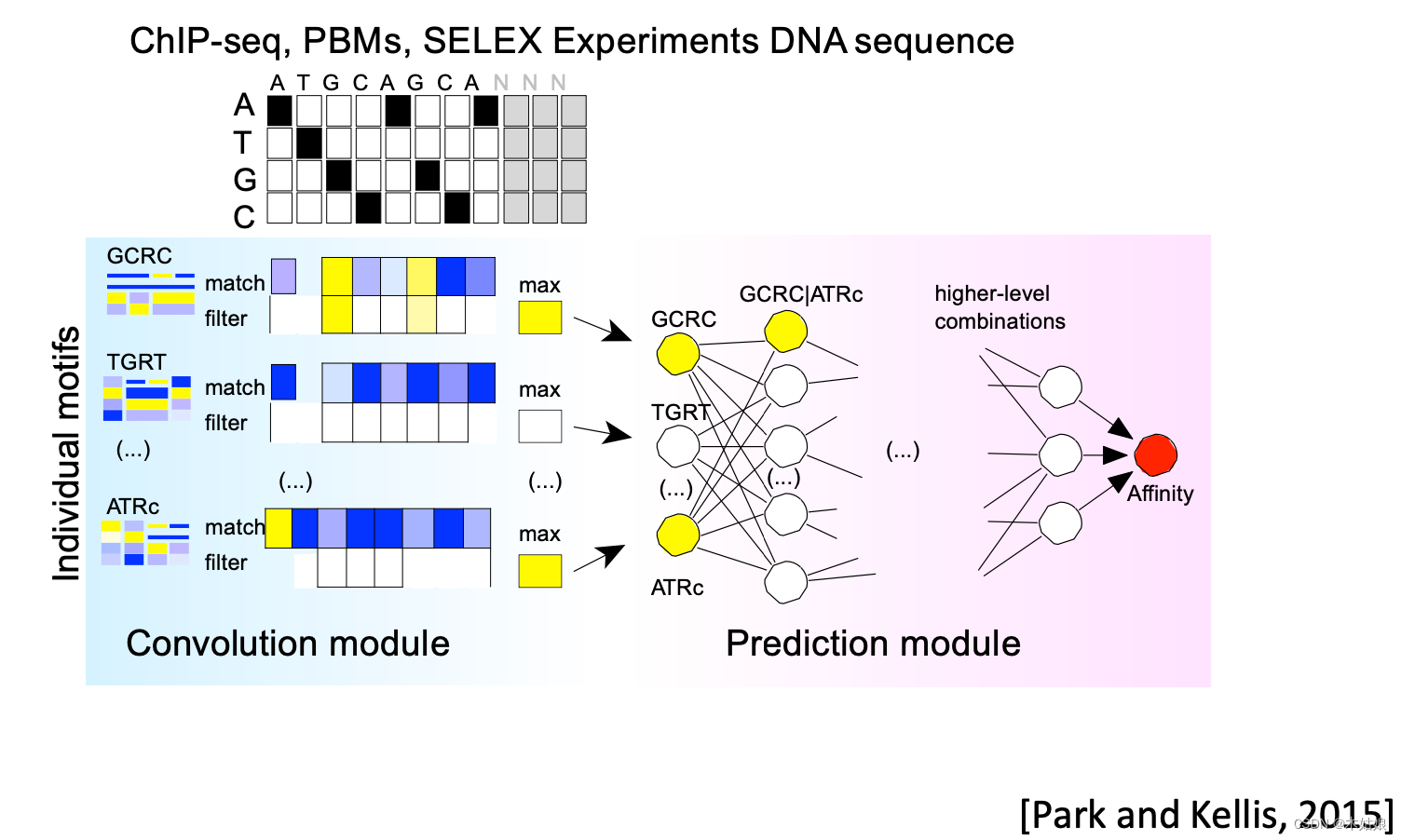
3.2 高维特征:学习卷积核<-> Motif discovery. Applying them<-> Motif matches
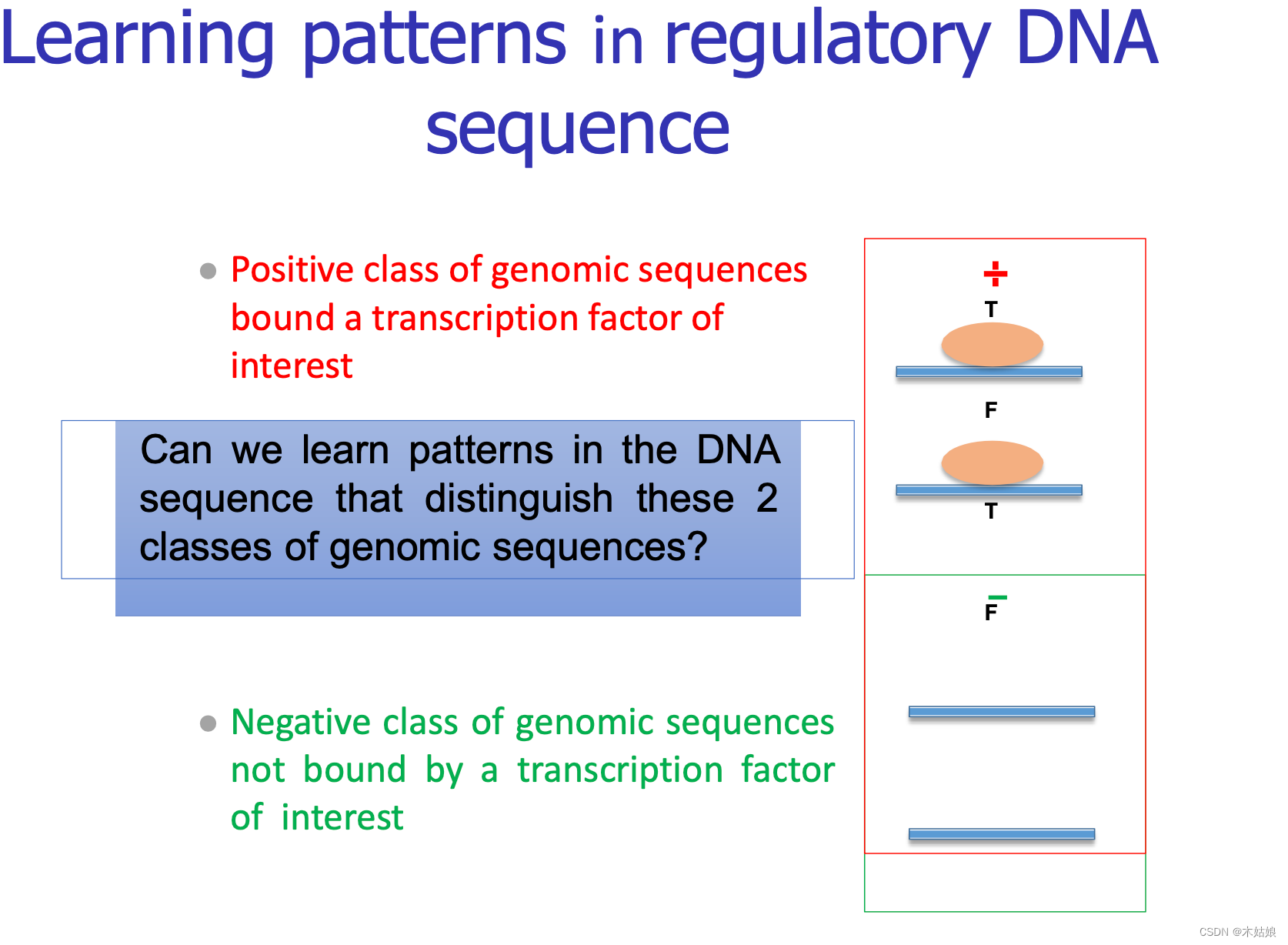
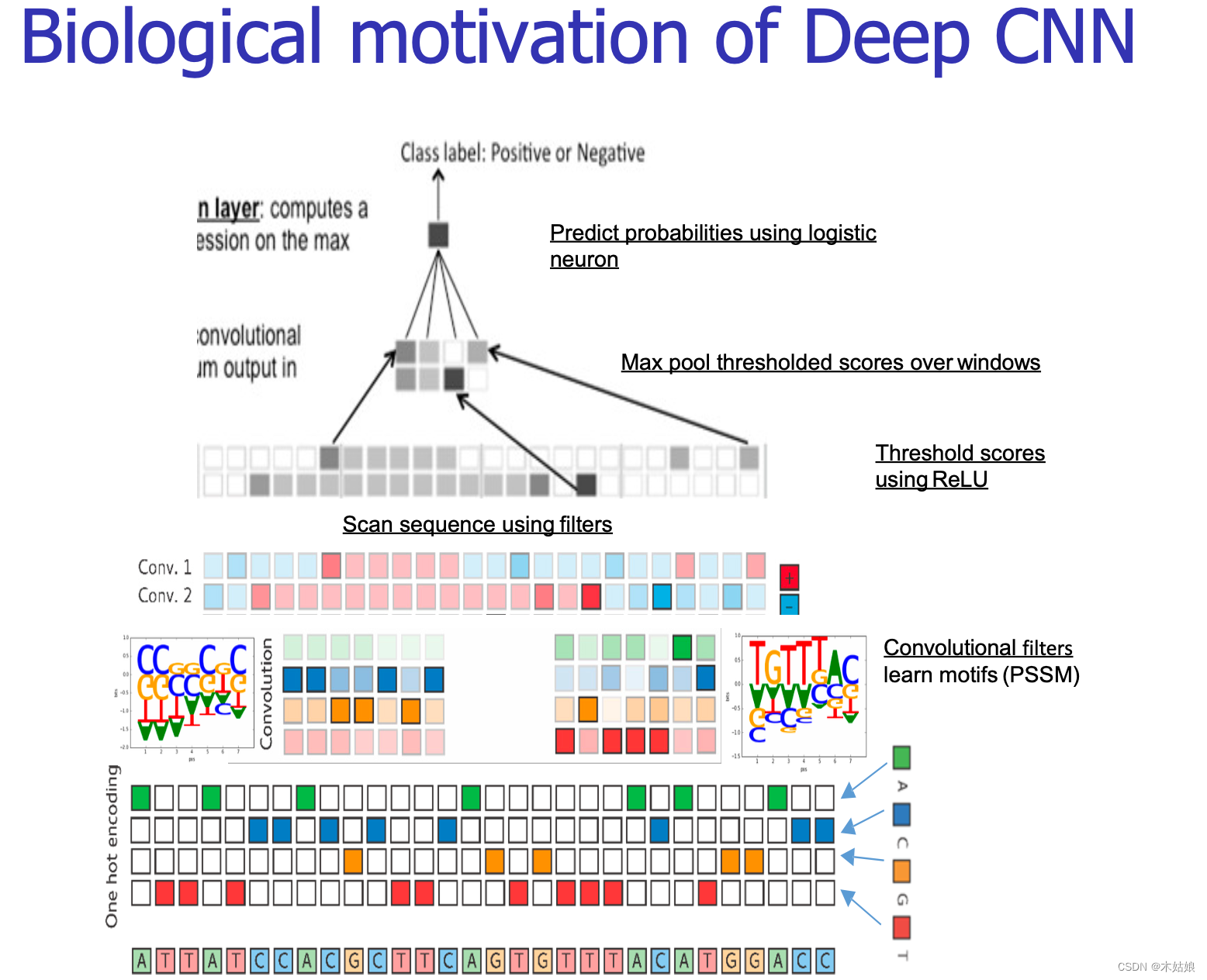
4 CNNS/RNNs在调控组学中的实际应用:各种架构
4.1 DeepBind: learn motifs, use in(shallow) fully-connected layer, mutations impact
链接: http://www.nature.com/nbt/journal/v33/n8/full/nbt.3300.html
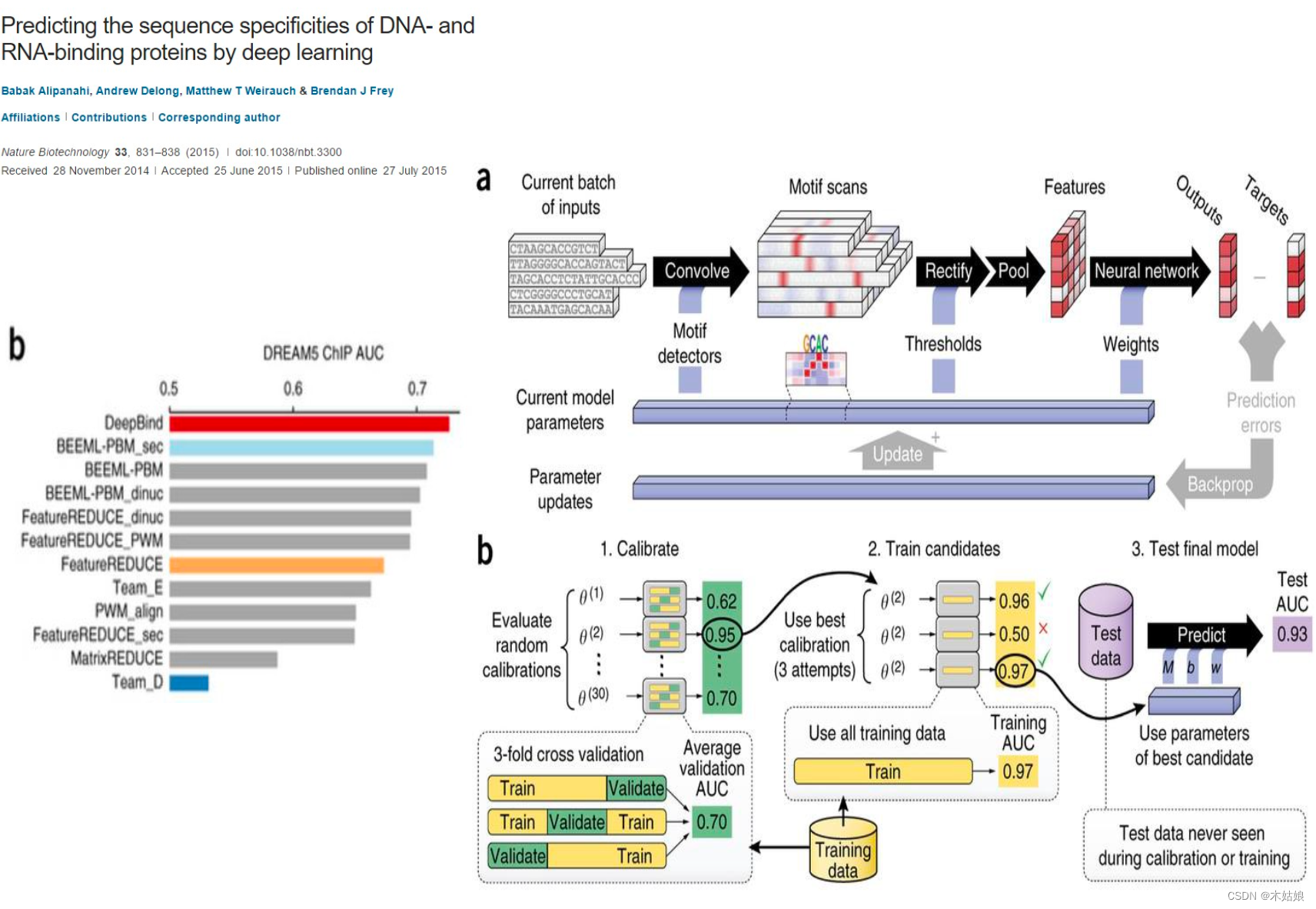
DeepBind summary
The key deep learning techniques:
- Convolutional learning
- Representational learning
- Back-propagation and stochastic gradient
- Regularization and dropout
- Parallel GPU computing especially useful for hyper-parameter search
Limitations in DeepBind:
- Require defining negative training examples, which is often arbitrary
- Using observed mutation data only as post-hoc evaluation
- Modeling each regulatory dataset separately
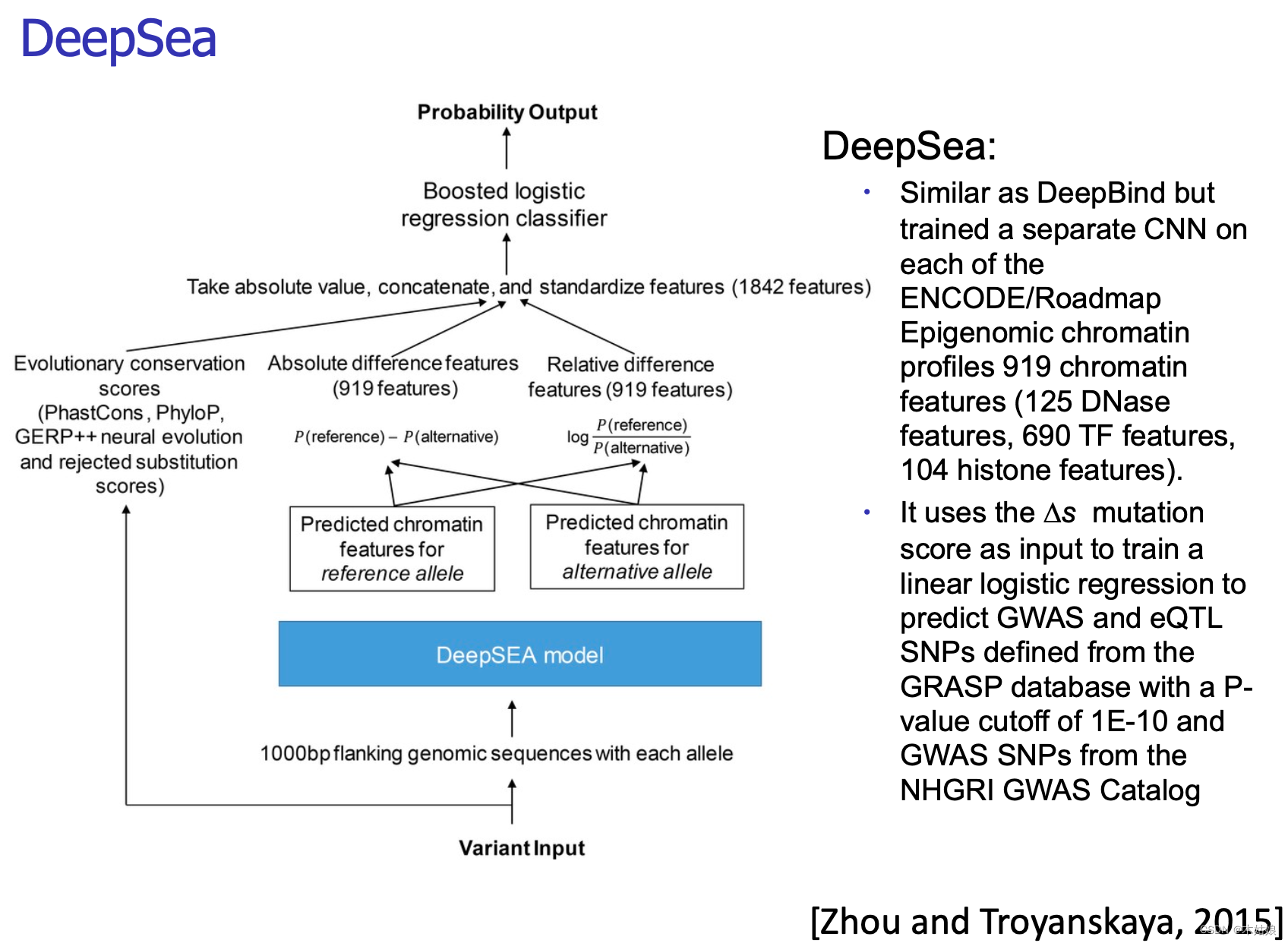
4.2 DeepSea: Train model directly on mutational impact prediction
4.3 Baseet: Multi-task DNase prediction in 164 cell types, reuse/learn motifs
Basset: Learning the regulatory code of the accessible genome with deep convolutional neural networks
4.4 ChromPuter: Multi-task prediction of different TFs, reuse partner motifs
4.5 DeepLIFT: Model interpretation based on neuron activation properties
5 讲座:斯坦福Anshul Kundaje,Deep Learning for Reg. Genomics
Deep learning at base-resolution revels cis-regulatory motif syntax
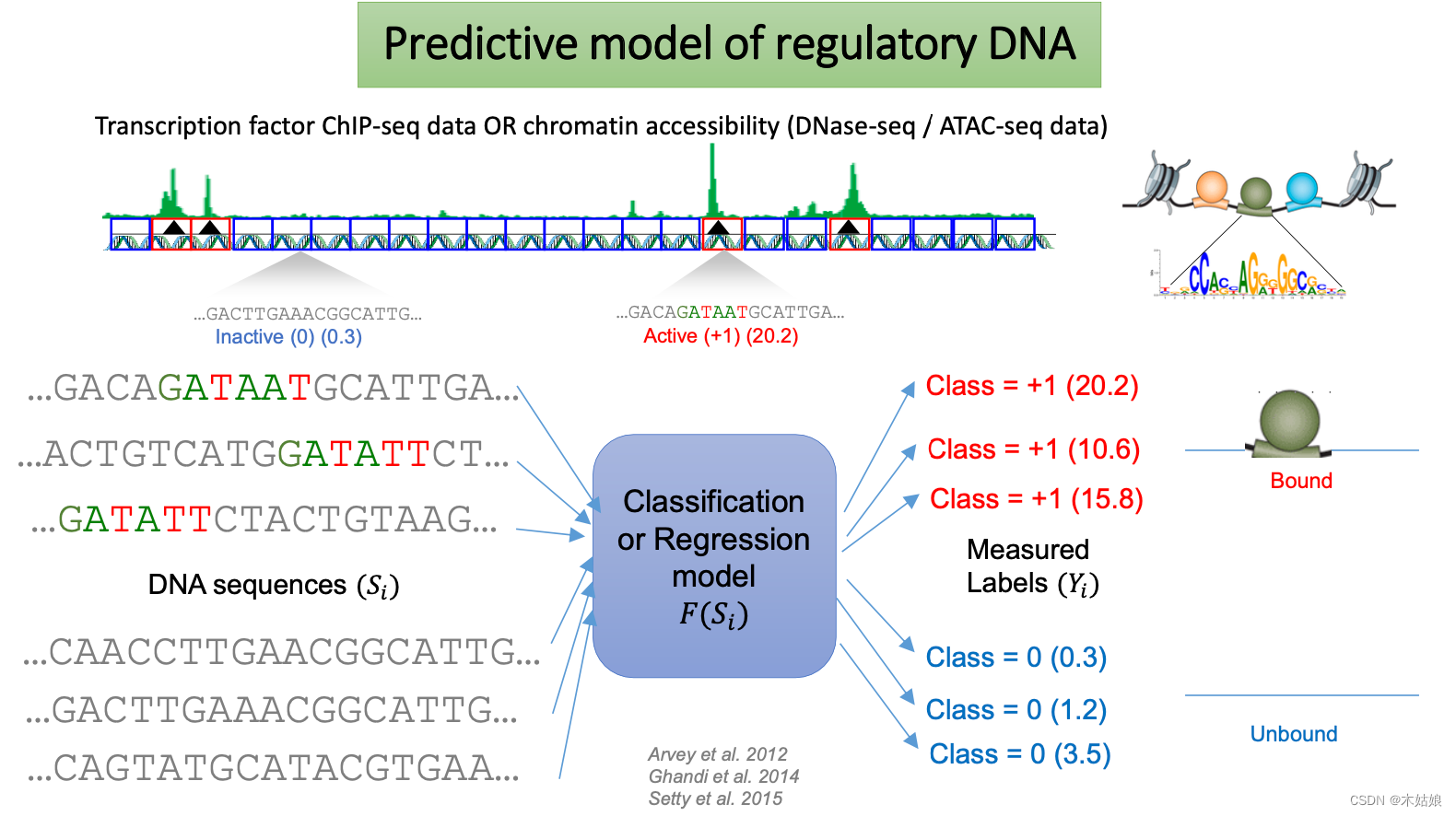
传统的做法:
使用传统测序方法:ATAC-seq / DNase-seq
机器学习方法:
利用转录因子或者染色质的可达性,表达峰值等作为有监督学习的标注
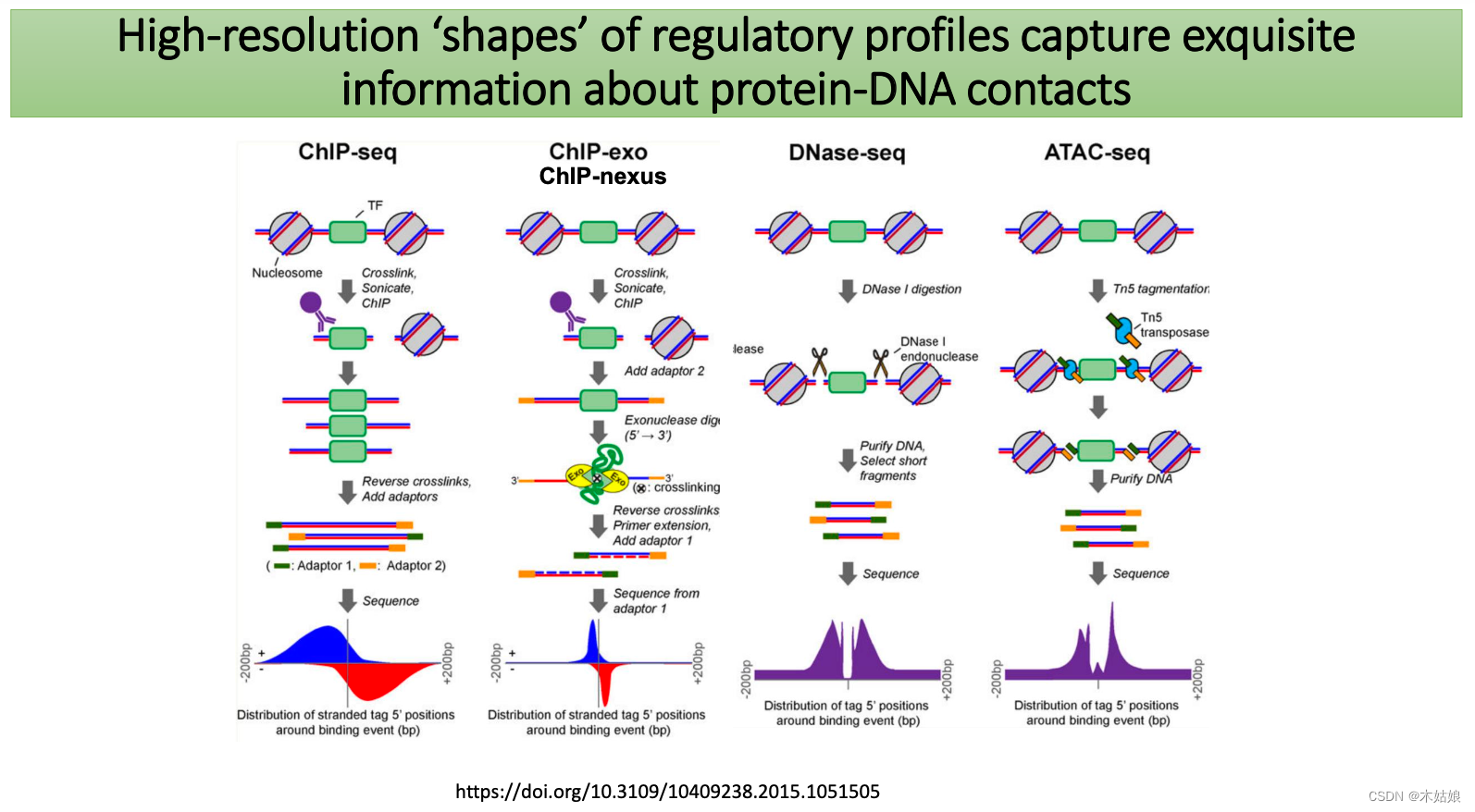
BPNet: DNA sequence to base-pair resolution profile regression
ChIP-exo/nexus: High resolution TF binding footprints
这篇关于MIT-6874-Deep Learning in the Life Sciences Week5的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!