本文主要是介绍生信格式 | BedGraph(基因组浏览器绘制),希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
一、特点及适用场景:
- 存放区间的坐标轴信息和相关评分(score)的文件,主要用于存储稀疏,不连续的数据
- 后缀名
.bedGraph - 一般UCSC不建议采用该格式作为基因组浏览器输入文件,因为考虑到数据集大小与索引构建,都不如 bigwig 更高效,尤其在如果bedGraph数据集非常大(超过5000万行 ),推荐转为 bigwig 文件
- 使用
WigTobigWig将 bedGraph 转换为bigWig 文件,文件格式转换详解 - 需要注意 bedGraph 文件不能转换为 wig 文件
二、格式
一共包含四列:
chromA chromStartA chromEndA dataValue
分别为:
- 染色体号
- 起始位点:染色体坐标从
0开始,这意味着第一个染色体位置为0,而长度为N的染色体的最后位置将为N-1。 - 终止位点
- 数据值:数据值可以是整数或实数,正值或负值。输入数据中列出的位置必须按数字顺序,并且仅会绘制指定位置的图。
参数
track type=bedGraph name=track_label description=center_label
visibility=display_mode color=r,g,b altColor=r,g,b
priority=priority autoScale=on|off alwaysZero=on|off gridDefault=on|off
maxHeightPixels=max:default:min graphType=bar|points viewLimits=lower:upper
yLineMark=real-value yLineOnOff=on|off
windowingFunction=maximum|mean|minimum smoothingWindow=off|2-16
三、例子
1、bedGraph 文件
下面的文件可以描述为:
在第19号染色体的49,302,001到49,304,701区域的三个轨迹中指定9个独立的数据点。
browser position chr19:49302001-49304701
browser hide all
browser pack refGene encodeRegions
browser full altGraph
# 300 base wide bar graph, autoScale is on by default == graphing
# limits will dynamically change to always show full range of data
# in viewing window, priority = 20 positions this as the second graph
# Note, zero-relative, half-open coordinate system in use for bedGraph format
track type=bedGraph name="BedGraph Format" description="BedGraph format" visibility=full color=200,100,0 altColor=0,100,200 priority=20
chr19 49302000 49302300 -1.0
chr19 49302300 49302600 -0.75
chr19 49302600 49302900 -0.50
chr19 49302900 49303200 -0.25
chr19 49303200 49303500 0.0
chr19 49303500 49303800 0.25
chr19 49303800 49304100 0.50
chr19 49304100 49304400 0.75
chr19 49304400 49304700 1.00
2、粘贴上面的代码,点 Submit
https://genome.ucsc.edu/cgi-bin/hgCustom

3、绘制轨迹
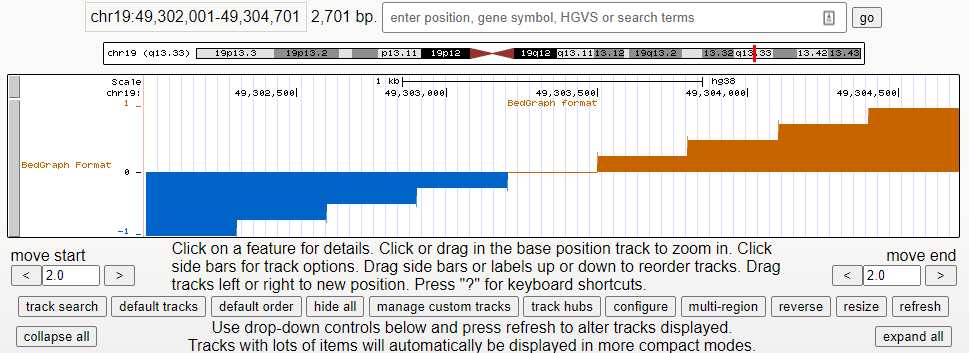
上面的示例是一个定制轨迹,其中包括
track type=一行特定于在浏览器中加载数据的行。此行将导致原始bedGraph数据文件无法通过
validateFiles浏览器外部的其他工具进行验证。
这篇关于生信格式 | BedGraph(基因组浏览器绘制)的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







