jcvi专题
「JCVI教程」如何绘制CNS级别的共线性图(中)
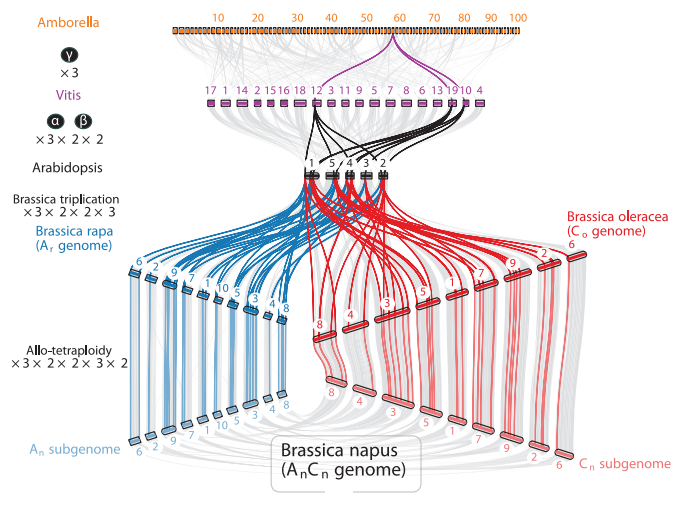
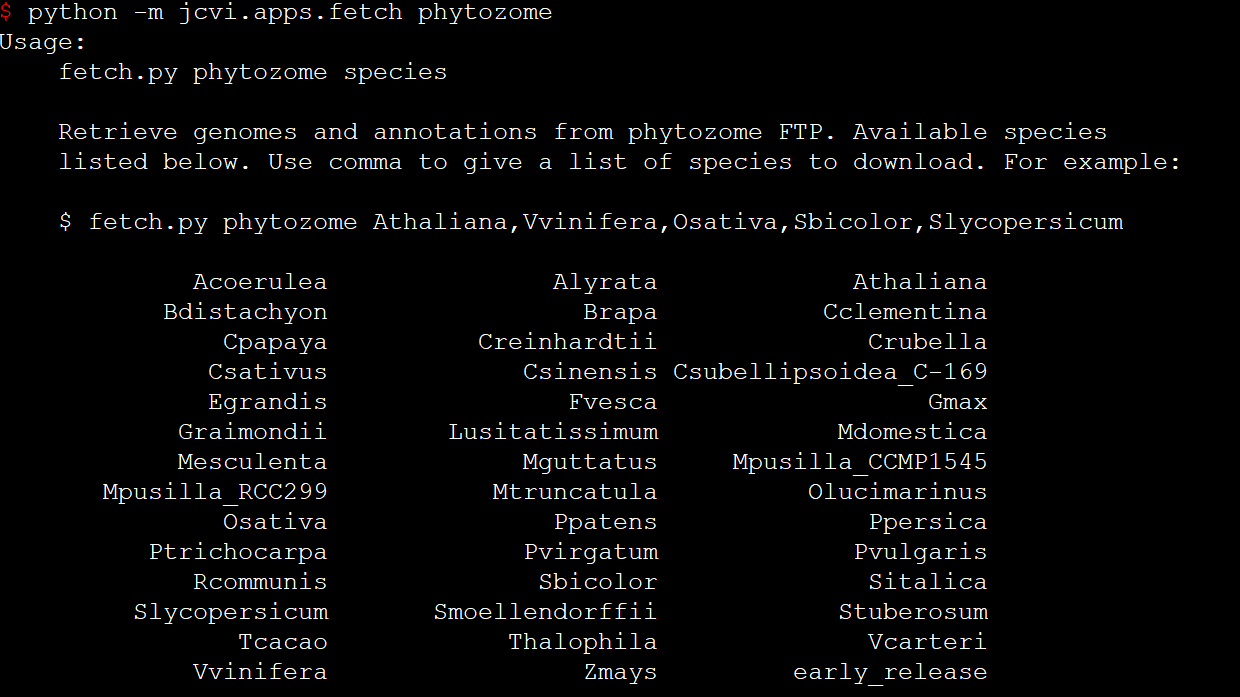
在「JCVI教程」编码序列或蛋白序列运行共线性分析流程(上)还是有一个尴尬的事情,就是只用到两个物种,不能展示出JCVI画图的方便之处,因此这里参考https://github.com/tanghaibao/jcvi/wiki/MCscan-(Python-version)的分析,只不过画图部分拓展下思路。 首先要运行如下代码获取目的数据 python -m jcvi.apps.fetch p
「JCVI教程」如何绘制CNS级别的共线性图(上)
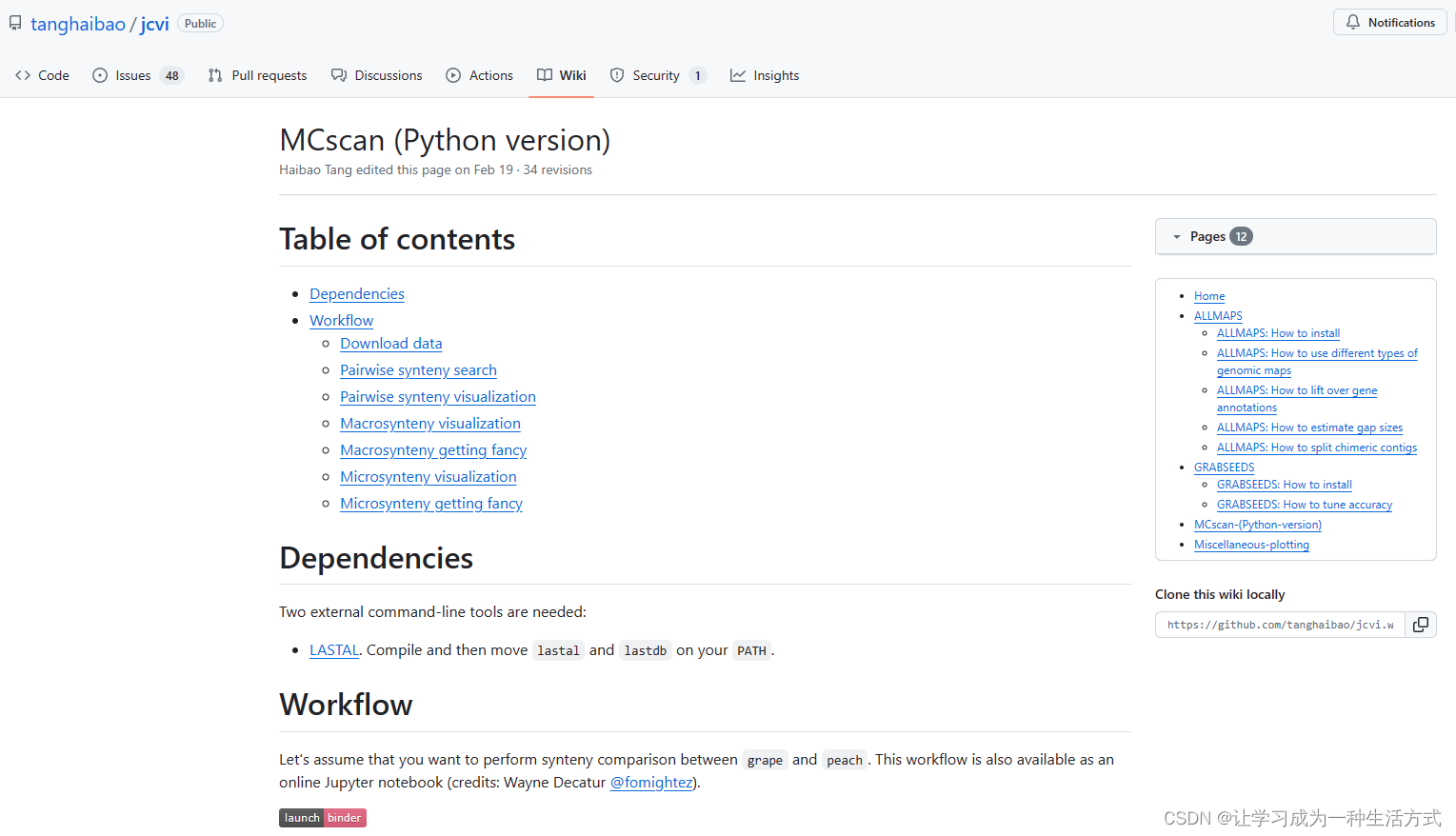
本教程借鉴https://github.com/tanghaibao/jcvi/wiki/MCscan-(Python-version). 我们先从http://plants.ensembl.org/index.html选择两个物种做分析, 这里选择的就是前两个物种,也就是拟南芥和水稻(得亏没有小麦和玉米) 选择物种 我们下载它的GFF文件,cdna序列和蛋白序列 #A
「JCVI教程」如何基于物种的CDS的blast结果绘制点图(dotplot)
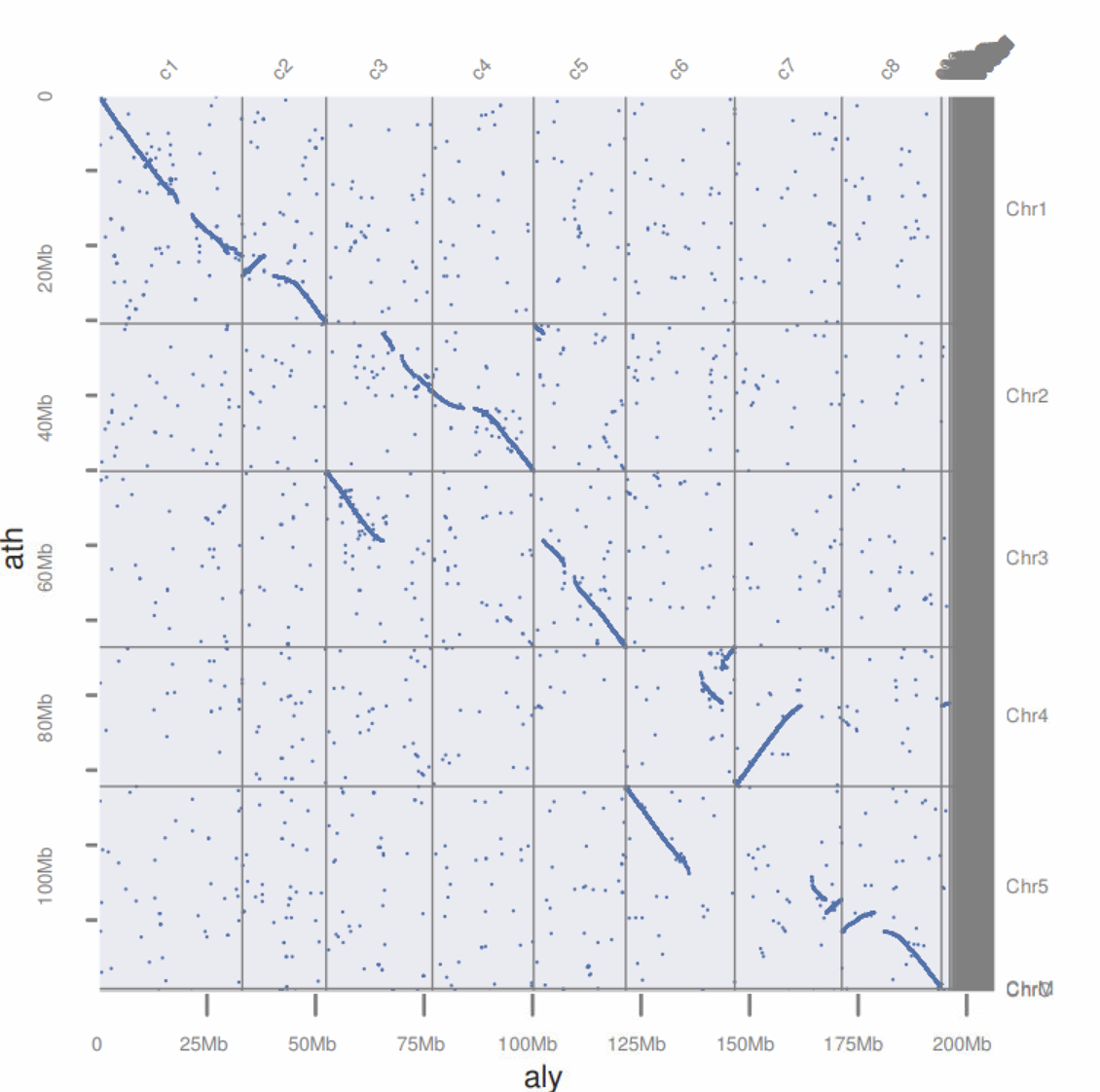
这是唐海宝老师GitHub上的JCVI工具的非官方说明书。 该工具集的功能非常多,但是教程资料目前看起来并不多,因此为了能让更多人用上那么好用的工具,我就一边探索,一边写教程 这一篇文章教大家如何利用JCVI里面的工具绘制点图,展现两个物种之间的共线性关系。 在分析之前,你需要从PhytozomeV11 下载A.thaliana和Alyrata的CDS序列,保证文件夹里有如下内容 Al
jcvi.apps里的fetch模块使用介绍
这是唐海宝老师GitHub上的JCVI工具的非官方说明书。 该工具集的功能非常多,但是教程资料目前看起来并不多,因此为了能让更多人用上那么好用的工具,我就一边探索,一边写教程 fetch模块里封装了好几个数据库的下载方式 bisect: 搜索给定accession的版本,(目前没想到怎么用)ensembl: 从ENSEMBL上下载基因组和注释entrez: 从ENTREZ里获取记录phy
「JCVI教程」JCVI的模块简介
JCVI的使用基本格式为python -m jcvi.模块.子功能 实际操作,因此了解JCVI的模块能够帮助我们去找到需要的功能。根据JCVI的源代码,我们可以知道它目前一共有10个模块,如下 algorithms: 算法模块 algorithms annotation: 注释模块 annotation assembly: 组装模块 assemb
「JCVI教程」如何在Linux下安装JCVI
JCVI是利用Python编写的,能用于基因组组装、注释和比较基因组学分析。之前写过几篇介绍它的使用,这篇单独介绍它的安装。此外,虽然JCVI最早是由Python2编写,但是目前基本上已经迁移到Python3。 先提供最简单的conda安装方法,也就是conda create -c bioconda -n jcvi jcvi, 新建一个JCVI环境用于安装JCVI,之后通过conda activ
JCVI-筛选blast最佳结果(生物信息学工具-015)
通常,大家会问我们经过了NR注释,SwissProt注释,那么如何进行,如何挑选最佳比对结果? 同理,存在一个问题,如何挑选最佳的blast比对结果?什么事最优的同源序列? 唐海宝老师开发的工具jcvi(jcvi.formats.blast)解决了这一问题,基本上jcvi等价于MCscan。 01 安装 普通安装需要安装许多依赖,由于服务器等配置不能轻易修改,所以我们采用最便捷的方
「JCVI教程」JCVI的模块简介
JCVI的使用基本格式为python -m jcvi.模块.子功能 实际操作,因此了解JCVI的模块能够帮助我们去找到需要的功能。根据JCVI的源代码,我们可以知道它目前一共有10个模块,如下 algorithms: 算法模块 algorithms annotation: 注释模块 annotation assembly: 组装模块 assemb