本文主要是介绍jcvi.apps里的fetch模块使用介绍,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
这是唐海宝老师GitHub上的JCVI工具的非官方说明书。
该工具集的功能非常多,但是教程资料目前看起来并不多,因此为了能让更多人用上那么好用的工具,我就一边探索,一边写教程
fetch模块里封装了好几个数据库的下载方式
- bisect: 搜索给定accession的版本,(目前没想到怎么用)
- ensembl: 从ENSEMBL上下载基因组和注释
- entrez: 从ENTREZ里获取记录
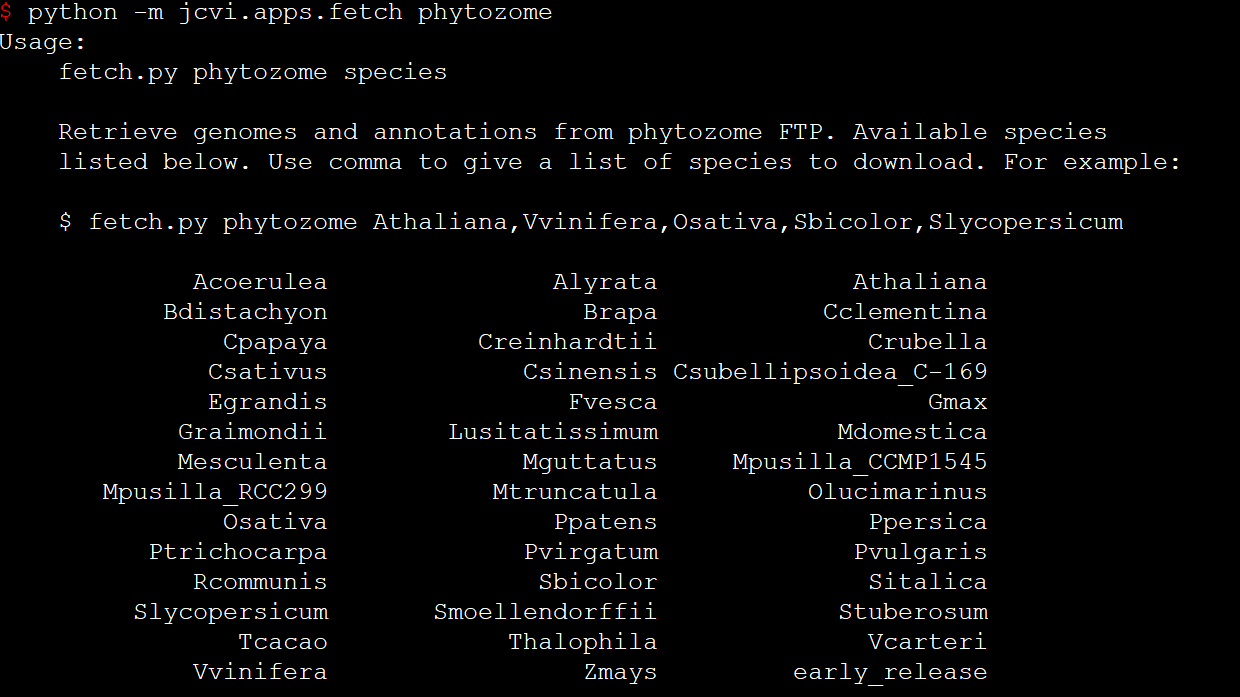
- phytozome: 从phytozome的FTP里下载基因组和注释
- pytozome10: 基于Globus的API从phytozome里下载基因组和注释, 需要额外配置
- sra: 调用wget从SRA的FTP里下载数据(感觉不如直接调用prefetch)
其中ensembl和phytozome是我比较喜欢的工具,因为它可以让我轻松地去下载一些注释得比较好的基因组。而且用法也很简单,直接输入python -m jcvi.apps.fetch ensembl或python -m jcvi.apps.fetch phytozome就会告诉你你可以下载哪些基因组,
这篇关于jcvi.apps里的fetch模块使用介绍的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!






