bed专题
全外显子测序分析流程3 - Exon.Interval.bed文件生成和BAM文件标记重复
全外显子测序分析流程3 - Exon.Interval.bed文件生成和BAM文件标记重复 分析流程步骤其他相关文章: Python处理生信分析流程配置文件4种方法 全外显子测序分析流程1 - Fastq质控与去接头、低质量和引物序列 全外显子测序分析流程2 - BWA-MEM比对到参考基因组与BAM统计 1. 封装流程特点 python封装, 参数控制配置文件设置核心参数,便于全流程
资料总结分享:SAM,bam,bed文件格式
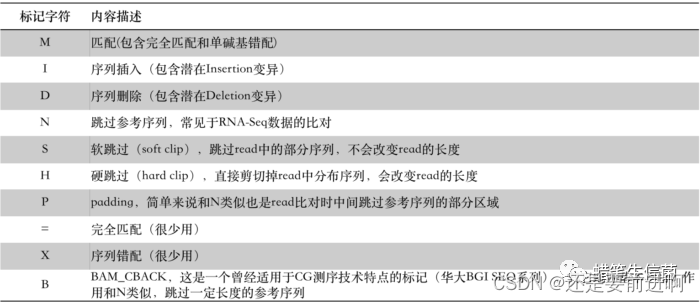
目录 sam文件 bam文件 bed 文件 sam文件 SAM(Sequence Alignment/Map)文件是存储测序数据比对结果的一种常见格式。SAM文件通常用于存储DNA或RNA测序数据在参考基因组上的比对结果。 SAM文件由多行文本组成,每一行代表一个比对结果。SAM文件中的每一行包含了比对的序列ID、比对的标志、参考序列的名称、序列的起始位置、比对得分、序列的序
生物信息数据格式:bed格式
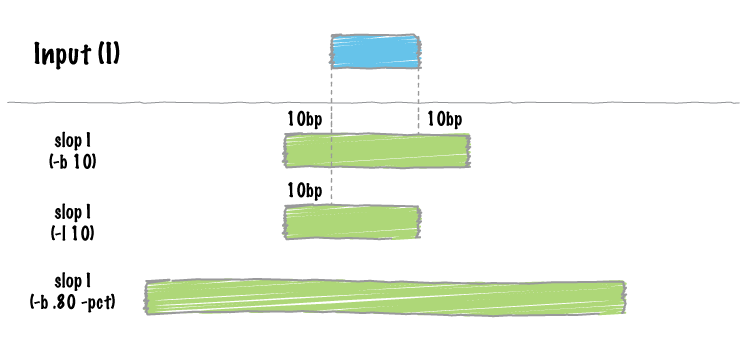
文章目录 BED format(基因组的注释文件)基本列附加列示例[Bedtools简介](https://bedtools.readthedocs.io/en/latest/index.html)下载安装演示版的bed文件 (demo.bed)我们的基因组文件(genome.txt)[bedtools slop](http://bedtools.readthedocs.io/en/late
输出bed格式,并用bedtools能够自动识别并转换正负链
往TraesCS6B03G0578500.1.bed写入文件 Chr6B IWGSC_v2.1 exon 299893989 299894056 100 - . ID=TraesCS6B03G0578500.1.exon2;Parent=TraesCS6B03G0578500.1;Name=TraesCS6B02G215300.1.exon1;Target=TraesCS6B02G215300.