本文主要是介绍Echarts绘制差异代谢产物分类与KEGG通路分类的旭日图,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
文章目录
- 前言
- 数据准备工作
- 绘图
- excel旭日图的绘制
- ECharts旭日图的绘制
- ECharts旭日图
- 菜鸟编辑器修改后的旭日图
- 附演示代码
前言
整理实验数据,进行可视化,旭日图有利于简洁明了的展示多层次结构
数据准备工作
- 对公司返回回来的代谢组结果进行整理,在MSMS二级质谱结果中找到需要分析组别的csv格式文件,直接用excel打开(p值过滤后的数据)
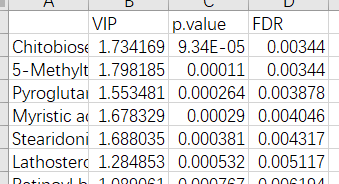
- 在总表的结果文件中找到全部物质的KEGG信息和HMDB信息
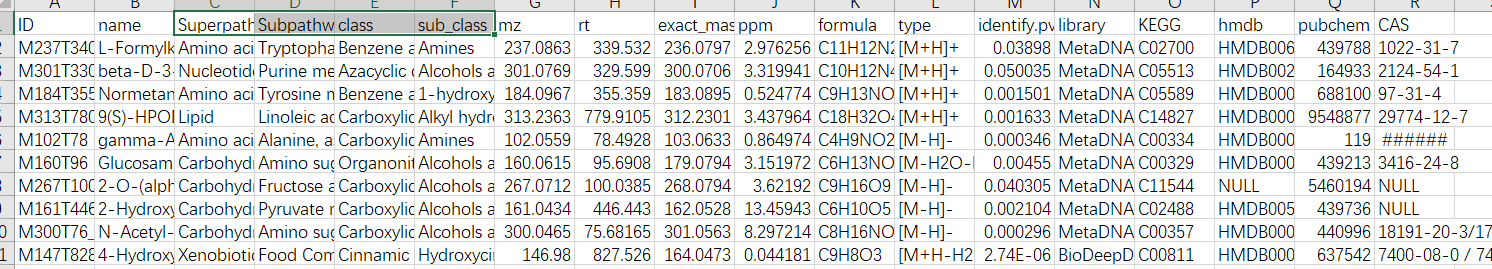
-
利用
vlookup函数对目的分组数据进行所需信息的匹配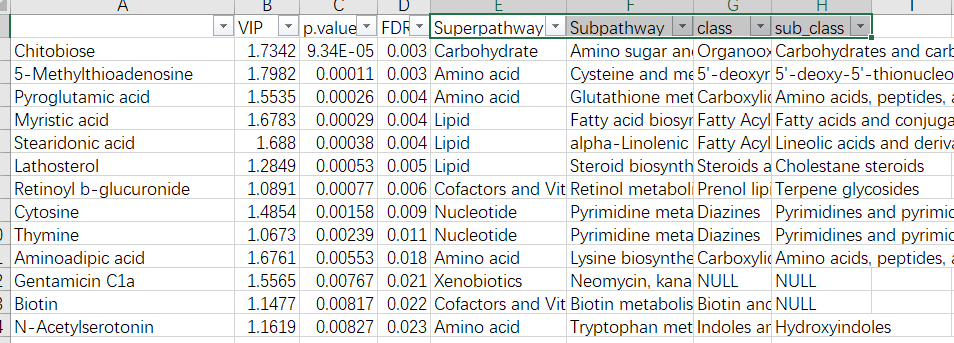
-
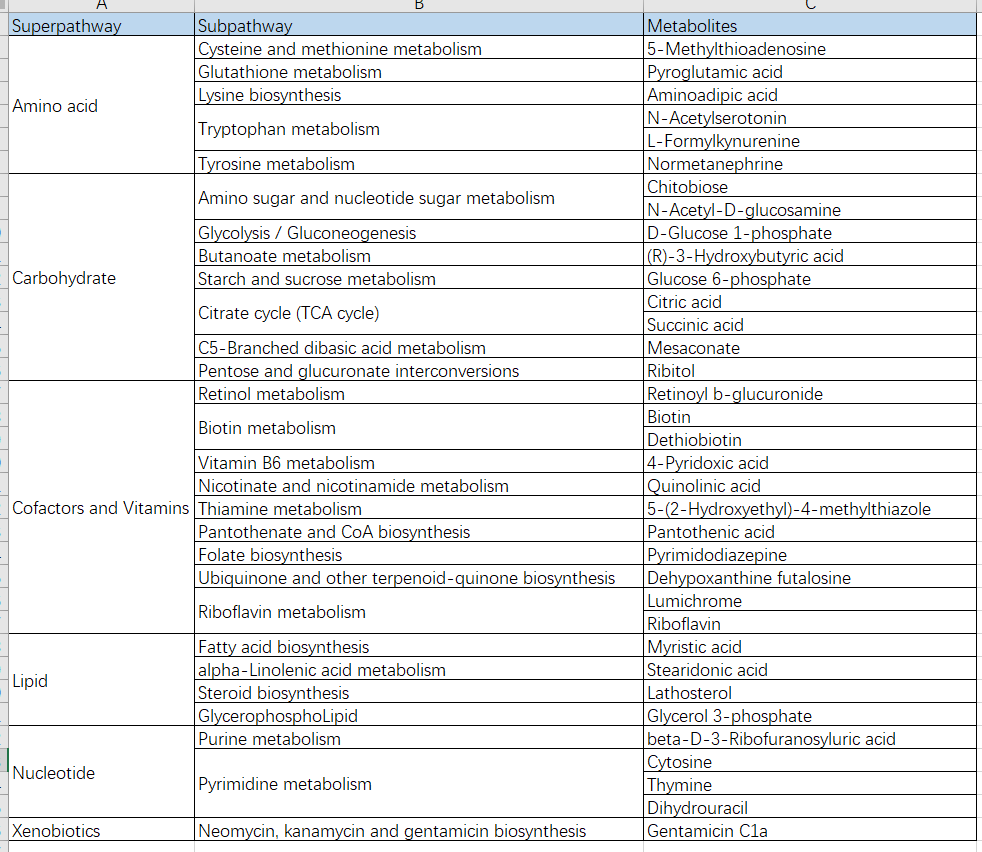
对匹配后的物质进行手动筛选分类整理
绘图
excel旭日图的绘制
- 对上面分类汇总的数据设置如下(不要合并单元格,最后一列一定要赋值)
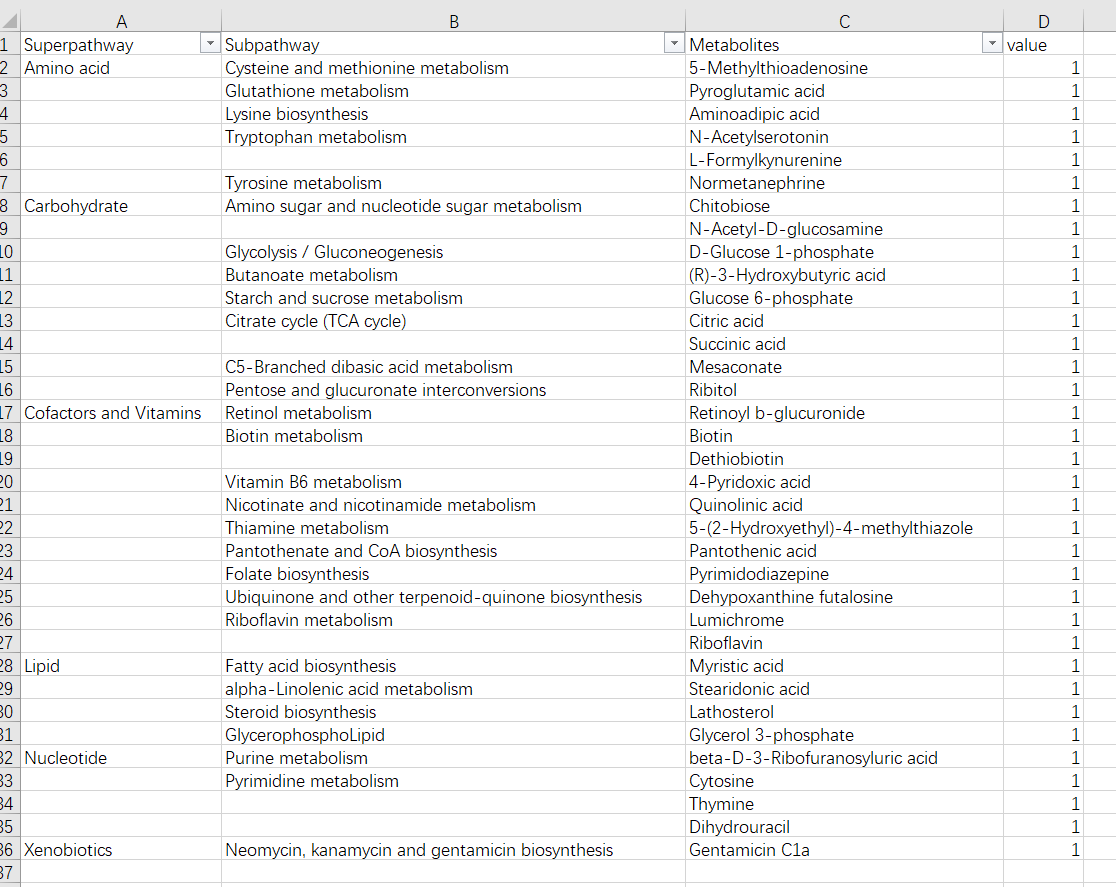
- 选中A列到D列区域的有效数据,
插入-插入层次结构图表-旭日图,生成如下
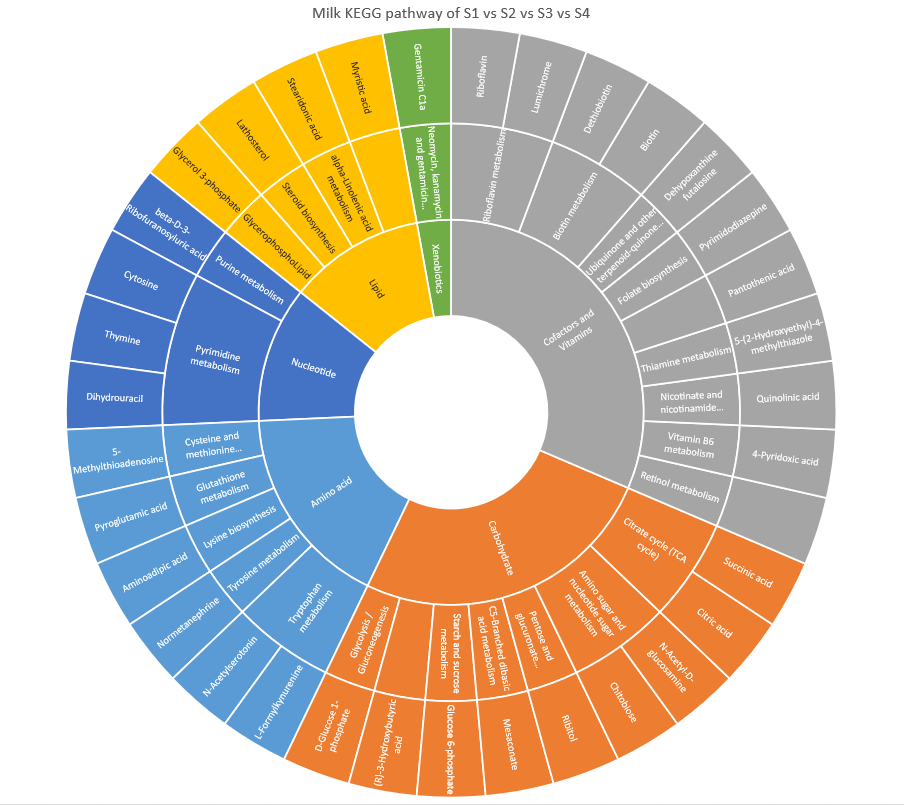
- 注意:多层子目录不要命名,写一个,下面同样的空白即可;最后一列一定要赋值,没有具体的值可以像我一样全部写1,不然无法生成旭日图
ECharts旭日图的绘制
- excel一键绘制旭日图虽然快捷简便,但许多细节无法按需调整,色彩饱和度过高,数据标签的摆放,圈内外径的调整等等都不方便,而下面使用echarts绘制旭日图尽管录入数据麻烦,但是出图美观,调整方便
- 可直接在 ECharts旭日图 (我这里用的drink flavor,更适合我的展示需求)按数据准备中整理后的结果替换模板,实时可视化,便于调整,可出png和svg两种格式结果图
- 可根据 RGB颜色对照表 自行替换想要颜色
- 画布大小问题
- 在线的echarts的画布大小是根据浏览器页面调整的,有时候会出现超出页面且下载后的图片也显示不全的情况(在线的就是显示什么样,下载下来就什么样),这时可调整最后
option参数中r0和r的半径范围来控制整个图片大小 - 如果数据太多仍然显示不全,可使用另一个 菜鸟在线编辑器 ,可设置画布大小(这里每次调整需重新运行,不能实时运行,建议在echarts中调整差不多后再到这边调整出图大小)。菜鸟只能直接出png格式,且默认最外层字体颜色随子扇叶(个人觉得更美观,不喜欢可以
color: #000000)
- 在线的echarts的画布大小是根据浏览器页面调整的,有时候会出现超出页面且下载后的图片也显示不全的情况(在线的就是显示什么样,下载下来就什么样),这时可调整最后
ECharts旭日图
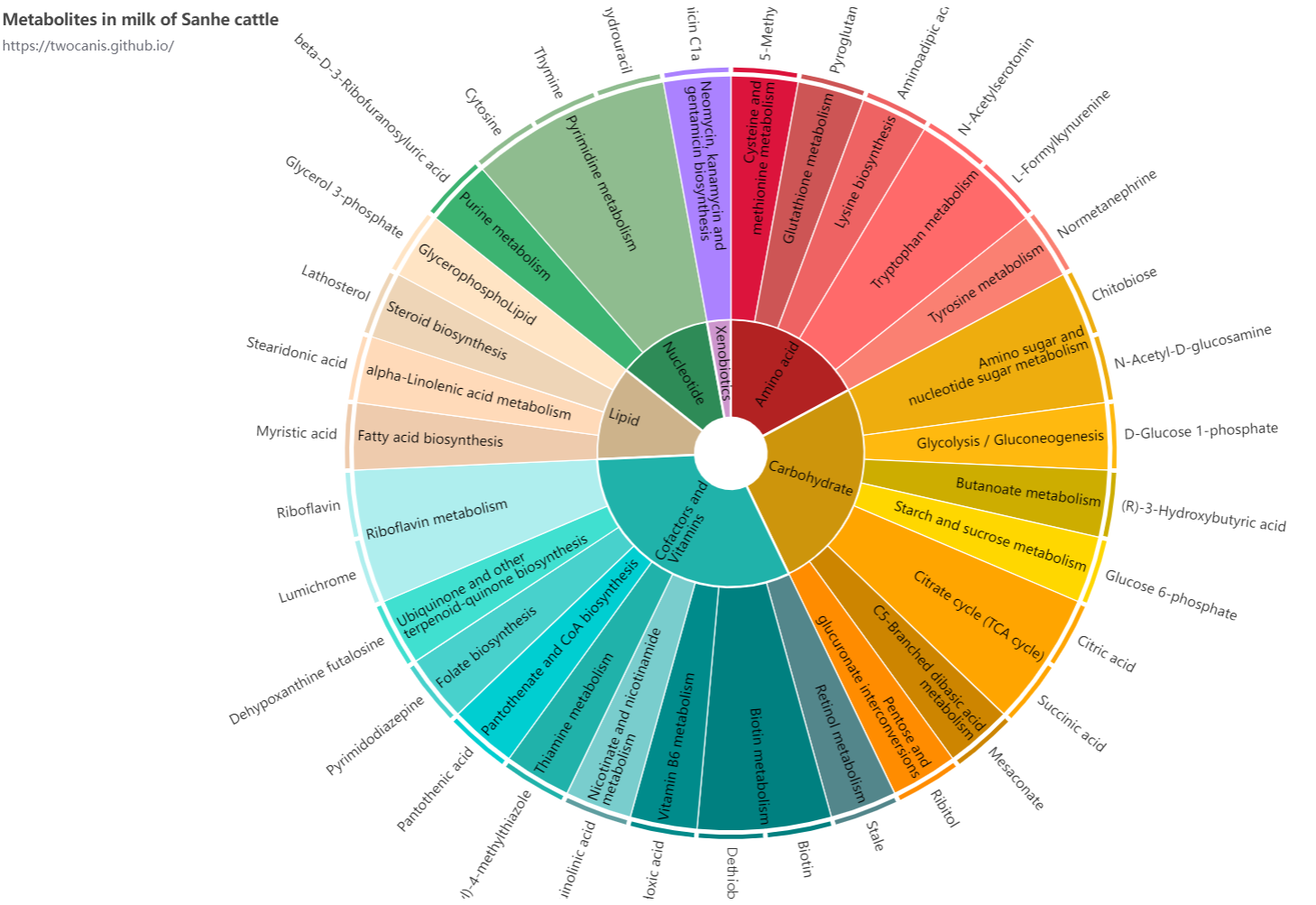
菜鸟编辑器修改后的旭日图
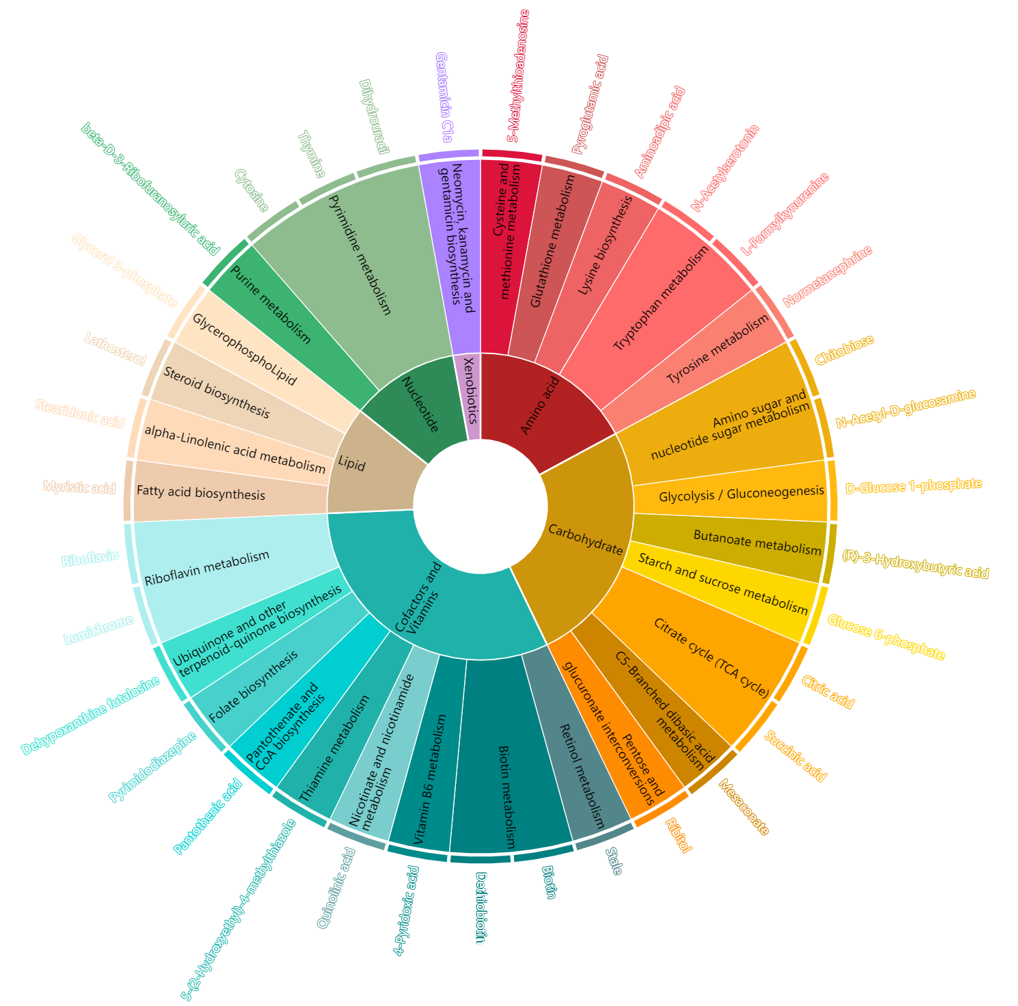
附演示代码
附编辑器内全部演示代码,echarts则只用var data部分即可。
<!DOCTYPE html>
<html>
<head><meta charset="utf-8"><title>ECharts 实例</title><!-- 引入 echarts.js --><script src="https://cdn.staticfile.org/echarts/4.3.0/echarts.min.js"></script>
</head>
<body><!-- 为ECharts准备一个具备大小(宽高)的Dom --> //设置画布大小<div id="main" style="width: 1200px;height:1200px;"></div> <script type="text/javascript">// 基于准备好的dom,初始化echarts实例var myChart = echarts.init(document.getElementById('main'));var data = [{name: 'Amino acid', //最里面第一层圈itemStyle: {color: '#B22222'},children: [ //第二层子圈{name: 'Cysteine and\n methionine metabolism',itemStyle: {color: '#DC143C'},children: [ //第三层子圈{name: '5-Methylthioadenosine',value: 1,itemStyle: {color: '#DC143C'}},]},{name: 'Glutathione metabolism',itemStyle: {color: '#CD5555'},children: [{name: 'Pyroglutamic acid',value: 1,itemStyle: {color: '#CD5555'}},]},{name: 'Lysine biosynthesis',itemStyle: {color: '#EE6363'},children: [{name: 'Aminoadipic acid',value: 1,itemStyle: {color: '#EE6363'}},]},{name: 'Tryptophan metabolism',itemStyle: {color: '#FF6A6A'},children: [{name: 'N-Acetylserotonin',value: 1,itemStyle: {color: '#FF6A6A'}},{name: 'L-Formylkynurenine',value: 1,itemStyle: {color: '#FF6A6A'}},]},{name: 'Tyrosine metabolism',itemStyle: {color: '#FA8072'},children: [{name: 'Normetanephrine',value: 1,itemStyle: {color: '#FA8072'}},]},]},{name: 'Carbohydrate',itemStyle: {color: '#CD950C'},children: [{name: 'Amino sugar and\n nucleotide sugar metabolism',itemStyle: {color: '#EEAD0E'},children: [{name: 'Chitobiose',value: 1,itemStyle: {color: '#EEAD0E'}},{name: 'N-Acetyl-D-glucosamine',value: 1,itemStyle: {color: '#EEAD0E'}},]},{name: 'Glycolysis / Gluconeogenesis',itemStyle: {color: '#FFB90F'},children: [{name: 'D-Glucose 1-phosphate',value: 1,itemStyle: {color: '#FFB90F'}},]},{name: 'Butanoate metabolism',itemStyle: {color: '#CDAD00'},children: [{name: '(R)-3-Hydroxybutyric acid',value: 1,itemStyle: {color: '#CDAD00'}},]},{name: 'Starch and sucrose metabolism',itemStyle: {color: '#FFD700'},children: [{name: 'Glucose 6-phosphate',value: 1,itemStyle: {color: '#FFD700'}},]},{name: 'Citrate cycle (TCA cycle)',itemStyle: {color: '#FFA500'},children: [{name: 'Citric acid',value: 1,itemStyle: {color: '#FFA500'}},{name: 'Succinic acid',value: 1,itemStyle: {color: '#FFA500'}},]},{name: 'C5-Branched dibasic acid\n metabolism',itemStyle: {color: '#CD8500'},children: [{name: 'Mesaconate',value: 1,itemStyle: {color: '#CD8500'}},]},{name: 'Pentose and \nglucuronate interconversions',itemStyle: {color: '#FF8C00'},children: [{name: 'Ribitol',value: 1,itemStyle: {color: '#FF8C00'}},]},]}, {name: 'Cofactors and\n Vitamins',itemStyle: {color: '#20B2AA'},children: [{name: 'Retinol metabolism',itemStyle: {color: '#53868B'},children: [{name: 'Stale',value: 1,itemStyle: {color: '#53868B'}},]},{name: 'Biotin metabolism',itemStyle: {color: '#008080'},children: [{name: 'Biotin',value: 1,itemStyle: {color: '#008080'}},{name: 'Dethiobiotin',value: 1,itemStyle: {color: '#008080'}},]},{name: 'Vitamin B6 metabolism',itemStyle: {color: '#008B8B'},children: [{name: '4-Pyridoxic acid',value: 1,itemStyle: {color: '#008B8B'}},]},{name: 'Nicotinate and nicotinamide \nmetabolism',itemStyle: {color: '#79CDCD'},children: [{name: 'Quinolinic acid',value: 1,itemStyle: {color: '#5F9EA0'}},]}, {name: 'Thiamine metabolism',itemStyle: {color: '#20B2AA'},children: [{name: '5-(2-Hydroxyethyl)-4-methylthiazole',value: 1,itemStyle: {color: '#20B2AA'}},]},{name: 'Pantothenate and \nCoA biosynthesis',itemStyle: {color: '#00CED1'},children: [{name: 'Pantothenic acid',value: 1,itemStyle: {color: '#00CED1'}},]}, {name: 'Folate biosynthesis',itemStyle: {color: '#48D1CC'},children: [{name: 'Pyrimidodiazepine',value: 1,itemStyle: {color: '#48D1CC'}},]}, {name: 'Ubiquinone and other \nterpenoid-quinone biosynthesis',itemStyle: {color: '#40E0D0'},children: [{name: 'Dehypoxanthine futalosine',value: 1,itemStyle: {color: '#40E0D0'}},]},{name: 'Riboflavin metabolism',itemStyle: {color: '#AFEEEE'},children: [{name: 'Lumichrome',value: 1,itemStyle: {color: '#AFEEEE'}},{name: 'Riboflavin',value: 1,itemStyle: {color: '#AFEEEE'}},]},]},{name: 'Lipid',itemStyle: {color: '#CDB38B'},children: [{name: 'Fatty acid biosynthesis',itemStyle: {color: '#EECBAD'},children: [{name: 'Myristic acid',value: 1,itemStyle: {color: '#EECBAD'}},]},{name: 'alpha-Linolenic acid metabolism',itemStyle: {color: '#FFDAB9'},children: [{name: 'Stearidonic acid',value: 1,itemStyle: {color: '#FFDAB9'}},]},{name: 'Steroid biosynthesis',itemStyle: {color: '#EED5B7'},children: [{name: 'Lathosterol',value: 1,itemStyle: {color: '#EED5B7'}},]},{name: 'GlycerophosphoLipid',itemStyle: {color: '#FFE4C4'},children: [{name: 'Glycerol 3-phosphate',value: 1,itemStyle: {color: '#FFE4C4'}},]},]},{name: 'Nucleotide',itemStyle: {color: '#2E8B57'},children: [{name: 'Purine metabolism',itemStyle: {color: '#3CB371'},children: [{name: 'beta-D-3-Ribofuranosyluric acid',value: 1,itemStyle: {color: '#3CB371'}},]},{name: 'Pyrimidine metabolism',itemStyle: {color: '#8FBC8F'},children: [{name: 'Cytosine',value: 1,itemStyle: {color: '#8FBC8F'}},{name: 'Thymine',value: 1,itemStyle: {color: '#8FBC8F'}},{name: 'Dihydrouracil',value: 1,itemStyle: {color: '#8FBC8F'}},]},]},{name: 'Xenobiotics',itemStyle: {color: '#CD96CD'},children: [{name: 'Neomycin, kanamycin and\n gentamicin biosynthesis',itemStyle: {color: '#AB82FF'},children: [{name: 'Gentamicin C1a',value: 1,itemStyle: {color: '#AB82FF'}},]},]},
];
option = {title: {textStyle: {fontSize: 14,align: 'center'},subtextStyle: {align: 'center'},sublink: 'https://twocanis.github.io/'},series: {type: 'sunburst',data: data,radius: [0, '95%'],sort: undefined,emphasis: {focus: 'ancestor'},levels: [{},{r0: '13%', //内径r: '30%', //外径itemStyle: {borderWidth: 2},label: {align: 'right', //调整文字摆放方向fontSize: 14, //调整label字符大小color:'#000000', //使文字显示为黑色}},{r0: '30%', //下一个圈的内径要和上一个全2外经一致才不会出现交错r: '68%',label: {align: 'right',fontSize: 14,color:'#000000'}},{r0: '68%',r: '70%',label: {position: 'outside',padding: 3,fontSize: 14, //这里不额外设置颜色则和母扇叶颜色一致silent: false},itemStyle: {borderWidth: 4}}]}
};// 使用刚指定的配置项和数据显示图表。myChart.setOption(option);</script>
</body>
</html>
这篇关于Echarts绘制差异代谢产物分类与KEGG通路分类的旭日图的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







