本文主要是介绍组蛋白脱乙酰酶介导的胃癌肿瘤微环境特征及协同免疫治疗(多组学文献学习),希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
目录
①HDAC转录组多数据NMF一次聚类
②ACRG队列中HDAC单独NMF聚类
③HDS评分在胃癌中的临床特征和基因组特征
④高 HDS 可能提示胃癌的“热”肿瘤状态
⑤HDS是胃癌免疫治疗效果的有力预测指标
⑥单细胞转录组测序揭示了高HDS和低HDS患者的TME
⑦内皮细胞和成纤维细胞可抑制T细胞和NK细胞通过MIF信号通路浸润
⑧低 HDS 样品中,CCL17+ 浆细胞样树突状细胞可能通过 MIF 信号通路抑制 T 细胞和 NK 细胞浸润
⑨实验验证机制
一、抑制MIF信号转导可增强肿瘤内T细胞浸润和细胞毒性
二、抑制肿瘤细胞中GPX4的表达可能会激活免疫微环境
三、GPX4敲低可抑制胃癌的肿瘤生长和肝转移
四、GPX4 敲低可能促进 CD8+ T 细胞浸润和细胞毒性
Histone deacetylase-mediated tumor microenvironment characteristics and synergistic immunotherapy in gastric cancer (thno.org)
作者研究背景:
据报道,有18种组蛋白脱乙酰酶(HDAC1-11,SIRT1-7)在染色体结构修饰和基因表达调控中起重要作用。组蛋白乙酰化解离 DNA 和组蛋白八聚体,允许转录因子和协同转录因子特异性结合 DNA 结合位点并激活基因转录。相反,组蛋白的脱乙酰化具有相反的效果。HDAC可逆地调节TME发育过程中组蛋白和非组蛋白的乙酰化状态。此外,HDAC6参与免疫系统中几个关键因子的上调,如PD-1和PD-L1受体,它们是主要的癌症免疫治疗靶点。Brune 等人。发现HDAC抑制剂治疗可以增强Foxp3的表达,从而诱导和维持免疫抑制Treg表型。Kai 等人。研究发现,HDAC3抑制剂可以增强CD8+ T细胞向细胞毒性效应细胞的分化。上述研究表明,HDAC可能是调控TME的重要靶点。然而,不同类型的HDAC在调节TME细胞浸润水平方面存在异质性,单靠单个分子或单一一类分子靶点抑制剂无法精确控制TME的变化。因此,迫切需要系统地分析HDAC表达谱和相应的TME特征,为胃癌的临床治疗策略和预后评估提供理论基础。(也就是从转录组层面研究18种组蛋白脱乙酰酶的共表达模式与胃癌TME之间的关联)
个人总结亮点:①利用转录组数据识别肿瘤亚型;②构建的评分与免疫细胞浸润进行相关性分析识别肿瘤免疫冷热关系;③TIDE与submap还有免疫队列结合分析模型预测免疫性能;肿瘤预测TIDE的使用_tide算法-CSDN博客submap 使用说明-CSDN博客④单细胞及配对转录组数据结合分析不同评分之间的TME差异;⑤实验验证层面不仅验证了MIF信号通路与肿瘤免疫浸润,还根据低HDS评分组的药物分析确定了低HDS敏感的药物,从而挖掘了GPX4 抑制剂。⑥关于实验细胞系的确定,CCLE数据库分析细胞的HDS值,发现AGS细胞的HDS最低,使验证更加充分。基因在各个细胞系表达情况-CSDN博客
①胃癌单细胞和配对转录组揭示胃肿瘤微环境(文献和数据)-CSDN博客
①HDAC转录组多数据NMF一次聚类
首先分析了基因表达与通路和细胞富集之间的相关性,ssGSEA用于通过“GSVA”包评估签名的分数。18 个 HDAC 包括 4 个 I 类 HDAC(HDAC1、HDAC2、HDAC3 和 HDAC8)、6 个 II 类 HDAC(HDAC4、HDAC5、HDAC6、HDAC7、HDAC9 和 HDAC10)、7 个 III 类 HDAC(SIRT1、SIRT2、SIRT3、SIRT4、SIRT5、SIRT6 和 SIRT7)和 1 个 IV 类 HDAC(HDAC11)。六个队列(TCGA-STAD、GSE15459、GSE34942、GSE57303、ACRG 和 GSE84437)进行 combat 批次校正然后NMF识别肿瘤亚型,有3个HDAC肿瘤亚型。
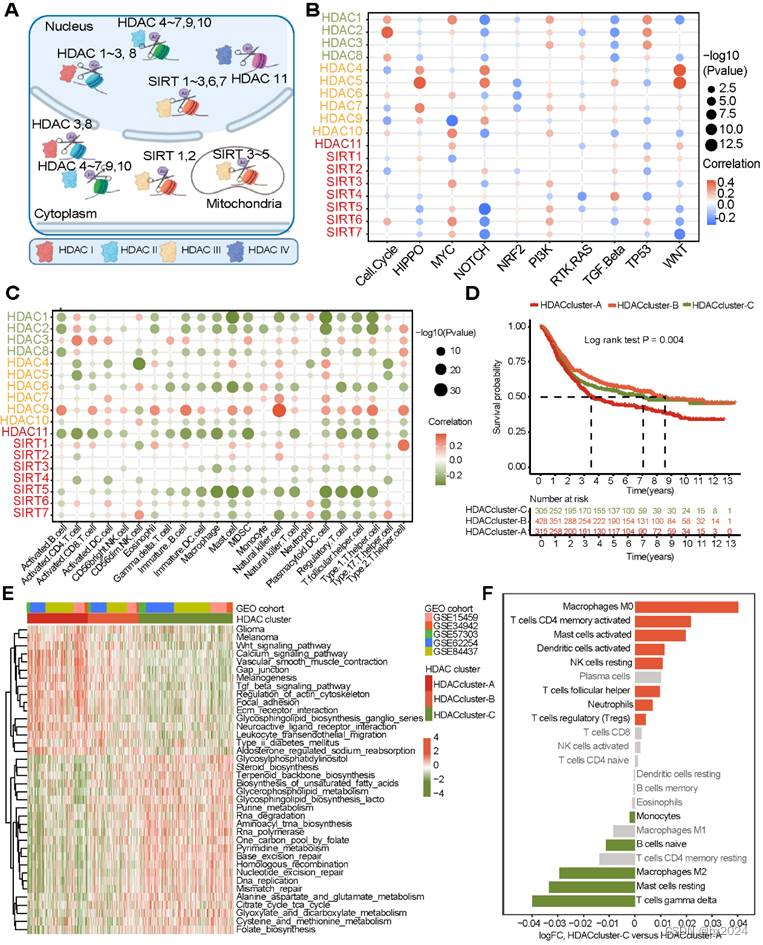
(A)细胞中HDACs的机制。(B) HDAC表达与十种癌症相关途径的关联。(C) HDAC mRNA表达水平与免疫细胞浸润之间的相关性。(D) 通过HDACs的mRNA表达确定的三种具有显着预后差异的胃癌亚型。(E)KEGG功能富集分析,了解不同HDAC簇的特征。(F) 不同HDAC簇之间TME细胞浸润水平的差异(CIBERSORT算法)。

②ACRG队列中HDAC单独NMF聚类
单独对ACRG进行聚类分析,然后进行构建评分模型。limma包用于鉴定ACRG队列中不同HDAC簇之间差异表达的基因。FDR值 0.01 被认为表明表达有显著差异。单因素Cox回归分析确定了具有显著预后价值的DEGs。仅保留P值<0.05的基因。最后,采用Boruta算法对具有显著预后价值的DEGs进行降维。HR<为1的基因被定义为基因集A,HR>为1的基因被定义为基因集B。构建评分:

(A) HDAC mRNA在三个HDAC簇中的差异表达。主成分分析揭示了3个HDAC簇的分布情况。不同 HDAC 之间的预后差异。(B)三个HDAC簇中的差异表达基因。热图显示了 103 个基因(A 基因和 B 基因)在 Boruta 算法降维后的表达谱。(C)HDAC特征A基因和B基因的生物学过程。(D)桑基图,显示3个HDAC簇和3个基因簇之间的关系。(E) 不同基因簇之间的预后差异。(F) 通过主成分分析和评估不同HDAC簇和基因簇之间HDS的差异来构建HDS模型。*P < 0.05, **P < 0.01, ***P < 0.001, ****P < 0.0001.
③HDS评分在胃癌中的临床特征和基因组特征

(A) 高 HDS 组和低 HDS 组(TCGA 队列)之间的预后差异。胃癌亚型 HDS 的差异(TCGA 队列)。(B) 高 HDS 组和低 HDS 组(ACRG 队列)之间的预后差异。(C) 胃癌亚型和临床特征(ACRG 队列)中 HDS 的差异。(D-E)采用单因素和多因素COX回归分析HDS和临床特征对总生存期(OS)和无复发生存期的影响。(F) HDS与抗原呈递分子、共刺激分子和共抑制分子之间的相关性。*P < 0.05, **P < 0.01, ***P < 0.001, ****P < 0.0001.
④高 HDS 可能提示胃癌的“热”肿瘤状态
为了研究 HDS 和 TME 之间的关系,我们分析了 TCGA 和 GEO 数据库中的六个胃癌队列。结果显示,CD4+ T细胞、CD8+ T细胞、NKT细胞和Th17细胞等免疫细胞的富集评分与HDS呈显著正相关(图4A)。然而,M2巨噬细胞、MDSC、pDCs和基质细胞(成纤维细胞和内皮细胞)等免疫抑制细胞的评分与HDS呈显著负相关。有趣的是,HDS和Treg细胞之间存在显着的正相关关系(图4A)。此外,我们发现HDS与五个胃癌队列中血管生成分子的表达呈显著负相关(图4B)。上述研究表明,高HDS可能表明“热”肿瘤,但与此同时,这些肿瘤表现出免疫抑制。
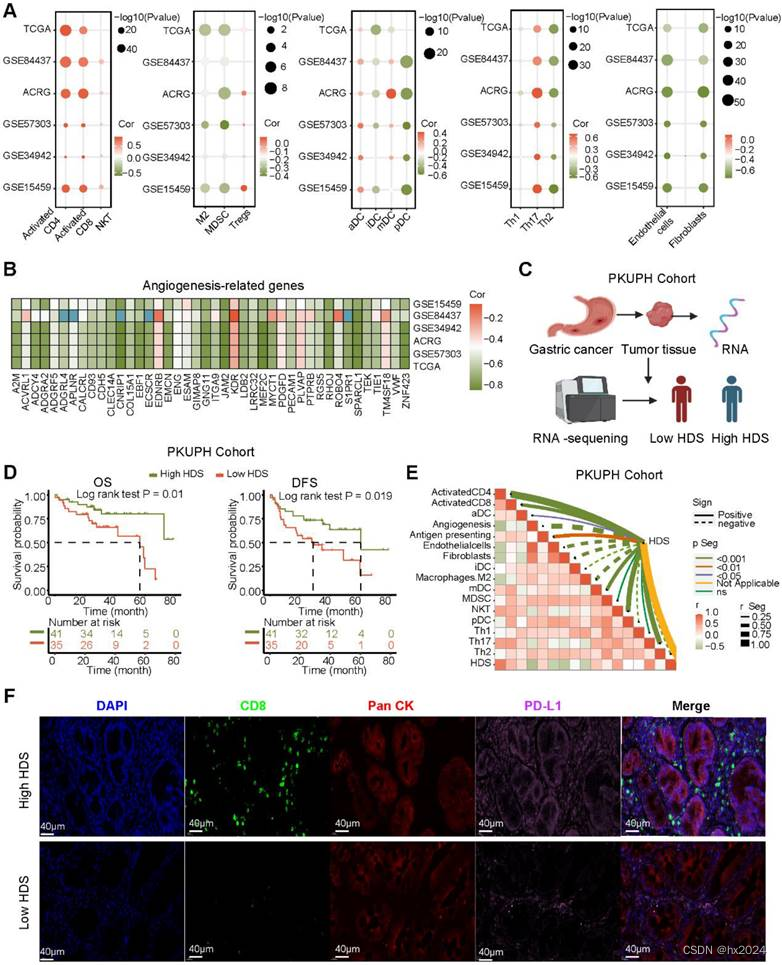
(A) 在五个胃癌队列中分析了 HDS 和 TME 细胞富集评分之间的相关性。(B) 在5个胃癌队列中,HDS与血管生成分子的表达呈显著负相关。(C) RNA-seq的流程图。(D)生存分析显示,HDS高的患者OS和DFS更长。(E) 在 PKUPH 队列中分析 HDS 和 TME 细胞富集评分之间的相关性。(F) CD8 和 PD-L1 蛋白在高 HDS 和低 HDS 样品中的表达水平。
⑤HDS是胃癌免疫治疗效果的有力预测指标

(A)TIDE算法和submap算法预测高HDS和低HDS肿瘤对PD-1抑制剂的反应。(B) 不同反应组(CR/PR 和 SD/PD)的 HDS 差异(PRJEB40416队列)。(C)ROC曲线揭示了HDS、CPS和HDS+CPS预测免疫治疗疗效的准确性。(D-E)不同反应组(CR/PR 和 SD/PD)的 HDS 差异(PRJEB25780队列)。(F)ROC曲线的AUC值揭示了不同指标预测免疫治疗疗效的准确性。(G) 列线图显示 HDS 结合 MSI 状态和 CPS,以预测胃癌免疫治疗的疗效。
⑥单细胞转录组测序揭示了高HDS和低HDS患者的TME
用单细胞和配对的转录组数据进行分析。
①胃癌单细胞和配对转录组揭示胃肿瘤微环境(文献和数据)-CSDN博客

(A) 分析了 24 个单细胞测序数据集和 20 个配对批量测序数据集。(B)根据标记基因的定义,通过T分布随机邻域嵌入(t-SNE)共鉴定出9个簇。(C) tSNE 图显示了高低 HDS 患者 TME 细胞的特征。 (D) 堆叠图揭示了每个细胞簇中高和低 HDS 的比例。(E) 多次免疫荧光染色验证了高 HDS 和低 HDS 样品中 CD4+ T 细胞和 CD8+ T 细胞的差异。
⑦内皮细胞和成纤维细胞可抑制T细胞和NK细胞通过MIF信号通路浸润
在低HDS组中,内皮细胞、成纤维细胞和髓样细胞中向CD4+ T细胞、CD8+ T细胞和NK细胞的信号通路的数量和强度显著高于高HDS组。

(A)基于CellChat分析了高HDS和低HDS样品中细胞相互作用数量和强度的差异。(B) 圆图和热图显示了高 HDS 和低 HDS 下每个细胞簇相互作用的数量和强度的差异。 (C) tSNE 分析揭示了基质细胞的重聚图。气泡图显示了每个细胞簇的标记基因。(D) 堆叠图显示了每个簇中高HDS和低HDS样品的比例。(E) CellChat用于分析内皮细胞、成纤维细胞、T细胞和NK细胞之间的通讯。(F)气泡图和圆图显示,在低HDS样品中,SFRP2+成纤维细胞、MYH11+成纤维细胞、CD234+内皮细胞和CD69+成纤维细胞可能通过MIF信号频繁与T细胞和NK细胞交流。
⑧低 HDS 样品中,CCL17+ 浆细胞样树突状细胞可能通过 MIF 信号通路抑制 T 细胞和 NK 细胞浸润

(A) tSNE分析揭示了髓样细胞的重新聚类图谱。(B)气泡图显示每个细胞簇中定义的基因的表达。(C)气泡图显示每个细胞簇的标记基因。(D)基于CellChat分析了高HDS和低HDS样品中细胞相互作用数量和强度的差异。(E) 气泡图显示低 HDS 肿瘤中上调的信号通路。(F) 小提琴图显示了 CCL17+ pDC、T 细胞和 NK 细胞中 MIF 信号通路相关配体和受体的表达水平。(G) KEGG展示了CCL17+ pDC的功能。(H) CCL17+ pDCs富集评分与HDS(ACRG队列)之间的相关性。(I) CCL17+ pDCs富集评分对胃癌预后的影响(ACRG队列)。
⑨实验验证机制
一、抑制MIF信号转导可增强肿瘤内T细胞浸润和细胞毒性

A) MIF抑制剂治疗胃癌的方案。(B)MIF抑制剂显著抑制胃癌的生长。(C)免疫荧光显示MIF抑制组和对照组T细胞浸润水平。(D-E)流式细胞术分析MIF抑制剂组和对照组CD8+ T细胞的比例和细胞毒性。(F-G)采用流式细胞术分析MIF抑制剂组和对照组CD4+ T细胞的比例和细胞毒性。*P < 0.05,**P < 0.01。
二、抑制肿瘤细胞中GPX4的表达可能会激活免疫微环境
关于这一步的联系:作者使用CTPR和PRISM数据库被用来识别可能对高HDS和低HDS肿瘤有效的化疗药物(图10A)。发现 HDS 低的胃癌肿瘤可能对 GPX4 抑制剂(ML210、ML162 和 1S3R-RSL-3)、存活素抑制剂 (YM-155) 和达沙替尼敏感。关于低HDS 细胞系的选择:利用CCLE数据库分析细胞的HDS值,发现AGS细胞的HDS最低,然后进行分析。
(A) CTRP和PRISM数据库预测了低HDS样品的有效化合物。(B) 五种胃癌细胞系的 HDS 值。GPX4 mRNA在不同干预组中的表达。(C) HDACs在高HDS组和低HDS组中的差异表达。(D) 敲低GPX4后HDACs的表达。(E) NC组和GPX4敲低组差异表达基因的GO和KEGG功能富集分析。(F) GPX4敲低后共刺激分子和共抑制分子表达的差异分析。(G) 高HDS组和低HDS组免疫细胞浸润和免疫相关特征的差异分析。*P < 0.05, **P < 0.01, ***P < 0.001, ****P < 0.0001.
三、GPX4敲低可抑制胃癌的肿瘤生长和肝转移
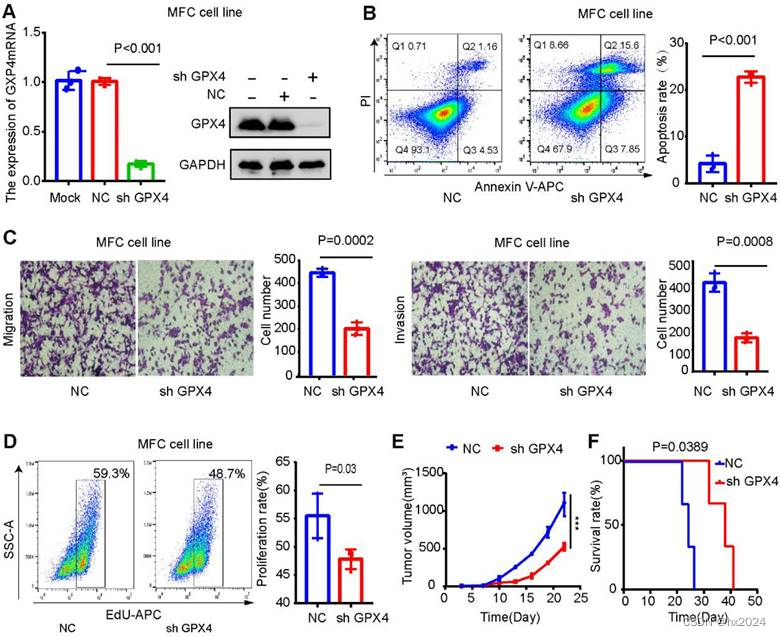
(A) Western blot和qPCR检测验证了GPX4在MFC细胞中的敲低效率。(B)通过细胞凋亡法检测GPX4敲低对细胞凋亡的影响。(C) 通过细胞迁移和侵袭试验检测 GPX4 敲低对细胞迁移和侵袭的影响。(D) EdU 测定法检测 GPX4 敲低对细胞增殖的影响。(英-法)小鼠皮下异种移植模型显示,敲低GPX4可显著抑制肿瘤细胞生长,延长小鼠存活时间。**P < 0.01。
四、GPX4 敲低可能促进 CD8+ T 细胞浸润和细胞毒性
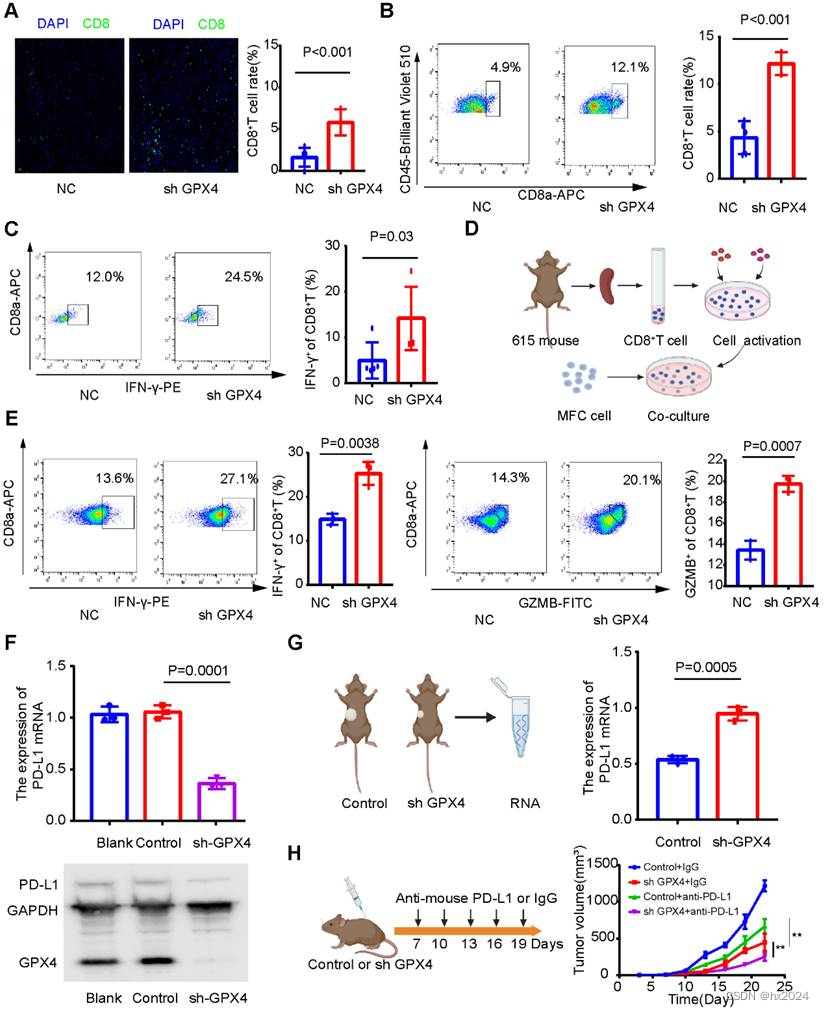
(A)免疫荧光试验显示,GPX4敲低显著促进CD8+T细胞浸润水平。(B)采用流式细胞术分析GPX4敲除后肿瘤组织中CD8+ T细胞的浸润水平。(C)流式细胞术显示,GPX4敲低显著促进CD8+T细胞分泌IFN-γ,增加CD8+T细胞毒性。(D) CD8+ T 细胞的活化并与肿瘤细胞共培养。(E) 通过流式细胞术测定共培养后 CD8+ T 细胞的细胞毒性。(F) GPX4敲低显著抑制PD-L1 mRNA和蛋白的表达。(G)PD-L1 mRNA在小鼠皮下肿瘤中的表达水平。(H) GPX4敲低联合PD-L1抑制剂显著提高免疫治疗疗效,抑制肿瘤生长。
文献:Histone deacetylase-mediated tumor microenvironment characteristics and synergistic immunotherapy in gastric cancer
这篇关于组蛋白脱乙酰酶介导的胃癌肿瘤微环境特征及协同免疫治疗(多组学文献学习)的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







