本文主要是介绍使用Yolov7进行LUNA16的肺结节检测,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
1、图像预处理
1.1 从mhd文件中获取CT图像,保存3张切片
def normalizePlanes(npzarray):maxHU = 400minHU = -1000npzarray = (npzarray - minHU)/(maxHU - minHU)npzarray[npzarray>1] = 1npzarray[npzarray<0] = 0npzarray *= 255return (npzarray.astype(int))def make_mask(center,diam,z,width,height,spacing,origin):''''''mask = np.zeros([height,width]) #匹配图像#定义结节所在的体素范围v_center = (center-origin)/spacingv_diam = int(diam/spacing[0]+5)v_xmin = np.max([0,int(v_center[0]-v_diam)-5])v_xmax = np.min([width-1,int(v_center[0]+v_diam)+5])v_ymin = np.max([0,int(v_center[1]-v_diam)-5])v_ymax = np.min([height-1,int(v_center[1]+v_diam)+5])v_xrange = range(v_xmin,v_xmax+1)v_yrange = range(v_ymin,v_ymax+1)x_data = [x*spacing[0]+origin[0] for x in range(width)]y_data = [x*spacing[1]+origin[1] for x in range(height)]#结节周围全都填充1for v_x in v_xrange:for v_y in v_yrange:p_x = spacing[0]*v_x + origin[0]p_y = spacing[1]*v_y + origin[1]if np.linalg.norm(center-np.array([p_x,p_y,z]))<=diam:mask[int((p_y-origin[1])/spacing[1]),int((p_x-origin[0])/spacing[0])] = 1.0return (mask)luna_path = "E:/LUNA16/src/subset0/"
output_path = "E:/VSCode/lung/npy"
file_list = glob("E:\LUNA16\src\subset0/" + "*.mhd")# 获取数据中的每一行
def get_filename(case):global file_listfor f in file_list:if case in f:return (f)# 节点的位置
df_node = pd.read_csv('E:\LUNA16\src/annotations.csv')
df_node["file"] = df_node["seriesuid"].apply(get_filename)
# df_node: 文件的路径 example:E:\LUNA16\src\subset0\1.3.6.1.4.1.14519.5.2.1……
df_node = df_node.dropna()# 循环遍历图像文件
#fcount = 0
for fcount, img_file in enumerate(tqdm(file_list)):print("Getting mask for image file %s" % img_file.replace(luna_path, ""))mini_df = df_node[df_node["file"] == img_file] # 得到所有结节if(len(mini_df) > 0): #跳过没有结节的文件#读取数据itk_img = sitk.ReadImage(img_file)img_array = sitk.GetArrayFromImage(itk_img) #(z,y,x)num_z,height,width = img_array.shape #heightXwidth constitute the transverse plane origin = np.array(itk_img.GetOrigin()) #世界坐标系下的x,y,z(mm)spacing = np.array(itk_img.GetSpacing()) #世界坐标中的体素间隔(mm)#遍历所有节点for node_idx,cur_row in mini_df.iterrows():node_x = cur_row["coordX"]node_y = cur_row["coordY"]node_z = cur_row["coordZ"]diam = cur_row["diameter_mm"]#保留三个切片imgs = np.ndarray([3,height,width],dtype=np.float32)masks = np.ndarray([3,height,width],dtype=np.uint8)center = np.array([node_x,node_y,node_z]) #结点中心v_center = np.rint((center-origin)/spacing) #体素坐标系的结点中心(x,y,z)for i,i_z in enumerate(np.arange(int(v_center[2])-1,int(v_center[2])+2).clip(0,num_z-1)): #clip防止超出zmask = make_mask(center,diam,i_z*spacing[2]+origin[2],width,height,spacing,origin)masks[i] = mask#imgs[i] = img_array[i_z]imgs[i] = normalizePlanes(img_array[i_z])np.save(os.path.join(output_path,"images_%04d_%04d.npy" % (fcount, node_idx)),imgs)np.save(os.path.join(output_path,"masks_%04d_%04d.npy" % (fcount, node_idx)),masks)1.2 获取肺实质
for img_file in file_list:imgs_to_process = np.load(img_file).astype(np.float64) print("on image",img_file)for i in range(len(imgs_to_process)):img = imgs_to_process[i]#标准化mean = np.mean(img) #均值std = np.std(img) #标准差img = (img-mean)/std#寻找肺部附近的平均像素,以重新调整过度曝光的图像middle = img[100:400,100:400]#使用Kmeans算法将前景(放射性不透明组织)和背景(放射性透明组织,即肺部)分离。#仅在图像中心进行此操作,以尽可能避免图像的非组织部分。kmeans = KMeans(n_clusters=2,n_init=10).fit(np.reshape(middle,[np.prod(middle.shape),1]))#np.prod 计算给定数组中所有元素的乘积#np.reshape 低维变高维 .flatten() 高维变1维centers = sorted(kmeans.cluster_centers_.flatten())threshold = np.mean(centers)thresh_img = np.where(img<threshold,1.0,0.0) #阈值化图像,二值化处理#腐蚀和膨胀eroded = morphology.erosion(thresh_img,np.ones([4,4]))dilation = morphology.dilation(eroded,np.ones([10,10]))labels = measure.label(dilation)#对二值图像进行标记,标记连通区域label_vals = np.unique(labels) #获取标记值的唯一值,即标记的数量regions = measure.regionprops(labels) #标记区域good_labels = []for prop in regions:B = prop.bbox #边界框if B[2]-B[0]<475 and B[3]-B[1]<475 and B[0]>40 and B[2]<472:good_labels.append(prop.label)#np.ndarray 创建多维数组mask = np.ndarray([512,512],dtype=np.int8)mask[:] = 0 #肺部maskfor N in good_labels:mask = mask + np.where(labels==N,1,0)mask = morphology.dilation(mask,np.ones([10,10]))imgs_to_process[i] = masknp.save(img_file.replace("images","lungmask"),imgs_to_process)1.3 获取xml和jpg文件
通过遍历只含结节图像nodule_annos.xls,该文件在另一篇文章中生成。点此跳转
这里也可以通过遍历官方文件annotations.csv获得
获取结节图像的结节信息生成xml文件,生成xml文件的方法参考了这位博主的方法,可以去学习,生成不了也可以去下载该博主共享的数据集,点此跳转
获取名称匹配lungmask图像获得jpg图像
count = 0
for i in range(len(img_list)):origin_img = np.load(img_list[i])lung_mask = np.load(lungmask_list[i])for j in range(len(origin_img)):img = origin_img[j]lung_img = lung_mask[j]segment = img * lung_imgsegment = Image.fromarray(segment)segment = segment.convert('RGB')segment.save("E:\VSCode\lung/" + '{:04d}.jpg'.format(count+1))count = count + 1
2、模型
YOLOV7
3、训练
2023.4.14更
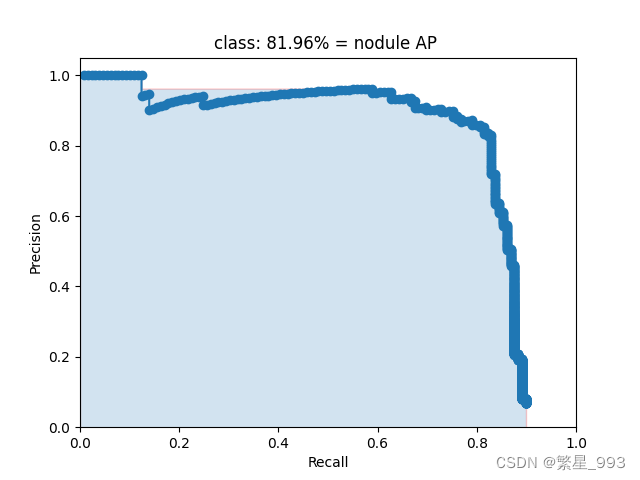
![]()
当然后续继续训练也是有很大的提升的,模型没有完全收敛
2023.5.15 更
有小伙伴私信问我说xml中的filename和图片名称对不上,具体的原因是因为我删掉了一部分肺实质分割不太好的分割图,所以导致最后出现不匹配的原因。
不过labelimg查看结节位置都是对的,训练起来也没有太大的影响
这里用无删减的数据集训练了一下
训练结果
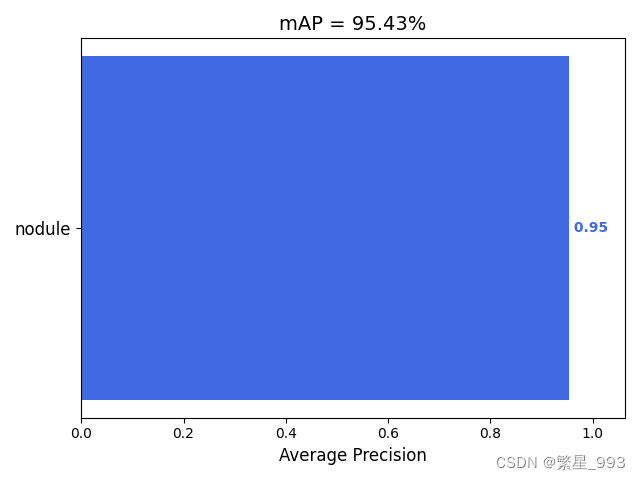
![]()
检测结果


这篇关于使用Yolov7进行LUNA16的肺结节检测的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!




