本文主要是介绍肺结节3D图像分割-VNet(一),希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
近期用Yolo训练肺结节检测模型感觉缺少3D结构信息,尝试一下3D图像分割,博文用以记录。
1、LUNA16数据集
1.1 Luna16数据集介绍
简介:来自于公开的LIDC/IDRI数据集。该数据集剔除了LIDC/IDRI数据集中切片厚度大于2.5mm的扫描数据,共产生了888套CT。Luna16数据集中结节的判定标准为四名放射科专家中至少有三名认定该结节半径大于3mm。因此在数据集的注释中,非结节、半径小于3mm的结节和被1名或两名放射科专家认为是半径大于3mm的结节被认定为无关的发现。
1.2 所用数据
Luna16数据集:
点此跳转
subset0-subset10:10个zip文件中包含的所有的CT图像。
annotations.csv:包含1186个结节的注释。
LIDC/IDRI数据集:
点此跳转
LIDC-XML-only.zip:放射科医生注释/分割(XML 格式)
1.3 mhd文件和raw文件
subset0-10中每一个病例的CT扫描都给出了.mhd和.raw
.mhd文件包含了CT图像的基本信息,.raw文件用来储存CT的图像
1.4 annotations.csv文件
文件包含了各个CT中结节的世界坐标和半径
2、图像预处理
2.1 读取结节图像信息
从subset0-9中找到.mhd文件,读取图像相关信息
def read_data(mhd_file): #读取图像数据,img, origin, spacing, flagwith open(mhd_file) as f:mhd_data = f.readlines()for i in mhd_data: if i.startswith('TransformMatrix'):tmp = i.split('=')[1]if tmp == '1 0 0 0 1 0 0 0 1\n': #判断是否翻转flag = Trueelse:flag = Falseitkimage = sitk.ReadImage(mhd_file)numpyImage = sitk.GetArrayFromImage(itkimage) #从mhd读取raw,图像print("读取数据,读取的图片大小(zyx):",numpyImage.shape)origin = itkimage.GetOrigin()print("读取数据,读取的坐标原点(xyz):",origin) #x,y,zspacing = itkimage.GetSpacing()print("读取数据,读取的像素间隔(xyz):",spacing) #x,y,zreturn numpyImage, origin, spacing, flag2.2 坐标变换
从annotations.csv文件读取到的坐标是世界坐标系下的坐标,我们需要将其转换为图像坐标系下的坐标,像素间隔为[1,1,1].
需要注意的是一张CT图像可能包含不止一个结节,我们需要将其全部遍历完才能遍历下一张图
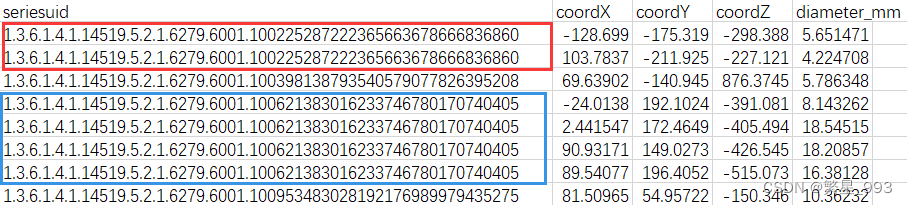
annos_all_list = [] #读取到的原始坐标for i in range(len(annos)):if annos[i][0] == img_name: annos_all_list.append(list(annos[i])) print(annos_all_list)读取到图像包含的所有结节后,将其进行坐标转换
for annos_one_list in annos_all_list: #其中一个结点print("annos_one_list:",annos_one_list) #世界坐标w_center = [annos_one_list[1], annos_one_list[2], annos_one_list[3]] #世界坐标:[x,y,z]print("世界坐标:",w_center)#世界坐标 --> 图像坐标#(世界坐标中心 - 原点) / 像素间隔v_center = list(abs(w_center - np.array(origin)))#/np.array(spacing) 此处像素间隔为[1,1,1] 图像坐标:[x,y,z]if flag is False: #图像翻转,拍CT时为俯拍,需转换为仰拍. #img.shape(z,y,x) v_center(x,y,z)v_center = [(img.shape[2]-1 - v_center[0]), (img.shape[1]-1 - v_center[1]), v_center[2]] #z轴不需要翻转diam = annos_one_list[4] #直径print("结节直径:",diam)将转换后的结节坐标保存下来
one_annos = []
one_annos.append(v_center[0]) #图像坐标x
one_annos.append(v_center[1]) #图像坐标y
one_annos.append(v_center[2]) #图像坐标z
return_list.append(one_annos)
print("one_annos:",one_annos,"[坐标(x,y,z)]")2.3 结节信息保存
所有结节全部转换完后,将保存的数据保存输出至nodule_annos.xls文件
nodule_annos = pd.DataFrame(nodule_list) # 把nodule_list转换成excel文件
nodule_annos.to_excel(nodule_annos_path) #保存至指定路径查看输出文件
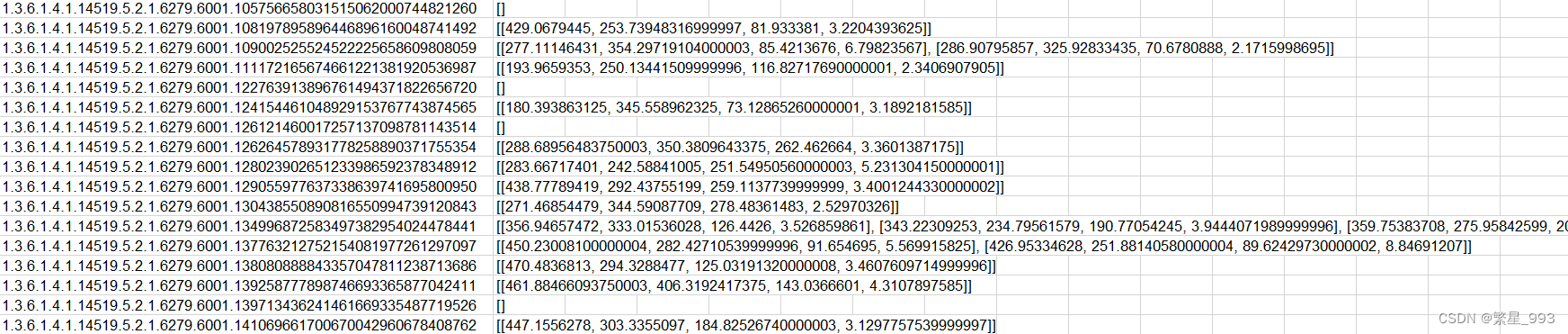
2.4 滤除不含结节图像
def annos_clean():annos = pd.read_excel(nodule_annos_path)annos = annos.tolist()annos_c = []for i in annos:if i[2] != "[]":annos_c.append(i[1])return annos_c这篇关于肺结节3D图像分割-VNet(一)的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







