本文主要是介绍论文导读:用DNA作为计算和数据存储的通用化学基质,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
近日,国际著名学术期刊《自然-化学综述》(Nature Reviews Chemistry)在线刊登了上海交通大学材料科学与工程学院Stephen Mann教授团队与合作团队的综述文章“DNA as a universal chemical substrate for computing and data storage”,为DNA计算及DNA数据存储等领域提供了前瞻方向。该论文以上海交通大学为第一单位,上海交通大学材料科学与工程学院Stephen Mann院士、化学化工学院樊春海院士及荷兰埃因霍温理工大学Tom F. A. de Greef教授为通讯作者,杨朔助理研究员为共同第一作者。

主要内容:
生物计算有望解决传统硅基计算在分子诊断、数据存储和信息安全等领域的应用局限。由于高度的可编辑性和可预测性,DNA是首选的计算及信息存储的基质。在化学综述中,团队重点探讨了DNA计算,尤其是在神经网络和区室化DNA电路等方向的研究最近进展。作者还讨论了DNA数据存储中包括DNA编码数据的写入、读取、检索和合成后编辑的新进展。最后,团队提出DNA计算与DNA数据存储的结合为未来的重要方向,将对信息技术和分析诊断的发展带来重大意义。
摘自:
https://cloud.tencent.com/developer/article/2393531
https://mp.weixin.qq.com/s/jC6YBYb5UmS_uOmCw2P4Ig
引言:
DNA计算和DNA数据存储是新兴领域,它们为信息技术和诊断学解锁了新的可能性。这些方法利用DNA分子作为计算基质或存储介质,提供了纳米级的紧凑性,并能在非传统介质(包括水溶液、水包油微乳液和自组装的膜化隔室)中操作,这些都是超越传统基于硅的计算系统的应用。为了构建一个能够处理和存储分子信息的功能性DNA计算机,需要持续发展计算和数据存储的策略,并且桥接这两个领域之间的差距。
活体系统由物理限制和化学控制的自主代理体组成,展现出计算和学习能力,这些能力到目前为止用传统的基于硅的设备难以模仿。此外,将基于硅的设备直接与活细胞中的过程接口也面临挑战。分子计算是一种吸引人的替代方案,它使用化学物种进行计算,在生物学和化学环境中运作,这些环境与用于分子诊断、数据存储和信息安全的传统电子设备不兼容。在生物学中,信息被编码在DNA中,DNA是一种由四种核苷酸组成的生物聚合物——腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)和鸟嘌呤(G),通过脱氧核糖和磷酸骨架连接。核苷酸通常通过A-T和C-G对之间的Watson–Crick碱基配对进行相互作用(图1)。序列对在溶液中的双链形成可以通过溶液的温度、pH值和离子强度来控制,并可以使用最近邻热力学模型准确预测。这种可编程和可预测的碱基配对性质对于构建由DNA制成的大规模化学反应网络至关重要。DNA作为计算的替代基质在1994年浮现,当时Adleman展示了旅行商问题可以通过DNA反应网络的大规模并行操作来解决。两年后,Davis展示了一个视觉图标可以转化为DNA序列并存储在细菌中,表明了使用DNA作为数据存储介质的可能性。与其他生物分子(如蛋白质和糖)或合成聚合物相比,DNA由于其可预测和特异性的碱基配对以及支持未来发展的化学和生物工业的成熟度而被优先作为计算基质。虽然构建基于DNA的计算机有几种方法,但自2000年Yurke等人提出的DNA链置换(DSD)已成为DNA计算最常见的方法之一。酶反应的实施扩展了工程DNA计算系统的工具箱。此外,DNA自组装的纳米结构,已被用于设计计算任务的算法,并且从无机物质到生物分子的纳米材料也已被利用以促进DNA计算。与DNA计算相比,DNA数据存储的发展较慢。自20世纪60年代以来,DNA的天然信息携带功能使研究人员推测它如何可以被用作数据存储介质,但直到2012年,实用且可扩展的DNA数据存储才成为现实。信息已被编码在DNA的结构中,但更广泛使用的方法是在核苷酸序列中编码信息,很像自然界的做法。

图 1
尽管DNA计算和DNA数据存储处于不同的发展阶段,但DNA纳米技术领域已促进了这些领域的整合,研究人员目标是开发一种基于DNA的计算架构,能够感知、处理和存储大量的分子输入。这篇综述提供了基于DNA的计算和数据存储在信息技术和体外诊断中的概览。
DNA计算的进展
基于硅的计算使用电子逻辑门执行多样化任务,这些逻辑门接收并处理电子输入以产生输出信号。类似的逻辑门已经能通过使用核酸作为信号处理基质。DNA基础的神经网络已成为DNA计算中的重要架构,借鉴了受大脑启发的人工神经网络的成功,展示了基于DNA系统的模式识别和决策制定能力。为了实现这些多样化的功能,研究人员建立了各种分子工具箱,范围从仅DNA系统和酶辅助DNA反应到DNA纳米结构和隔室化DNA电路。
无酶电路
在电路层面,对可预测和可编程行为的需求导致了发展“托基介导的DNA链置换(DSD)”作为构建连接电路层、神经网络或水介质中隔室化电路的常见构件(图1b)。托基介导的DSD是一个过程,其中一个单链DNA(ssDNA)分子(称为输入)替换掉一个部分互补的预杂交双链复合体(称为门)的现有链。该反应在门复合体的单链托基域启动,并通过分支迁移进行,接着是一系列可逆的单核苷酸结合和解离步骤,最终导致现有链作为输出。通过改变托基的长度和序列,DSD的反应动力学可以在大约六个数量级上量化调节。结合这些简单有效的原语使DSD能够执行与电子电路相同的基本操作,如布尔逻辑、算术操作和神经网络功能。
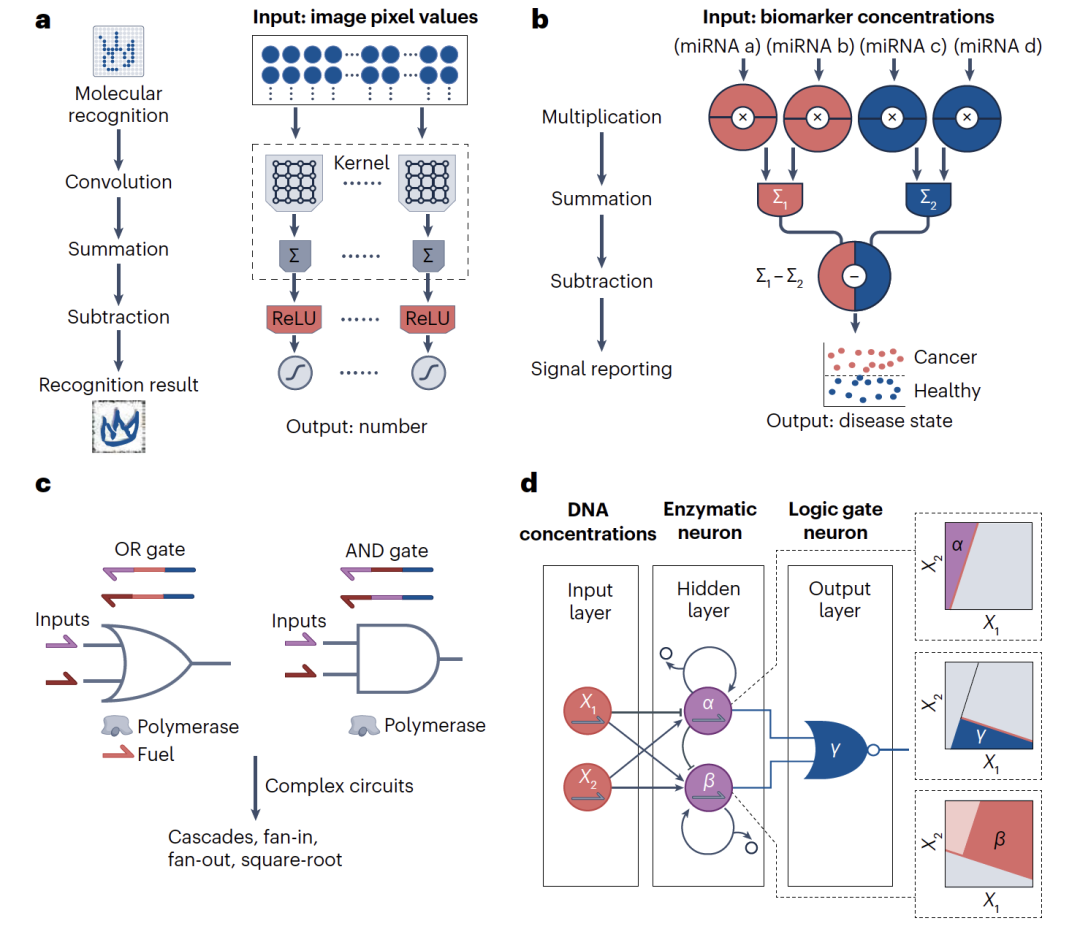
图 2
除了逻辑和算术计算外,DNA反应网络还被编程用作分子神经网络(图2)。想象一下,我们可以创建一个由DNA组成的网络,它能够学习和记忆像人脑那样的模式。科学家们实际上已经做到了这一点,他们构建了一个简单的DNA网络,能够识别和回忆特定的DNA序列。这就像给网络一个线索,它就能找到与之匹配的信息。这种技术的应用非常广泛,特别是在疾病诊断方面。由于某些疾病可以通过检测体内特定的分子标志来诊断,DNA计算可以帮助我们识别这些标志。比如,通过训练DNA分类器来识别不同的基因表达模式,科学家们可以区分细菌感染和病毒感染。还有一种方法是用DNA计算来分析miRNA(图2b),这有助于诊断癌症。
酶辅助电路
在合成生物学的自下而上方法中,DNA反应网络通常是通过使用具有高特异性和优越催化能力的简单酶促反应来构建的,与仅包含DNA的系统相比,这种方法更为高效。以聚合酶介导的DNA链置换为例,它通过更简单的电路结构和更快的计算速度实现了高效计算。DNA电路面临的一个主要挑战是“泄漏”,这通常是由于序列设计不完美和多链复合体由于合成错误而未能完美杂交导致的。但由于聚合酶介导的DNA链置换允许使用更简单的结构作为门复合体,因此它减轻了泄漏问题(图2c)。科学家们已经开发出了用于制备单链DNA和RNA电路的方法,这些电路使用核酸生物标志物作为输入,并通过酶促反应执行计算,这使得电路有可能与生物系统接口,用于诊断和生物传感等领域。要保持酶辅助DNA电路的稳定性能,需要通过仔细控制批次质量、缓冲条件和温度来确保酶的活性一致。除了聚合酶之外,CRISPR-Cas系统中的Cas核酸酶是DNA计算中特别有前景的一类酶。在这个系统中,Cas核酸酶绑定到一个引导RNA,该RNA通过互补碱基识别DNA目标。因此,通过合理设计互补的引导RNA,可以实现各种应用,比如控制Cas核酸酶活性以调节活细胞中的基因表达,以及用于体外应用(包括组织工程、生物电子学和诊断)的CRISPR响应性水凝胶。特定的Cas蛋白在目标结合后显示出不选择性的核酸酶活性,导致所有单链核酸的非选择性切割。通过利用这种不选择性的催化能力,Cas蛋白已被改造用于激活荧光单链DNA报告器,并与DNA电路集成,用于敏感的分子诊断。为了扩大Cas蛋白的应用范围,研究人员已经开发了基于Cas的DNA电路,使用部分催化不活跃的Cas蛋白来特异性切割特定链。另一方面,具有催化性质的DNA可以作为一种酶类物质。典型的,DNA酶是一种单链DNA结构,通过碱基配对识别目标核酸,随后执行切割或连接作用。利用它们的催化性质,DNA酶被用于构建逻辑门和控制DNA电路。然而,DNA酶的典型反应速率远低于基于蛋白的酶。
DNA纳米技术
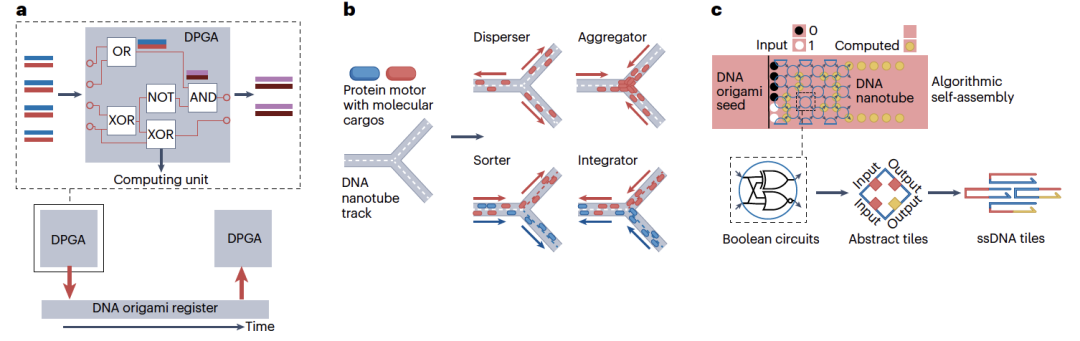
图 3
结构性DNA纳米技术始于Ned Seeman的开创性工作,他构建了固定的霍利戴交叉结构。此项工作随后扩展到了DNA片段的构建——由分支交叉组成的DNA纳米结构,这些结构作为自组装更高阶结构的基本构建块。在过去四十年中,这些初始片段得到了改进,使得能够控制DNA结构的几何形状、拓扑结构和连通性。通过开发DNA“折纸”技术,DNA自组装得到了进一步的推进,这是一种用于制作合理设计形状的强大且高效的技术。DNA折纸依赖于使用一个单链DNA脚手架,它与数百个短的定位片段一起折叠成DNA纳米结构。多个DNA折纸设计之前进一步组装成微米级结构,将结构性DNA纳米技术带入了显微世界。DNA纳米结构相比于DNA分子本身的一个主要优势是能够预先确定DNA电路的空间组织,以产生一个功能性且模块化的脚手架,用于解决特定任务,如货物排序、迷宫解决、有限状态机的构建和密码学。DNA纳米结构固有的表面可寻址性被利用来开发DNA折纸寄存器,这些寄存器可以临时存储中间数据并异步操作级联子电路,从而增加液相DNA数字计算的规模和电路深度(图3a)。在一个例子中,研究人员使用了一种混合方法在DNA纳米管上运输货物,利用蛋白质马达的高特异性和效率以及DNA纳米结构的可编程性。通过控制轨道上识别序列的方向,单向运动被用来执行任务,即分散、聚集、排序或整合分子货物(图3b)。除了作为脚手架之外,DNA纳米结构本身也可以作为DNA计算的单元。可重构的DNA纳米结构可以被编程与生物分子相互作用,并根据布尔逻辑在活细胞中做出决策。DNA纳米技术还可以用于细胞应用,如编程个别的DNA纳米结构,也可以组织和调节多个纳米结构以构建复杂的自组装网络进行DNA计算。
基于颗粒和隔室化的DNA电路
除了DNA纳米结构之外,其他材料如胶体、脂质、玻璃基板和合成隔室等也可能提供将DNA电路化的替代策略。由DNA反应网络指导的胶体系统能够产生有趣的模式和分布式决策行为,这激励了研究者将DNA逻辑电路与胶体系统集成,使得颗粒能够执行计算任务。比如,一种基于纳米颗粒的平台被用于构建DNA逻辑电路。在这个例子中,DNA功能化的纳米颗粒被放置在支撑的脂质双层上,作为计算单元。信息存储(记忆单元)和输出(报告单元)被固定在脂质膜上,而处理单元(浮动单元)可以自由移动并通过DNA杂交绑定到固定的颗粒上。记忆单元的状态依赖于溶液中提供的DNA输入,这反过来指导处理单元的行为,最终在报告模块上产生散射光信号作为输出。这种顺序策略允许构成一个感知器神经网络。此外,化学输入可以被处理并转换为使用马达化颗粒的机械输出。在这种情形下,微粒上的单链DNA与固定在表面的互补RNA链杂交。加入RNA核酸酶后,RNA链被水解,然后颗粒可以通过“burnt-bridge”机制移动。基于此,研究人员已经建立了颗粒之间的计算,并且最终电路的输出方式是颗粒的移动。通过使用不同大小和材料的颗粒来实现多路复用的颗粒输出。使用无标签输出避免了荧光素光谱带宽的限制,这可能增加并行计算的能力。然而,DNA电路在颗粒表面的固定需要仔细优化。由于DNA链直接暴露于环境中,链间的碱基配对需要高度正交,以最小化不期望的交叉反应。在二维表面上定位的DNA量相当少,导致DSD反应,特别是颗粒之间的反应速度变慢。在设计反应性DNA链与同一表面上的相邻DNA门互动的场景中,高密度植入是关键,以防止因扩散而导致的局部反应物的丢失。
DNA存储技术
随着基于DNA的分子计算技术的发展,DNA作为一种携带信息的天然分子,激发了研究人员将其作为合成数字数据存储介质的探索。相关生物医学领域,如基因组学和基因合成的进展,推动了这一领域的发展。使用DNA作为数据载体有几个优点。首先,DNA已被证明是一个强大的信息载体,其半衰期远远超过其他数字数据载体(至少几千年相比于几十年),并且通过在适当条件下存储DNA可以进一步延长这一时间。其次,因为信息是在分子层面上存储的,DNA基础数据存储的密度比典型数据载体高出大约六到八个数量级。第三,DNA在疾病和诊断中的重要性确保了读取和合成技术的相关性。最后,自然为研究人员提供了一个包含酶和其他生物分子的大工具箱,这些工具可以轻松地与DNA相互作用。
在DNA中编码和读取数据
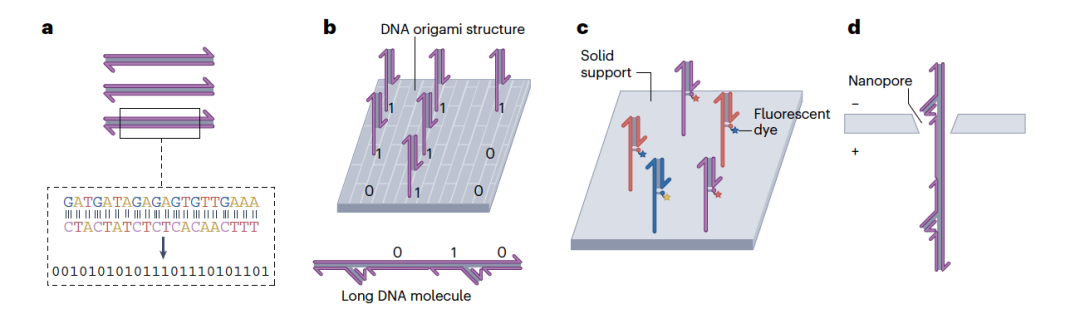
图 4
在DNA分子中编码数字数据可以通过多种方式实现,主要分为两种方法:核苷酸级编码(图4a)和结构编码(图4b)。作者首先描述在构成DNA分子的核苷酸序列中编码数字信息(比特)的方法。理论上,每个核苷酸可以存储2比特的信息(例如,使用A=00、C=01、T=10和G=11作为编码)。然而,由于防止某些模式被使用的限制、需要平衡GC含量以及防止长串同源多聚体(这些都可能导致DNA合成和测序过程中的错误)。此外,合成限制要求使用多个长度为100-200个核苷酸的DNA分子,而不是一个更长的DNA分子。常见的错误,如插入、缺失、替换或序列丢失,都可能对DNA中的数据存储产生负面影响。虽然通过物理冗余(即,存储相同序列的多份拷贝)可以保护DNA编码的数据免受这些和其他错误的影响,但这种方法严重限制了DNA作为存储介质的信息密度。因此,通常会使用某种形式的错误校正。对于DNA数据存储,最简单的错误校正形式包括在重叠片段中存储相同序列的多份拷贝,这样任何一个序列的丢失都不会导致信息的消失,冗余片段可以用来纠正替换错误。直接比较不同的编码算法具有挑战性,因此确切的性能难以量化,但基于核苷酸级数据编码的DNA数据存储已经展示了每个核苷酸1.14比特的逻辑数据密度,并实现了每克DNA17EB(艾字节)的物理密度。另一种方法是在DNA分子的结构中编码数据。虽然这种方法能够实现的数据密度低于核苷酸级编码,但其优势在于数据更易于读取。通常,基于结构的编码是通过在长DNA链的预定义位置引入结构元素(如切口或凸起结构)来实现的。结构编码的一个优点是可以使用生物DNA序列作为寄存器,这比完全合成的DNA更便宜。此外,信息可以通过将平面折纸结构上的位置映射到数据索引上来编码;比特则通过在给定位置标记寡核苷酸的缺席(0)或存在(1)来存储。
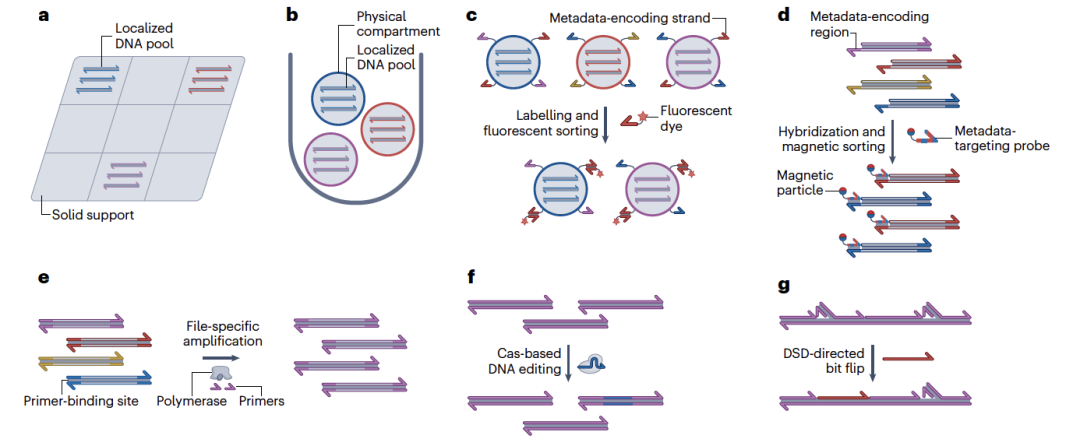
图 5
尽管最近取得了进展,但读取DNA编码数据仍然是一个成本高、耗时的过程,并且通常不需要同时读取所有数据。将编码数据的DNA分成更小的子集进行物理分离是一种可以避免过度读取的方法,研究人员为此开发了各种DNA组织方法。一个例子是在玻璃基板上组织不同的干燥DNA池(图5a);特定含DNA的斑点可以通过微流体技术重新水化并随后检索。为了实现更高的物理数据密度,最好将DNA编码数据分隔到不同的隔室中,这些隔室可以在一个容器中混合(图5b)。DNA数据存储的一种早期隔室化方法是将DNA编码数据存储在细菌内,利用自然界发展出来的天然DNA保护和修复途径。在这种方法中,数据被编码在随后整合到质粒中的DNA片段上,然后可以转化成细菌。然后可以通过培养细菌、质粒纯化和最后测序来检索细菌中的数据。由于细菌存储的DNA编码数据的数据密度相对较低,与干净DNA的存储相比,合成替代品可能提供更好的性能。合成替代品可以包括包裹DNA的玻璃颗粒,通过将DNA与水和空气隔离来分隔数据并增加长期稳定性,从而为长期存储稳定DNA。磁性颗粒和基于蛋白质-聚合物共轭的微隔室也被用来组织DNA编码数据,主要目标是使数据的可靠和重复访问成为可能。
编译 |
审稿 |
参考资料
1. 曾全晨. 王建民.Nat. Rev. Chem. | 用DNA作为计算和数据存储的通用化学基质
2. Yang, S., Bögels, B.W.A., Wang, F. et al. DNA as a universal chemical substrate for computing and data storage. Nat Rev Chem (2024). https://doi.org/10.1038/s41570-024-00576-4
这篇关于论文导读:用DNA作为计算和数据存储的通用化学基质的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!




