本文主要是介绍CUTTag技术:信噪比高,重复性好,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
CUT&Tag是蛋白质-DNA互作关系研究的新方法,2019年弗雷德哈钦森癌症研究中心的Henikoff博士在Nature Communication公开了该技术的详细结果与实验方案。与以往的ChIP-Seq、pA-MNase等方法相比,这种技术方法简便易行,信噪比高,重复性好,需要的细胞数量少至60个细胞,且有望将ChIP-Seq做到单细胞水平。
zui新开发的技术,使用核酸酶在靶标下切割和释放(CUT&RUN-sequencing)以及在靶标下切割和标记(CUT&Tag-sequencing),用于从低输入材料来绘制蛋白质-DNA相互作用,并显着提高了映射分辨率。然而,这两种技术的成本都较高。CUT&RUN-sequencing使用昂贵的 pA/MNase融合蛋白,该蛋白具有显着的A/T序列偏差,导致目标蛋白结合的DNA 区域谱受到MNase消化水平的严重影响。CUT&Tag-sequencing显示出相同的消化偏差,并且由于Tn5转座酶导致染色质的脱靶可及性,因此特异性较低。
为解决这些问题,EpiGentek开发了两种新技术—CUT&RUN Fast和CUT&Tag In-Place-Sequencing,用于快速富集与蛋白质结合的DNA,并绘制全基因组蛋白质/DNA相互作用图。新技术具有“两高一低”的优势:高分辨、高时效以及低输入。
艾美捷 EpiGente CUT&RUN和CUT&Tag技术来了。特别是 EpiQuik CUT&RUN M6A RNA富集试剂盒(P-9018)和EpiNext CUT&RUN M6A RNA-Seq试剂盒(P-9016),可以在zui小的非特异性背景水平上,从低输入RNA样本中特异性捕获和富集含有m6A修饰的RNA片段。富集的 RNA 特别适用于快速构建非条形码(单重)和条形码(多重)文库,允许以更小的偏差和高分辨率绘制 m6A 区域。
结果展示:
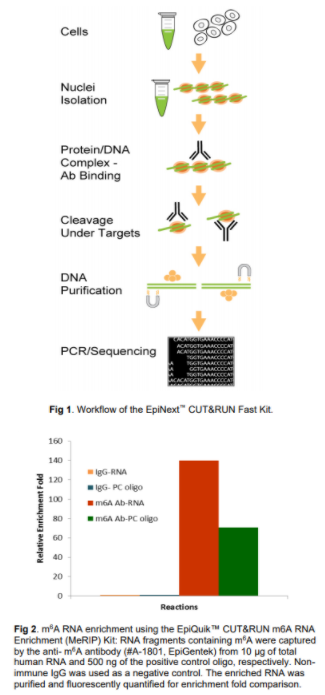
艾美捷 CUT&Tag技术 相关研究工具:
cat# 名称 时长
P-2028 EpiNext CUT&RUN Fast Kit <3小时
P-9018 EpiQuik CUT&RUN m6A RNA Enrichment (MeRIP) Kit <3小时
这篇关于CUTTag技术:信噪比高,重复性好的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!