本文主要是介绍单细胞RNA速率(RNA velocity),希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
RNA 丰度是单个细胞状态的有力指标。单细胞 RNA 测序可以以高定量精度、灵敏度和通量揭示 RNA 丰度。然而,这种方法仅捕获某个时间点的静态快照,这对分析胚胎发生或组织再生等时间分辨现象提出了挑战。在这里,我们表明,RNA 速度(RNA velocity)(基因表达状态的时间导数)可以通过区分常见单细胞 RNA 测序方案中未剪接和剪接的 mRNA 来直接估计。 RNA 速度是一种高维向量,可以预测单个细胞在数小时的时间尺度上的未来状态。我们验证了其在神经嵴谱系中的准确性,展示了其在多个已发表的数据集和技术平台上的使用,揭示了发育中的小鼠海马体的分支谱系树,并检查了人类胚胎大脑中的转录动力学。我们预计 RNA 速度将极大地帮助分析发育谱系和细胞动力学,特别是在人类中。
在发育过程中,分化发生的时间尺度为数小时至数天,这与 mRNA 的典型半衰期相当。可以利用新生(未剪接)和成熟(剪接)mRNA 的相对丰度来估计基因剪接和降解的速率,而不需要代谢标记,如之前在bulk中所示。我们推断,在单细胞 RNA 测序 (RNA-seq) 数据中可能可以检测到类似的信号,并且可以揭示整个转录组在动态过程中变化的速率和方向。
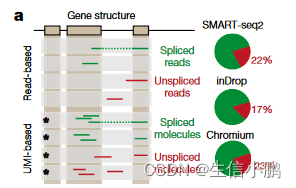
所有常见的单细胞 RNA 测序方案均依赖于oligo-dT 引物来富集聚腺苷酸化 mRNA 分子。尽管如此,在检查基于 SMART-seq2、STRT/C1、inDrop 和 10x Genomics Chromium 协议的单细胞 RNA-seq 数据集时,我们发现 15-25% 的读数包含未剪接的内含子序列(图 1a)。与之前在bulk4 (14.6%) 和single-cell5 (~20%) RNA-seq 中的观察结果一致。大多数此类读数源自内含子区域内的二次引发位置(扩展数据图 1)。在 10x Genomics Chromium 文库中,我们还发现来自更常见的内含子-polyT 序列的丰富不一致引发(扩展数据图 1),这可能是在 PCR 扩增过程中通过第一链 cDNA 引发而产生的。大量的内含子分子及其与外显子计数的相关性表明这些分子代表未剪接的前体 mRNA。通过对新转录的 RNA9 进行代谢标记,然后使用寡聚 dT 引发的单细胞标记逆转录 (STRT)10 进行 RNA 测序,证实了这一点(扩展数据图 2); 83% 的所有基因显示出与简单的一级动力学一致的表达时间过程,如果未剪接的读数代表新生 mRNA,则如预期的那样。
为了量化前体和成熟 mRNA 丰度之间的时间依赖性关系,我们假设了一个简单的转录动力学模型,其中剪接 mRNA 丰度(RNA 速度)的一阶时间导数由剪接 mRNA 产生之间的平衡决定未剪接的 mRNA 和 mRNA 降解(图 1b 和补充说明 1)(in which the first time derivative of the spliced mRNA abundance (RNA velocity) is determined by the balance between production of spliced mRNA from unspliced mRNA)。
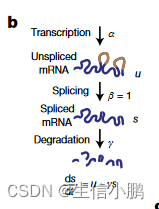
在这样的模型下,当转录速率 α 恒定时,渐近地接近稳态,剪接 (s) 和未剪接 (u) 分子的稳态丰度由 α 决定,并受限于固定斜率关系,其中 u = γs(补充说明 2 第 1 节)。平衡斜率 γ 结合了降解和剪接速率,捕获基因特异性调控特性、内含子和外显子长度的比率以及内部引发位点的数量。使用最近发表的小鼠组织概要11,我们发现多种细胞类型中大多数基因的稳态行为与单个固定斜率γ一致(扩展数据图3a-c)。然而,11% 的基因在不同的组织子集中显示出不同的斜率(扩展数据图 3d、e),表明组织特异性的选择性剪接(扩展数据图 3f)或降解率。
在动态过程中,转录率α的增加导致未剪接mRNA的快速增加,随后剪接mRNA的增加(图1c和补充说明2第1节),直到达到新的稳态。
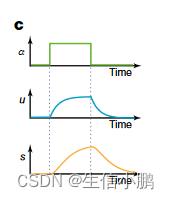
相反,转录速率的下降首先导致未剪接 mRNA 的快速下降,然后是剪接 mRNA 的减少。在基因表达诱导期间,未剪接的 mRNA 超出了基于平衡率 γ 的预期,而在抑制期间则相反(图 1d)。因此,未剪接和剪接 mRNA 丰度的平衡是成熟 mRNA 丰度未来状态的指标,也是细胞未来状态的指标。
为了证明如何使用这个简单的模型来推断未来的成熟 mRNA 丰度,我们检查了小鼠肝脏昼夜节律周期的批量 RNA-seq 测量的时间过程。每个时间点的未剪接 mRNA 水平始终与随后时间的剪接 mRNA 更加相似(图 1e),并且许多昼夜节律相关基因在上调过程中显示出相对于斜率 γ 而言预期的未剪接 mRNA 过量,以及相应的缺陷下调期间(图1f,g)。求解每个基因所提出的微分方程使我们能够推断整个昼夜节律周期中的每个测量结果,准确捕获昼夜节律周期进展的预期方向(图1h)。
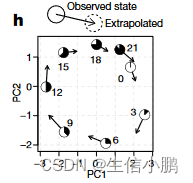
接下来,为了证明在单细胞测量中预测转录动态的能力,我们分析了最近发表的小鼠嗜铬细胞的单细胞 mRNA-seq 数据,这些数据是使用 SMARTseq获得的(图 2)。在发育过程中,很大一部分嗜铬细胞(肾上腺髓质的神经内分泌细胞)源自雪旺细胞前体,这提供了一个方便的测试案例,其中可以通过谱系追踪来验证分化方向。根据预测的稳态关系(图 2b、c),许多基因的相图显示了预期的偏差。
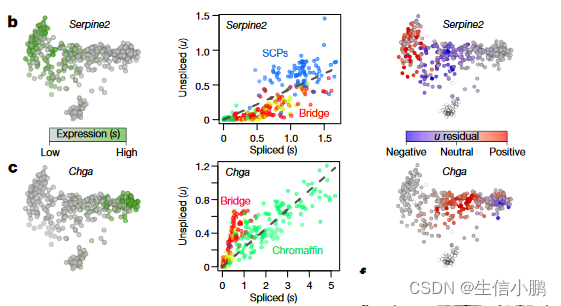
单个细胞的 RNA 速度估计准确地概括了该数据集中的转录动态,包括分化细胞向嗜铬细胞命运的一般运动(图 2d),以及朝向和远离中间分化状态的运动。该速度还捕获了涉及嗜铬分化的细胞周期动态,无论是在主成分分析 (PCA) 预测中还是在细胞周期相关基因的集中分析中(补充说明 2 第 5 节)。
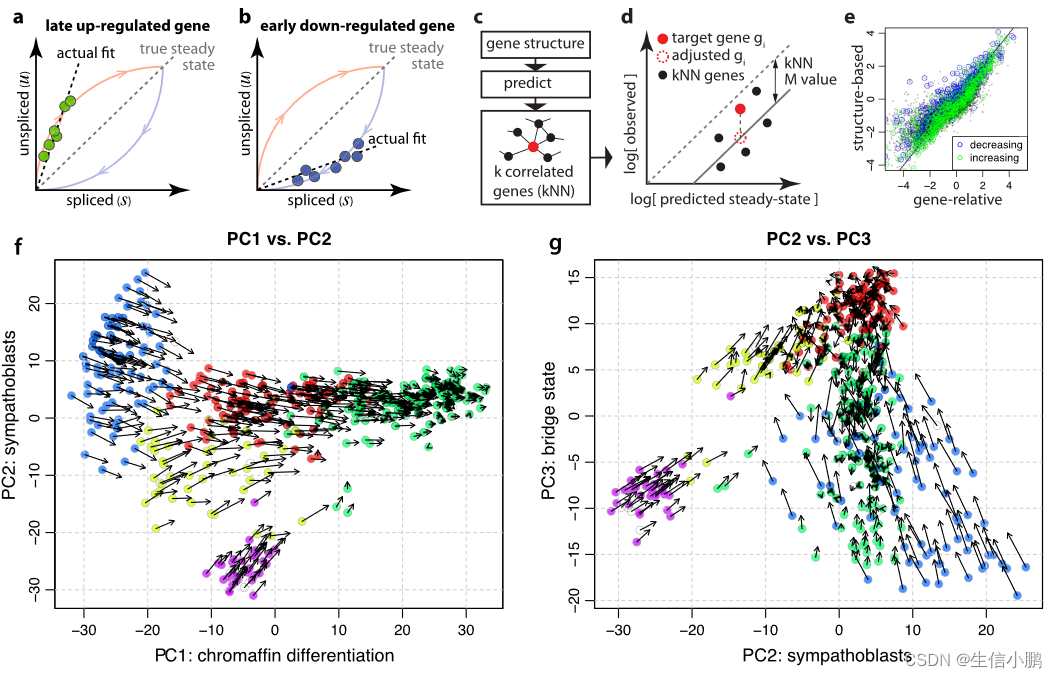
我们的速度估计程序包含多种功能,以适应剪接生物学的复杂性(补充说明1)。基因特异性平衡系数 γ 的估计是使用极端表达分位数的回归来进行的,即使大多数观察到的细胞处于稳定状态之外,也能确保稳健的估计(补充说明 2 第 2 节)。为了适应远远超出稳定状态的基因,我们还开发了一种基于基因结构的替代拟合(扩展数据图 4)。可以使用多种技术来可视化低维度的速度估计。
观察到的和外推的细胞状态可以共同嵌入到一个公共的低维空间中(The observed and extrapolated cell states can be jointly embedded in a common low-dimensional space)(例如图2d中的PCA)。
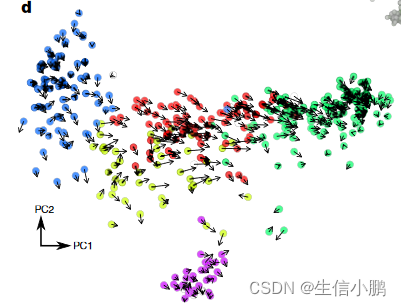
或者,基于外推状态与局部邻域中其他单元的相似性,可以将速度投影到现有的低维嵌入上,例如 t 分布随机邻域嵌入(t-SNE)(图 2h,参见补充说明1)。
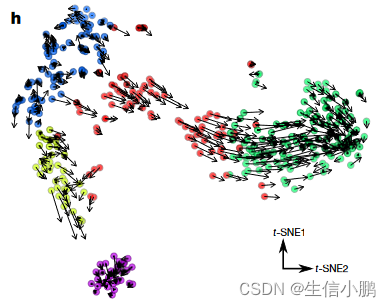
在大型数据集中,使用局部平均矢量场更容易可视化细胞速度的普遍模式(图 2i)。由于细胞可以同时沿着多个独立分量(例如分化、成熟和增殖)具有 RNA 速度,因此在解释低维表示时必须小心,因为在某一特定嵌入中缺乏表观速度的细胞仍然可以在某些子空间中具有相当大的速度,不是可视化的。细胞特异性 RNA 速度估计为细胞命运的定量建模提供了自然基础。代谢标记显示,对于大多数基因,剪接/未剪接比率的变化在 10-100 分钟后即可检测到(扩展数据图 2)。
另一方面,外推的有效时间尺度取决于所分析的生物过程。根据用 5-乙炔基-2′-脱氧尿苷 (EdU) 对嗜铬祖细胞进行脉冲标记(补充说明 2 第 6 节),我们估计我们能够推断出未来 2.5-3.8 小时(图 2f, g),这也与解决细胞周期事件的能力一致。然而,考虑到外推法的线性性质,这种预测时间尺度将取决于基因表达轨迹的形状(即表达流形的曲率)。通过在观察到的表达流形上追踪一系列小的外推步骤,可以在更长的时间尺度上预测细胞命运(补充说明 2 第 7 节)。
为了证明我们方法的通用性,我们分析了使用其他单细胞 RNA-seq 协议生成的数据。我们观察了小鼠骨髓中中性粒细胞成熟的转录动态,以及使用 inDrop 方案测量的小鼠皮层光诱导神经元激活的转录动态(扩展数据图 5),以及肠上皮细胞(扩展数据图 6)、少突胶质细胞的转录动态。使用 10x Genomics Chromium7 测量分化(扩展数据图 7)和海马体发育(见下文)。
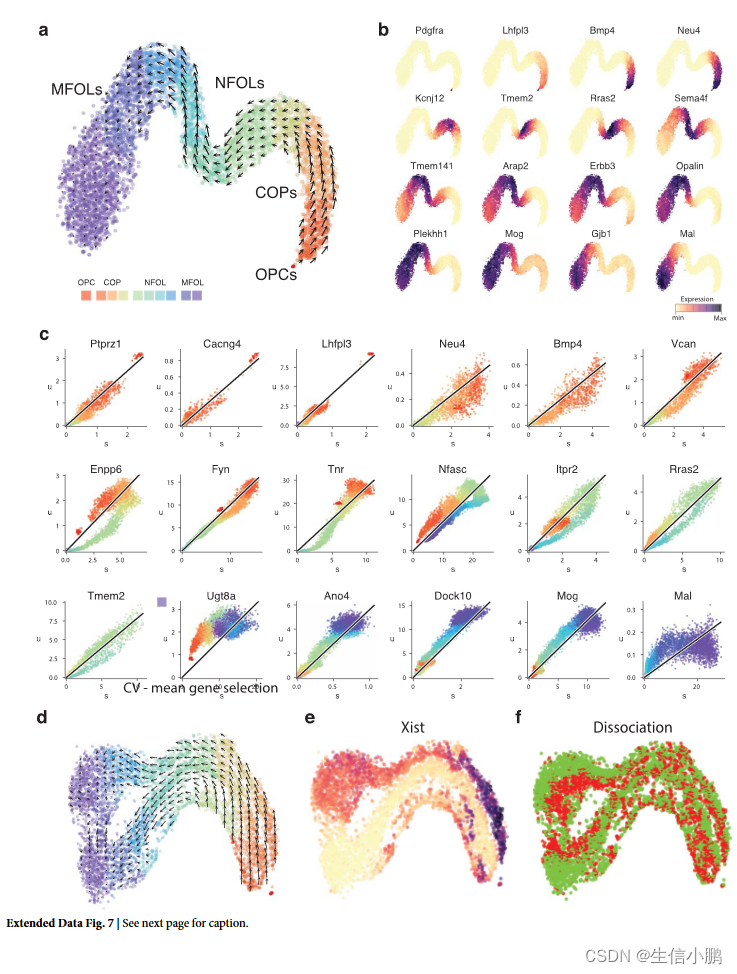
扩展数据图 7 | RNA 速度揭示了少突胶质细胞分化和髓鞘形成的动态。 a,t-SNE 投影显示青春期 (P20) 小鼠 (n = 6,307) 后脑 (pons) 少突胶质细胞谱系分化和髓鞘形成过程的景观。速度场反映了初始分化波的表达动态以及与髓鞘形成过程相关的后续表达变化。细胞簇按伪时间着色,如 c 中所示,以便于解释。 b,分化过程中标志性基因的表达模式。 Pdgfra 是少突胶质细胞前体 (OPC) 的典型标记,Neu4 标记定型少突胶质细胞前体 (COP),Tmem2 在新形成的少突胶质细胞 (NFOL) 中富集,Mog 的表达在髓磷脂形成少突胶质细胞的髓鞘形成过程开始时上调(MFOL)。 c,a 所示速度场下的一系列相图。 d,同一数据集的 t-SNE 投影和速度向量场,但使用更简单的特征选择进行分析,该特征选择保留了少突胶质细胞成熟度(性别和解剖日期)之上的其他变化轴。请注意,尽管群体分为 Xist+ 和 Xist− 轨迹,但速度场正确捕获了两个平行轨迹中从祖细胞到新形成的少突胶质细胞的进展。 e,Xist 的表达水平表明大部分额外变异是由动物性别驱动的。 f,细胞按实验进行当天着色。
RNA 速度的估计对于模型参数以及基因和细胞二次采样的变化是稳健的,最敏感的参数是预定义嵌入中速度可视化中使用的邻域大小(补充说明 2 第 10、11 节) 。大多数基因显示速度估计值与经验观察的表达导数之间呈正相关(扩展数据图 8),证实速度向量具有丰富的信息。
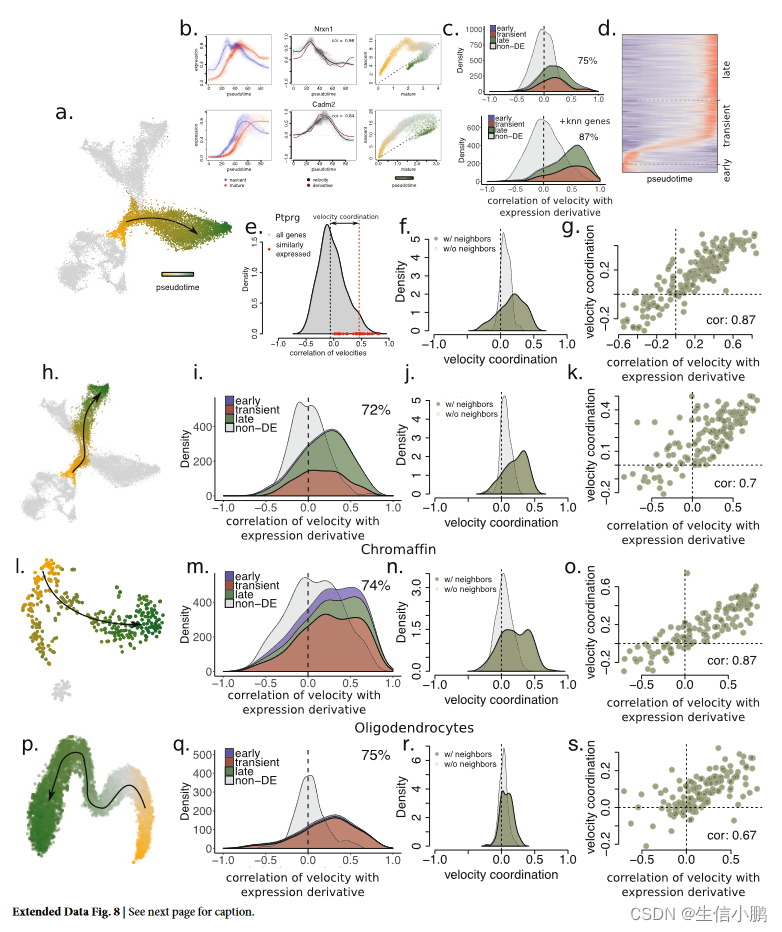
扩展数据图 8 |速度预测与观察到的表达导数的一致性。 a,小鼠海马数据集中颗粒神经元的成熟进程通过伪时间来近似(用主曲线估计)。 b,对于一对示例基因(行),图显示了沿着伪时间的未剪接和剪接的基因表达谱(左图)、根据经验估计的观察到的基因表达的平滑伪时间导数和估计的 RNA 速度(中图),如以及剪接和非剪接表达之间的关系(右图)。两个选定基因的速度估计与经验观察的导数高度相关,表明速度估计准确。 c,大多数(75%)沿着伪时间轨迹受到差异调节的基因与经验表达导数呈正相关。这些基因的分布根据轨迹相关基因的三类进行划分,如 d 所示。相比之下,沿着伪时间轨迹没有差异表达的基因的速度估计没有显示出这种相关性(灰色)。使用基因 kNN 聚类将有关共同调控基因的信息纳入速度估计(参见补充说明 1)可以显着提高速度预测的准确性(下图)。 d,轨迹相关基因根据其峰值表达时间分为早期、瞬时和晚期。 x 轴,按伪时间排序的单元格; y 轴,基因按其峰值表达时间排序。 e,在剪接表达模式方面与 Ptprg 密切相关的基因,也显示出其速度估计与 Ptprg 的高度相关性。为了评估共同调节基因速度的一致性程度,我们引入了给定基因的速度协调度量,作为共同调节基因的速度估计与所有基因的速度估计的平均相关性之间的差异。基因。对于 Ptprg,所比较的两个量用垂直虚线显示:灰色,与所有基因的平均速度相关性;红色,与顶级共同调控基因的平均速度相关性。速度协调为速度估计提供了公正的质量测量。 f,共同调控基因的速度是相关的。显示了具有共同调节基因的基因(即,在剪接表达模式方面具有良好相关基因邻居的基因,绿色)以及不具有共同调节基因的基因的基因速度协调值的分布。足够的共同调控基因(没有邻居,灰色)。 g,具有高速协调性的共同调控基因往往与经验导数具有高度相关性。显示 Spearman 相关系数。 h–k,锥体神经元成熟过程中的速度表现(h)。成熟过程中差异表达的基因与经验导数(i)具有高度相关性,共同调控的基因往往具有相关的速度估计(j),并且速度协调程度与其与经验导数(k)的相关性相关。 l,m,嗜铬细胞分化过程中的速度表现。 p–s,少突胶质细胞成熟过程中的速度表现。分析速度相关性的顶级共同调控基因的数量:200 (g)、150 (k、o、s)。
特定情况下的失败有几个明显的原因,包括仅观察到远离平衡的基因、非编码转录物的不均匀贡献以及导致测量群体中 γ 倍率的选择性剪接(补充说明 2 第 4 节)。
接下来,我们将 RNA 速度应用于发育中的小鼠海马体的分支谱系。去除血管和免疫细胞以及 GABA 能(γ-氨基丁酸释放)和 Cajal-Retzius 神经元(源自海马体外部)后,t-SNE 图显示了一个具有多个分支的复杂流形(图 3a)。我们使用已知的标记来识别对应于海马五个区域的星形胶质细胞、少突胶质细胞前体 (OPC)、齿状回颗粒神经元和锥体神经元的分支:下托、CA1、CA2、CA3 和门(扩展数据图 9)。单个基因的相图显示了沿流形的基因表达的特异性诱导和抑制(图3b和扩展数据图10)。例如,Pdgfra(OPCs的标记)在前OPCs中被诱导并在OPCs中维持;它在前 OPC 状态下显示出相应的正速度,但在 OPC 中显示为中性。类似地,Igfbpl1在神经母细胞中特异性表达,并显示从放射状胶质细胞到神经母细胞的正速度,但从神经母细胞到两个主要神经元分支的负速度。
RNA速度显示出朝向每个主要分支的强烈定向流(图3c和扩展数据图10),起源于排列成带状的一小群细胞(图3c,插图,虚线)。我们根据标记物的表达将这些细胞鉴定为放射状胶质细胞,包括Notch靶标Hes1和同源盒转录因子Hopx(扩展数据图9)。事实上,命运图谱此前已表明放射状胶质细胞是海马体谱系树的真正起源15。在速度场上使用马尔可夫随机游走模型,可以自动识别终端和根状态(图3c),这证明了RNA速度在无需事先了解发育过程的情况下定向谱系树的能力。一方面,速度指向星形胶质细胞(表达 Aqp4),而无需干预细胞分裂,或者进入前 OPC 状态,通过狭窄的通道进入增殖的 OPC。我们推测,狭窄的通道代表了少突胶质细胞谱系的定型时刻。在这个微观状态水平上,命运选择可能是一个非确定性过程,涉及基因表达向一种或另一种命运倾斜,一旦转录因子反馈回路建立,就会锁定最终命运16。比较从前 OPC 开始的细胞与从通向 OPC 的狭窄通道开始的细胞的未来状态的概率分布,发现了明显的差异——后一种细胞极有可能最终成为完全形成的 OPC,而前者则为完全形成的 OPC。可能保持在 OPC 之前的状态(图 3d)。
一些循环祖细胞(扩展数据图9b)表达神经源性转录因子(例如,Neurod2、Neurod4、Eomes),并且这些细胞显示出朝向未成熟神经母细胞状态的速度,通向神经母细胞上部的三个主要神经元分支。歧管。齿状回的颗粒神经元首先从海马体本身分裂,第二次分裂将海马细胞分别分为下托/CA1和CA2-4(扩展数据图9、10),与主要的功能和解剖学细分一致海马体。分支谱系的详细单细胞视图使我们能够质疑命运的选择。检查位于 CA 和颗粒命运之间分支点入口处的两个相邻神经母细胞(图 3e),我们发现虽然它们当前的状态是邻居(在基因表达空间中),但它们的未来已经倾向于不同的命运,区分通过激活 Prox1(图 3c,插图)。与这些发现一致的是,Prox1 是颗粒神经元形成所必需的,并且当 Prox1 被删除时,成神经细胞反而采用锥体神经元命运。
为了证明人类胚胎中的 RNA 速度是可检测的,我们在受孕后十周对发育中的人类前脑进行了基于液滴的单细胞 mRNA 测序,重点关注谷氨酸能神经元谱系(图 4a)。
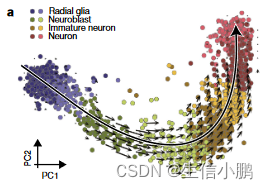
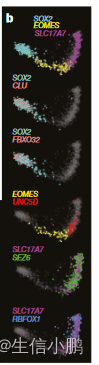
我们发现了一种强速度模式,源自增殖的祖细胞状态(放射状胶质细胞),并通过一系列中间神经母细胞阶段,发展到表达 SLC17A7(囊泡谷氨酸转运蛋白(也称为 VGLUT1))的更成熟的分化谷氨酸能神经元。前脑兴奋性神经元)。我们通过多重原位杂交验证了皮质神经元发育的已知和新标记物的表达(图4b,c),证实了心室区(放射状胶质细胞;由SOX2标记)中的CLU和FBXO32的预测表达,以及脑室区中的UNC5D的预测表达。中间区(神经母细胞;用 EOMES 标记)以及皮质板中的 SEZ6 和 RBFOX1(神经元;用 SLC17A7 标记)。这些基因在组织中的分层表达(图4c)与其在单细胞RNA-seq数据中表达的伪时间分布密切对应(图4b)。

我们使用主曲线分析根据分化假时间对细胞进行排序,并检查了人类原代细胞中转录的时间进程。我们证实,在上调和下调过程中,未剪接的 mRNA 始终先于剪接的 mRNA(图 4d)。
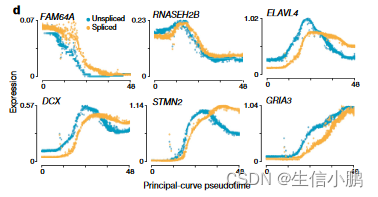
我们观察到快速和慢速动力学。例如,RNASEH2B 表现出快速动力学,未剪接和剪接的 RNA 之间几乎没有差异。相比之下,DCX、ELAVL4 和 STMN2 等基因显示出最初的快速转录爆发的证据,随后是水平降低的持续转录(如未剪接 RNA 曲线的形状所证明的,图 4d),随后是剪接转录本轨迹明显延迟。这种具有超调的动态诱导已被提出有助于快速诱导具有缓慢降解动力学的基因,但这在人类胚胎中尚不可能进行研究。
结论
由于 RNA 速度是以真实的转录动力学为基础的,因此这种方法有望为我们理解细胞分化过程中基因表达空间的动态提供更坚实的定量基础。我们设想未来的流形学习算法能够在 RNA 速度的基础上同时拟合流形和该流形上的动力学。RNA速度已经启用了详细的研究整个生物体的动态过程,并将极大地促进谱系分析,特别是人类胚胎中的谱系分析。
这篇文章是对RNA速率算法的介绍,一篇nature 的letter,作者的文章内容很多,也很深入,仔细读其实还是有点云里雾里,还需要一些背景知识。但是需要使用的话,有成熟的Rpackage和python包。可以使用起来。
Code availability. The software described in this paper, in the form of a pipeline called Velocyto (from velox, quick and κύτος, cell) is available at http://velocyto. org. This includes complete analysis libraries in R and Python, as well as R and Python notebooks.
学习文献
La Manno, G., Soldatov, R., Zeisel, A. et al. RNA velocity of single cells. Nature 560, 494–498 (2018). https://doi.org/10.1038/s41586-018-0414-6


这篇关于单细胞RNA速率(RNA velocity)的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!