本文主要是介绍R语言【rWCVP】——rWCVP生成可发表级别的物种发现记录矩阵,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
rWCVP生成可发表级别的物种发现记录矩阵
- 介绍
- 1. 查询一组示例数据
- 2. 生成和格式化出现矩阵
- 3. 额外地对国家进行处理
介绍
世界维管植物名录(WCVP)提供了已知的>340,000种维管植物物种的分布数据。该分布数据可用于构建植物物种名录的发现记录矩阵,rWCVP可以提供帮助。
除了 rWCVP 之外,还可以使用 tidyverse 包进行数据操作和绘图,并使用 gt 包来格式化表格。
先做好准备工作
library(rWCVP)
library(tidyverse)
library(gt)
在此示例中,使用==管道运算符 (%>%) 和 dplyr语法 ==- 如果不熟悉这些,我建议查看 https://dplyr.tidyverse.org/ 和其中的一些帮助页面。
1. 查询一组示例数据
对于这个例子,我们没有想要检查的特定区域或植物组,但这让我们有机会展示rWCVP中的其他功能之一!
我们想要一组物种,a)不太大,b)分布在几个WGSRPD 3级区域。巴西就是不错的选择,因为它有五个3级区域(对于这个目的来说,这是一个不错的数字,因为该表格将适合纵向页面)。让我们看看是否有一些大小不错的示例属,使用 wcvp_summary 函数:
wcvp_summary(taxon = "Myrtaceae", taxon_rank = "family", area=get_wgsrpd3_codes("Brazil"), grouping_var = "genus")%>%wcvp_summary_gt()
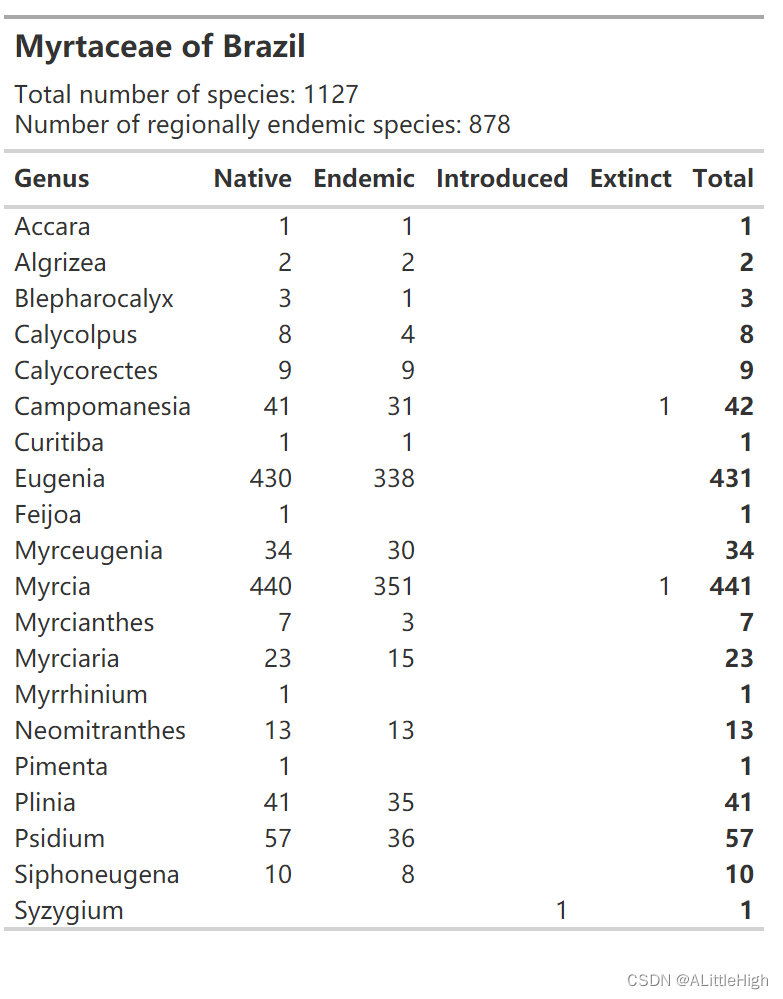
Calycolpus看起来又漂亮又整齐——让我们看看这 8 个物种是如何分布在 5 个区域的。
我们可以使用相同的函数,但限制我们的分类单元并将我们的分组变量更改为 area。
wcvp_summary(taxon = "Calycolpus", taxon_rank = "genus", area=get_wgsrpd3_codes("Brazil"), grouping_var = "area_code_l3")%>%wcvp_summary_gt()

嗯,也许有点太小了 - 它只出现在 5 个区域中的 3 个。Myrciaria呢?
wcvp_summary(taxon = "Myrciaria", taxon_rank = "genus", area=get_wgsrpd3_codes("Brazil"), grouping_var = "area_code_l3")%>%wcvp_summary_gt()

2. 生成和格式化出现矩阵
为该属生成出现矩阵就像使用 generate_occurence_matrix 函数一样简单。
m = wcvp_occ_mat(taxon = "Myrciaria", taxon_rank = "genus", area=get_wgsrpd3_codes("Brazil"))
# A tibble: 23 × 7plant_name_id taxon_name BZC BZE BZL BZN BZS<dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>1 473796 Myrciaria alagoana 0 1 0 0 02 534878 Myrciaria alta 0 0 1 0 03 534776 Myrciaria cambuca 0 1 1 0 04 131799 Myrciaria cordata 0 0 0 1 05 131802 Myrciaria cuspidata 1 1 1 0 16 131803 Myrciaria delicatula 1 0 1 0 17 131806 Myrciaria disticha 0 1 1 0 08 131810 Myrciaria dubia 1 0 0 1 09 491614 Myrciaria evanida 0 0 1 0 0
10 131814 Myrciaria ferruginea 0 1 1 0 0
# ℹ 13 more rows
# ℹ Use `print(n = ...)` to see more rows
没关系,但是我们可以使用 gt 包让它更漂亮。让我们执行以下操作:
- 删除 WCVP ID 列
- 将taxon_id更改为“物种”
- 使物种名称为斜体
- 加粗列标题
- 减少文本周围的空间,使字体大小为 12
- 删除内部边框
- 将 1 和 0 更改为 X 和空白
m_gt = m %>% select(-plant_name_id) %>%gt() %>%cols_label(taxon_name = "Species") %>%tab_style(style = cell_text(style = "italic"),locations = cells_body(columns = taxon_name)) %>%tab_options(column_labels.font.weight = "bold",data_row.padding = px(1),table.font.size = 12,table_body.hlines.color = "transparent",) %>%text_transform(locations = cells_body(),fn = function(x) {ifelse(x==0, "", x)}) %>%text_transform(locations = cells_body(),fn = function(x){ifelse(x==1, "X", x)})m_gt
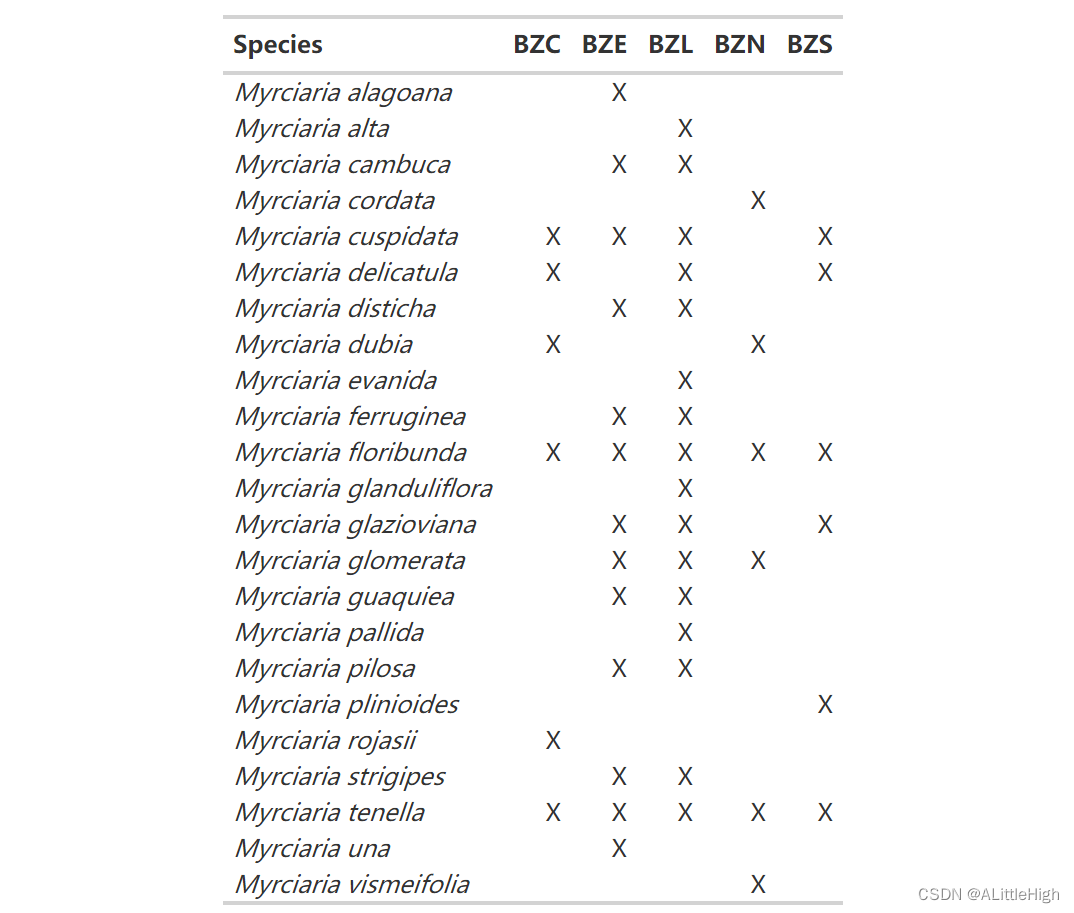
好多了!我们可以将此 gt 表另存为 HTML 表或图片。如果我们打算再制作几个表格,我们可以通过将表格样式保存为主题来节省空间(有关更多详细信息,请参阅 https://themockup.blog/posts/2020-09-26-functions-and-themes-for-gt-tables/)
occ_mat_theme <- function(x){x %>% cols_label(taxon_name = "Species") %>% #make species names italictab_style(style=cell_text(style="italic"),locations = cells_body(columns= taxon_name)) %>% tab_options(# some nice formattingcolumn_labels.font.weight = "bold",data_row.padding = px(1),table.font.size = 12,table_body.hlines.color = "transparent",) %>%# change the zeroes into blankstext_transform(locations = cells_body(),fn = function(x){ifelse(x == 0, "", x)}) %>% # change the 1s into Xtext_transform(locations = cells_body(),fn = function(x){ifelse(x == 1, "X", x)})
}
gt() 的最大问题是它不支持 Word - 要直接导出到 docx 文件,请查看 flextable 。包括或排除发生类型 如果我们只想知道本地或引进物种怎么办?此函数可以选择筛选其中一个。巴西Myrciaria在这方面看起来不是很有趣(我们可以从汇总表中看到只引入了一个物种),所以让我们看看一个更具入侵性的群体 - 北欧的Poa(2级区域)。
wcvp_summary(taxon = "Poa", taxon_rank = "genus", area=get_wgsrpd3_codes("Northern Europe"), grouping_var = "area_code_l3") %>% wcvp_summary_gt()
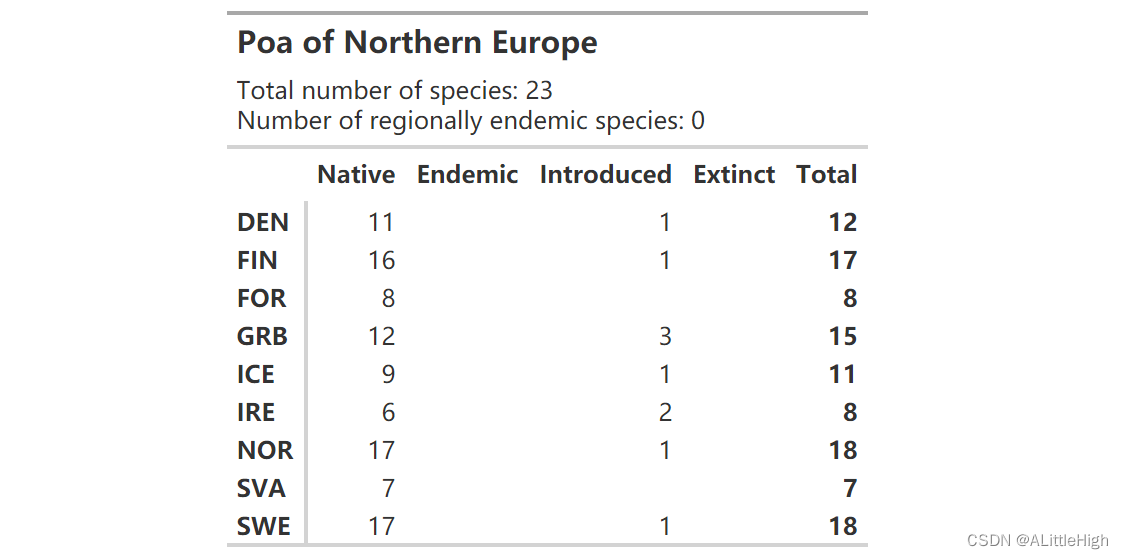
还有一些工作要做。首先,让我们只看本地物种:
p = wcvp_occ_mat(taxon = "Poa", taxon_rank = "genus", area=get_wgsrpd3_codes("Northern Europe"), introduced = FALSE, extinct = FALSE, location_doubtful = FALSE)
p
# A tibble: 20 × 11plant_name_id taxon_name DEN FIN FOR GRB ICE IRE<dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>1 435004 Poa abbreviata 0 0 0 0 0 02 435078 Poa alpigena 0 1 1 0 1 03 435085 Poa alpina 0 1 1 1 1 14 435167 Poa angustifol… 1 1 0 1 0 05 435194 Poa annua 1 1 1 1 1 16 435235 Poa arctica 0 1 0 0 0 07 435458 Poa bulbosa 1 1 0 1 0 08 435622 Poa compressa 1 1 0 0 0 09 435932 Poa flexuosa 0 0 0 1 1 0
10 435996 Poa glauca 0 1 1 1 1 0
11 436089 Poa hartzii 0 0 0 0 0 0
12 436146 Poa humilis 1 1 1 1 1 1
13 436189 Poa infirma 0 0 0 1 0 0
14 436383 Poa lindebergii 0 1 0 0 0 0
15 436600 Poa nemoralis 1 1 1 1 1 1
16 436739 Poa palustris 1 1 0 1 0 0
17 436906 Poa pratensis 1 1 1 1 1 1
18 437092 Poa remota 1 1 0 0 0 0
19 437424 Poa supina 1 1 0 0 0 0
20 437547 Poa trivialis 1 1 1 1 1 1
# ℹ 3 more variables: NOR <dbl>, SVA <dbl>, SWE <dbl>
我们可以像上面一样格式化这个矩阵,但让我们跳过那些步骤,直接处理引入的物种。我们正在做与以前相同的格式设置,但也添加一个标题 - html 函数可以将我们的属名和所有内容斜体化!
p = wcvp_occ_mat(taxon = "Poa", taxon_rank = "genus", area=get_wgsrpd3_codes("Northern Europe"),native = FALSE, introduced = TRUE, extinct = FALSE, location_doubtful = FALSE)
p %>% select(-plant_name_id) %>% #remove ID colgt() %>% occ_mat_theme() %>% #the theme we defined above#add a headertab_header(title=html("Introduced <em>Poa</em> species in Northern Europe"))
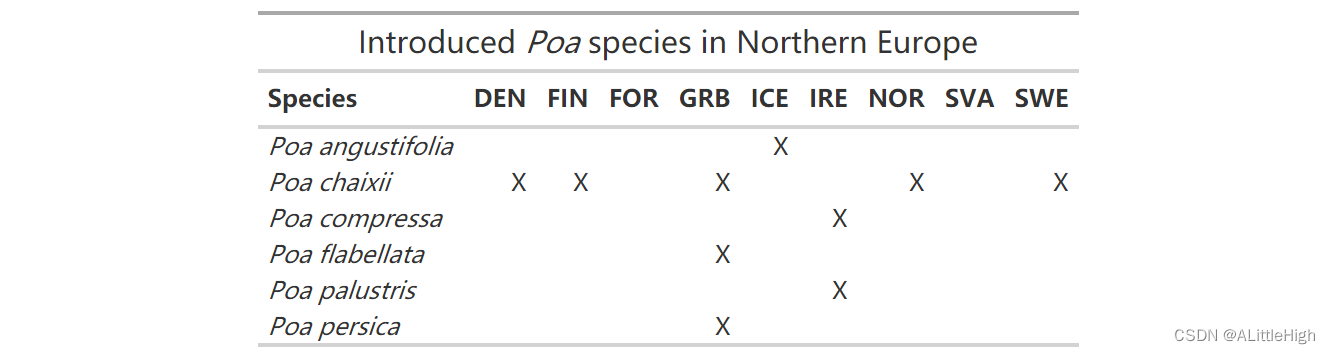
3. 额外地对国家进行处理
使用 gt 创建的表非常灵活 - 假设我们想查看美加边境出现的记录:
f = wcvp_occ_mat("Fritillaria", "genus", area=c("WAS","ORE","IDA","MNT","ABT","BRC"))
f_gt = f %>%select(-plant_name_id) %>%gt() %>%occ_mat_theme() %>%tab_header(title = html("<em>Fritillaria</em> species in Northwest USA and Southwest Canada"))
f_gt
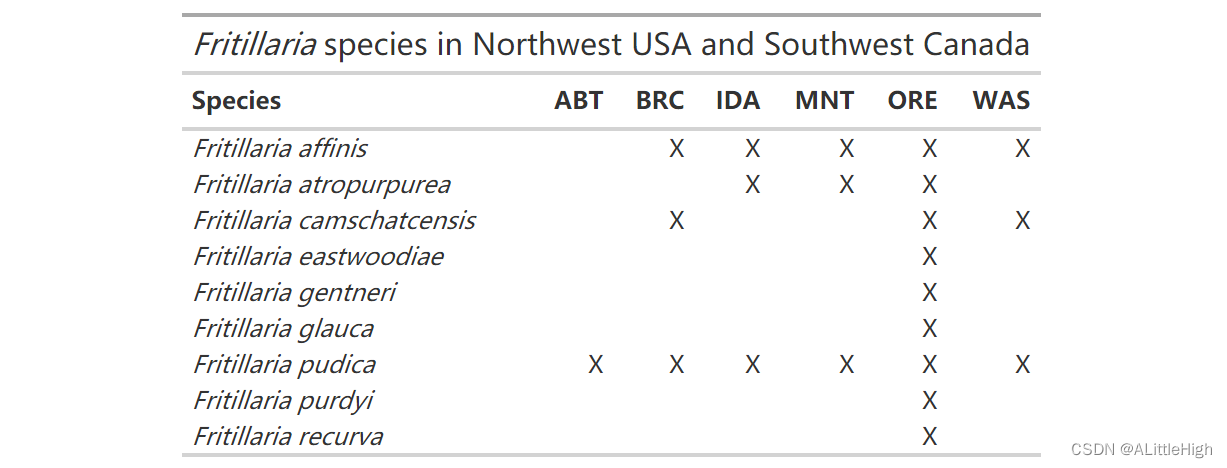
了解哪些代码在美国,哪些在加拿大非常有用。我们可以使用 rWCVP 中包含的数据来创建检索表。
wgsrpd_mapping %>%filter(LEVEL3_COD %in% c("WAS","ORE","IDA","MNT","ABT","BRC")) %>%select(LEVEL3_NAM, LEVEL3_COD, COUNTRY) %>%gt() %>%tab_options(column_labels.font.weight = "bold",data_row.padding = px(1),table.font.size = 12,table_body.hlines.color = "transparent",)
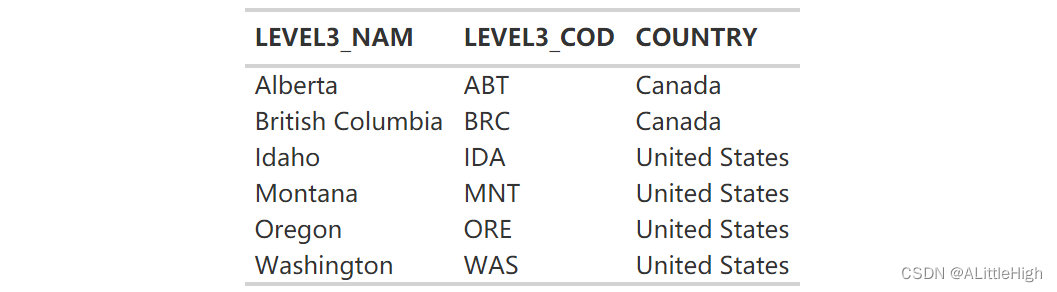
不过,将其放在发生矩阵上确实会更好。输入 tab_spanner():
f_gt %>%tab_spanner(label = "United States", columns = c(IDA, MNT, ORE, WAS)) %>%tab_spanner(label = "Canada", columns = c(ABT, BRC))
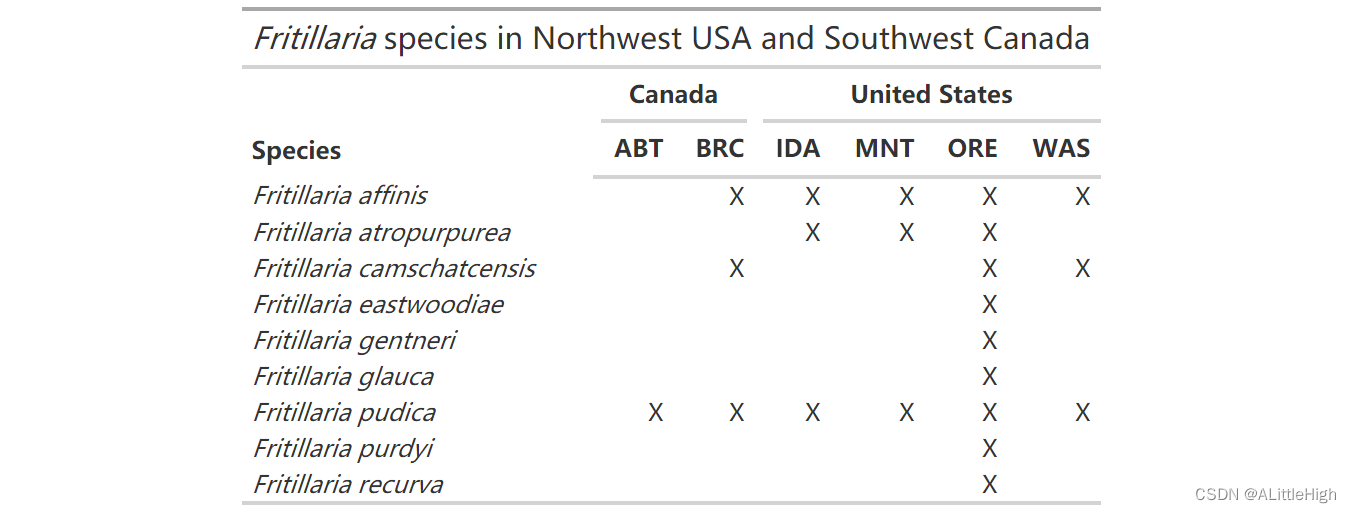
gt 还有很多事情可以做 - 请参阅 https://gt.rstudio.com/ 以获取帮助、示例和文档。
这篇关于R语言【rWCVP】——rWCVP生成可发表级别的物种发现记录矩阵的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







