本文主要是介绍Cancer Cell全新组学联合-癌症微生物探索,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!

在日新月异的多组学时代,各组学间的关联分析在生物学机制研究中变得尤为重要。而scRNA-seq技术的出现,为单细胞水平的多组学关联分析提供了强有力的支撑。凌恩生物引入单细胞平台,凭借本身丰富的微生态研究经验,是该类研究方案的优质合作伙伴。
本期为大家介绍一篇发表于Cancer Cell的文章,以单细胞转录组和微生物组关联分析为切入点,探究了宿主-微生物互作对胰腺癌中造成的影响。(DOI: 10.1016/j.ccell.2022.09.009)

-
期刊:Cancer Cell
-
影响因子:50.3
-
发表时间:2022.10
-
样本类型:胰腺组织
研究背景
微生物不仅存在于多种癌症组织中,即使在广泛被认为无菌的器官中也有被发现。为探究微生物与胰腺癌发生的关系,结合人类胰腺癌队列开发了一种宿主-微生物组相互作用的单细胞分析方法SAHMI,可从scRNA-seq数据中鉴定出可信度较高的微生物序列。
研究思路
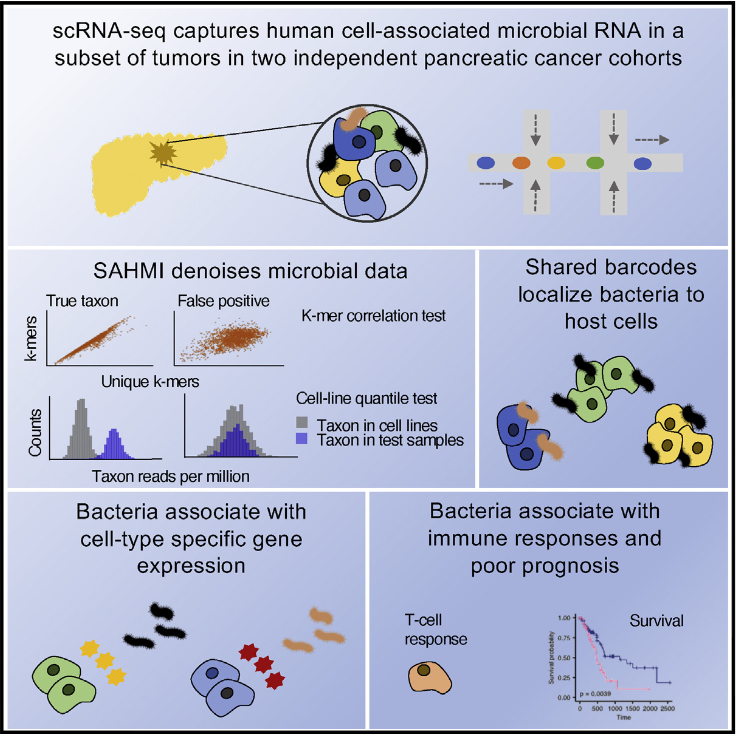
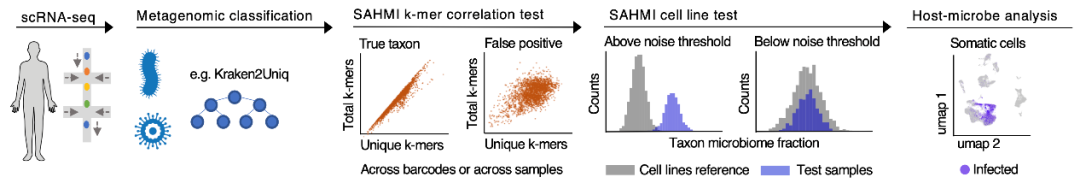
实验流程图
主要结论
1、在scRNA-seq数据中检测并验证微生物序列
利用2个人类胰腺癌队列产生的数据使用WGS,RNA-seq,scRNA-seq和16S rRNA检测样本中的微生物。scRNA-seq数据经过过滤之后只剩下0.01% reads被鉴定为微生物序列,鉴定污染和假阳性后确定这是样本中真实存在的微生物。比对上参考基因组的微生物序列没有出现区域偏好性,说明这些序列并非由实验或测序引入。作者使用mBodyMap数据库来推测SAHMI鉴定到的微生物来源(图1H),结果发现除了呼吸道和血液中发现的微生物外,样本中的微生物主要来自胃肠道(GI)区。
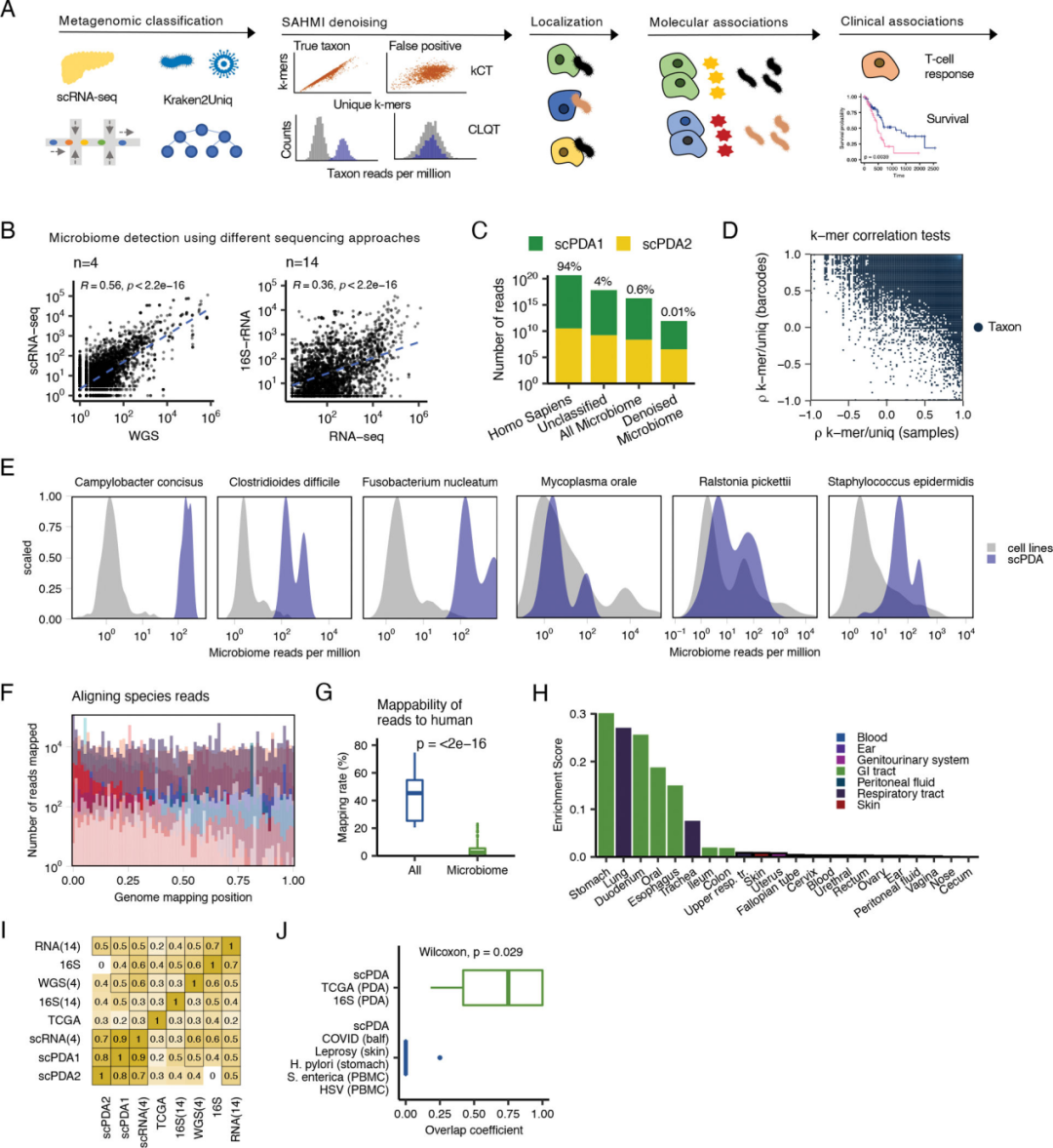
图1 PDA微生物组的检测和验证
2、细胞关联的细菌出现在胰腺肿瘤的部分细胞亚群中
在55个样本中鉴定出48个带有细菌的样本(图2A、B),将带有细菌标签的barcode的样本与其单细胞数据整合,在UMAP降维分析中,发现与微生物存在互作的细胞成簇存在(图2D),并非分散存在。按细胞类型计数来看,肿瘤细胞关联的细菌数量最多(图2E)。
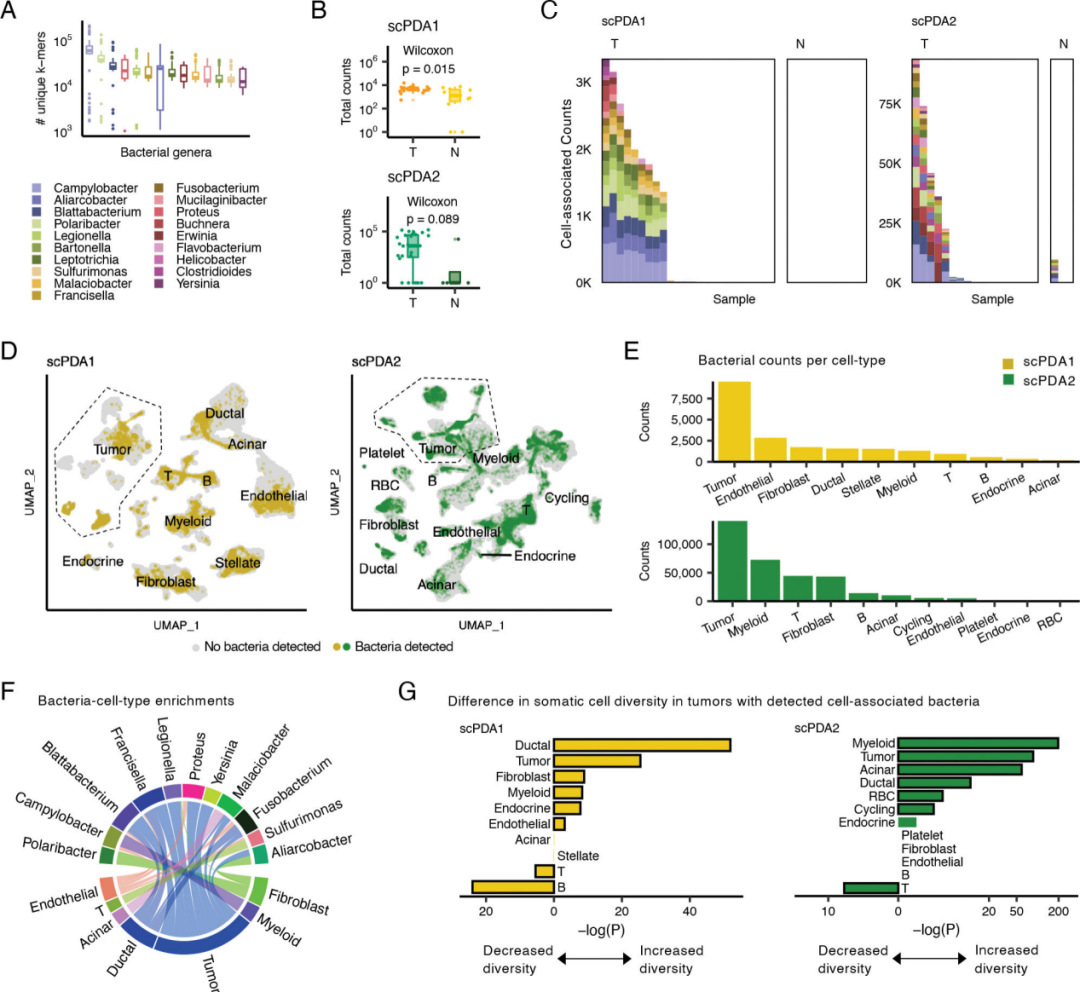
图2 部分肿瘤有细胞相关细菌
3 、细菌与细胞类型多样性和活动有关
分析与特定细菌互作的细胞中基因的差异表达中发现,在癌细胞中发现了较多数量的差异表达基因(图3B),包含角蛋白、黏蛋白和油脂相关基因(图3C),富集分析也鉴定出上述的DEGs(图3E)。
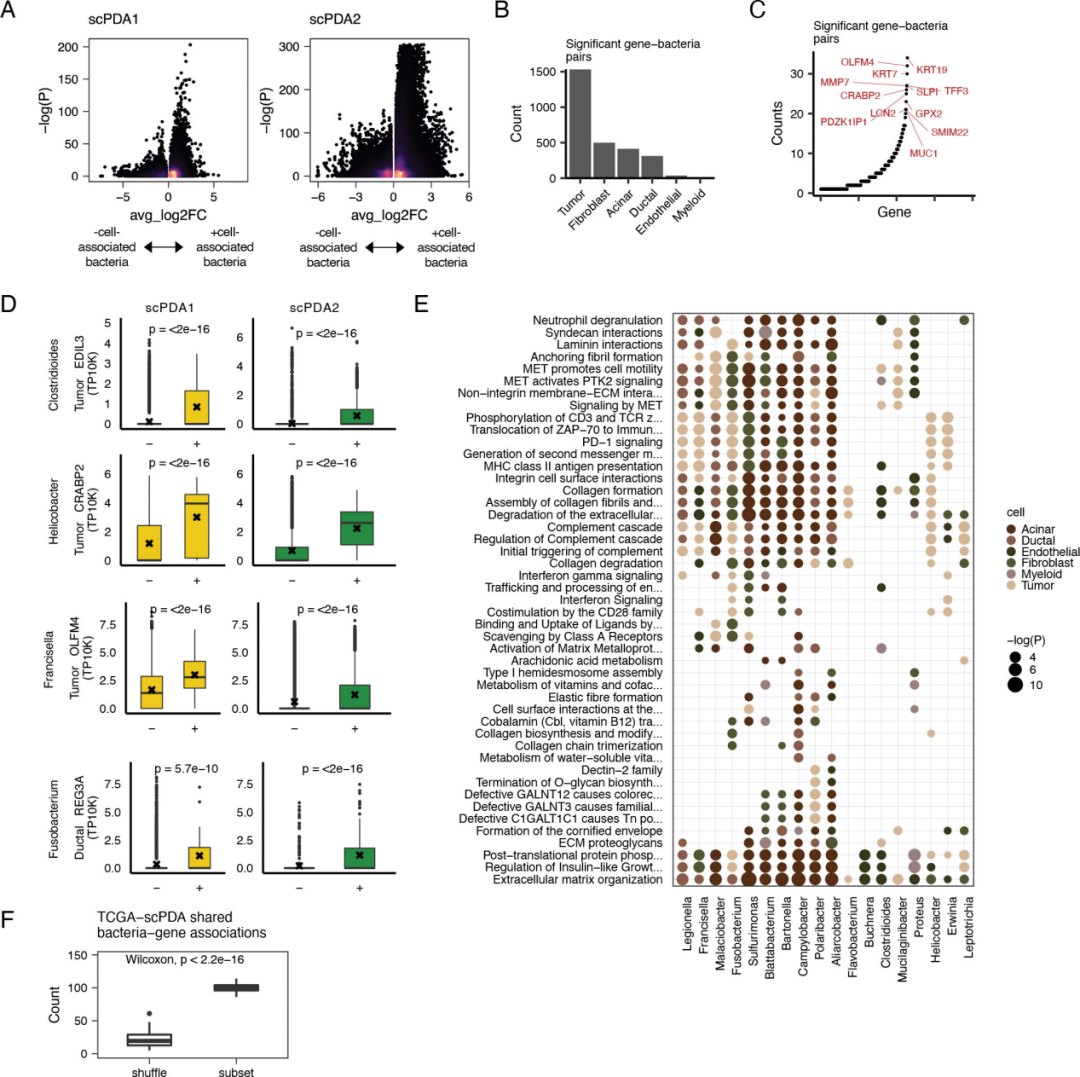
图3 细菌与细胞类型的特异性活动有关
4 、存在细菌关联的肿瘤中T细胞被激活
基于marker标注了T细胞亚群,包括调控、记忆、效应和自然杀伤T细胞(图4A, B),来自肿瘤组织且存在细菌关联的T细胞比没有细菌关联的具有更多的DEGs,并且在多个相关的通路中富集(图4C, D)。
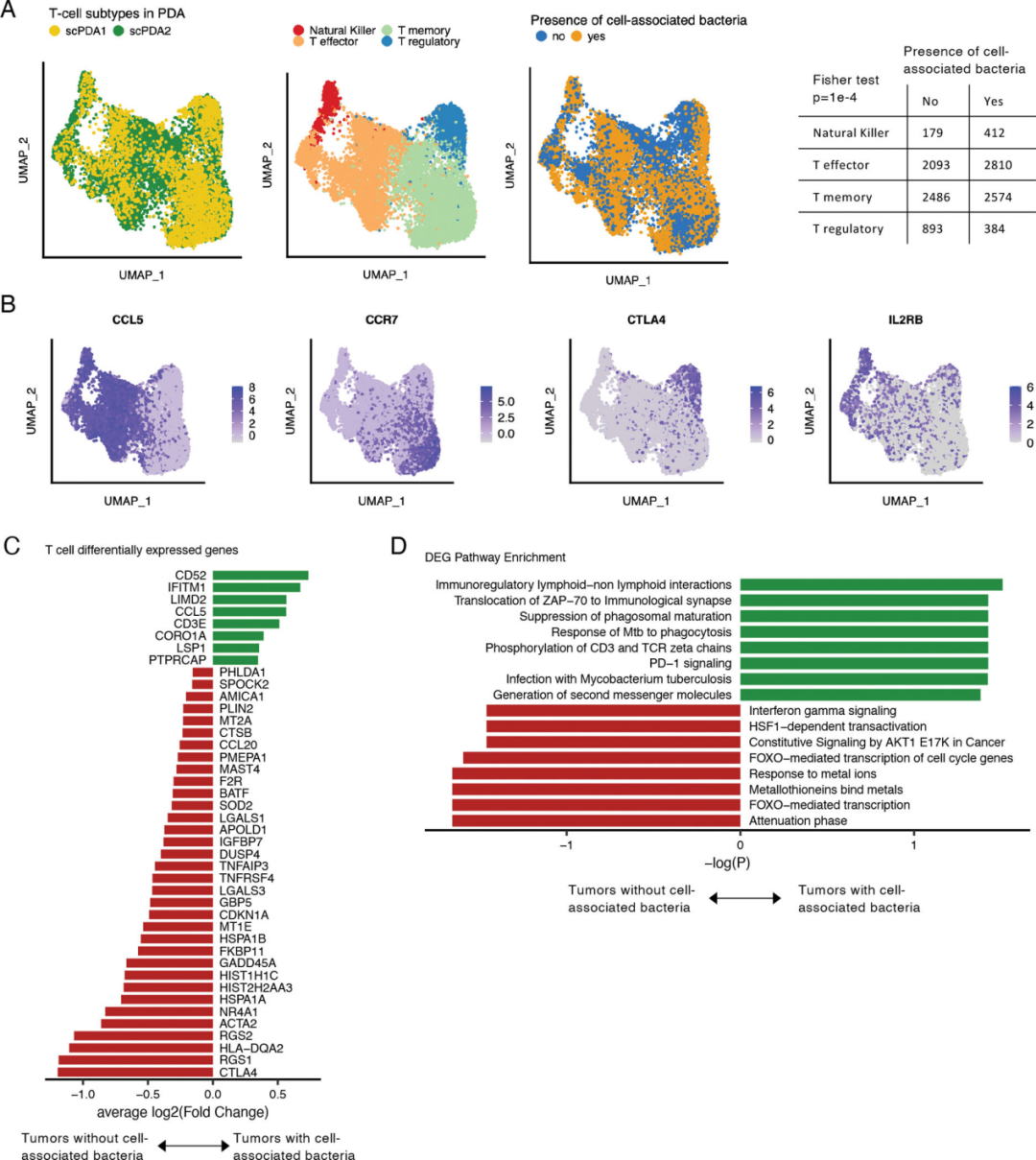
图4 肿瘤中的T细胞亚型
5 、基于微生物组对患者进行生存分析
研究了肿瘤内微生物特征是否与总生存期相关,并区分肿瘤中是否有互作微生物,预测胰腺癌肿瘤组织的微生物感染状态。根据其基因表达谱区分T细胞感染和肿瘤微环境反应,最终发现肿瘤微生物特征与胰腺癌患者的预后具有显著相关性(图5)。
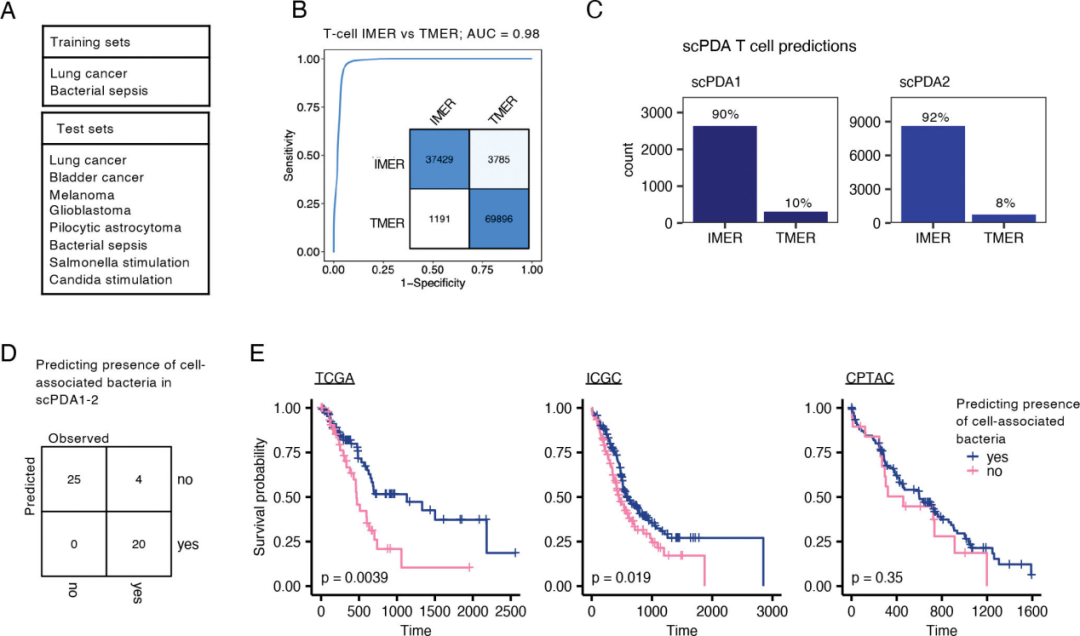
图5 T细胞微环境反应和生存模型
研究结论
使用SAHMI研究了两个人类胰腺癌队列中肿瘤与微生物组的相互作用。在一部分肿瘤中发现了体细胞相关细菌,而在非恶性组织中几乎没有这些细菌。这些细菌主要与肿瘤细胞配对,并且它们的存在与细胞类型特异性基因表达和通路活性(包括细胞运动和免疫信号传导)相关。建模结果表明,肿瘤浸润的淋巴细胞与来自感染组织的T细胞非常相似。最后,利用多个独立的数据集,细胞相关细菌的特征可预测临床预后。肿瘤-微生物组交互作用可能调节胰腺癌的肿瘤发生,对临床治疗具有意义。
参考文献
Ghaddar B, et al. Tumor microbiome links cellular programs and immunity in pancreatic cancer. Cancer Cell. 2022.
这篇关于Cancer Cell全新组学联合-癌症微生物探索的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!