本文主要是介绍python blast在线比对 每天最多数万条,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
python blast在线比对、每天可达数万条
背景:在碱基序列,如
TRINITY_DN931509_c0_g1_i1
GCACGTTCTTCTCGATGCTGACGGAGCGAGGCGCGCTGGCGGAGCTGATGGC
GGCGCAGCTGGAGCGAGGGCGCACGCCGCGGGTGCGCCGGCGCCGGCCGC
GCCCCAGGCCCAGGGCCAGGCCGCGCCCGAGGCCGAGGAGTAGGAAATAAT
GATCTGTGACGAAACTAACCCACCCCTCGAGCTTGCGGAAATGCTGGAATCTG
CGACGTAGGCACTACTCGAACTTGAG
需要比对(即Basic Local Alignment Search Tool,BLAST)时,采取的方式主要有如下 :
- 美国国立生物技术信息中心(National Center for Biotechnology Information ,NCBI)官网提供免费在线blast服务
- 搭建本地的blast环境。
- 使用云服务商提供的付费BLAST服务,如blast2go
当需比对碱基序列量少时,即不考虑时间的情况下,上述三种方式均可;但如果需要大量比对时,常规操作下三种方式各有利弊,第一种和第三种的共同点——慢;第二种的比对极大消耗本地服务器集群的算力。本文试图改良第一种方式提高效率
一.啰嗦一下在线BLAST的流程
1.首先进入比对网址:https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastx&PAGE_TYPE=BlastSearch&BLAST_SPEC=&LINK_LOC=blasttab&LAST_PAGE=blastn
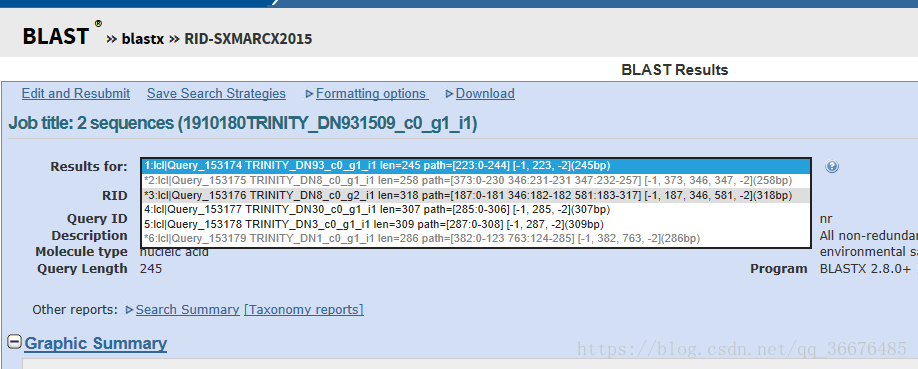
在线blast序列可采用两种上传方式,直接粘贴到输入框,或上传本地序列文件,如下图。
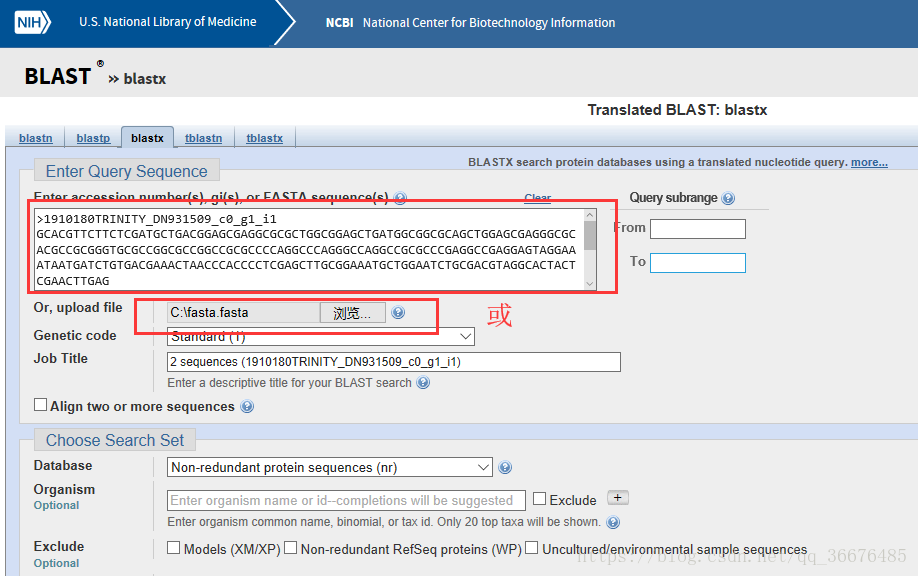
2.然后根据需要配置其他参数,点击下方的”BLAST” 按钮进入漫长的等待(主要时间为等待NCBI服务器处理提交的序列)如下图
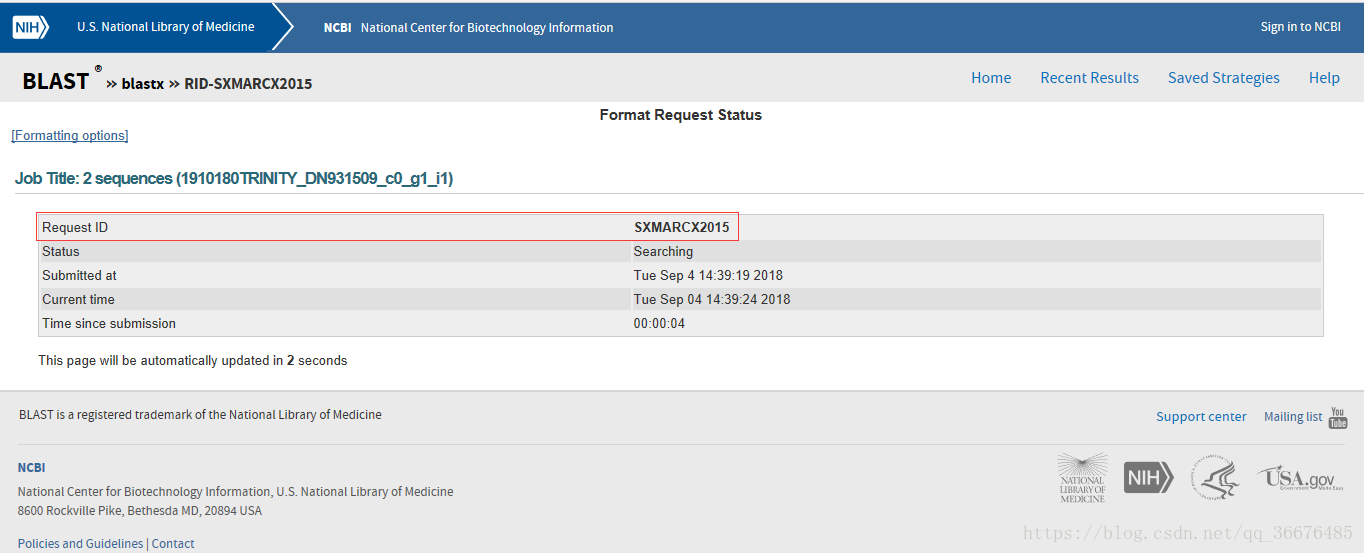
这个页面会自动刷新,当比对完成后刷新出的截面就是结果页面;请注意图中红框标出的”Request ID” 的值,后面要用到。
3.结果页面如下图
在多选框选定某特定碱基序列的选项,页面会自动跳转到该结果的页面(通过该页面定义为GetResults()的JavaScript函数),然后再根据需要再此页面索取信息。有时BLAST会匹配不到结果(这是序列自身与数据库中的碱基对没匹配上,请跟后面会提到的比对失败区别开),上图中,选项较浅且以 * 开头的为没有匹配到结果的序列,即这些序列的匹配结果为NULL或空。
二、分析
1.F12碱基序列提交后的等待页面发现页面在不断的发送一个POST包到指定地址,以询问是否出结果,如果出结果则跳转到结果页面(恕小弟才疏学浅没有发现POST包的构成规律)。
2.发现结果页面有GET方法可以访问https://blast.ncbi.nlm.nih.gov/Blast.cgi?CMD=Get&RID=***********;其中***********为上文提到的Request ID;也即是说,提交碱基序列过程和信息抓取过程可以分开,浏览器不必一直等待结果。可以提交序列,然后得到Request ID,另一个程序或设备用Request ID来抓取数据。
3.访问到结果页面后,通过不断选择下拉框的方式来获得同一批提交的不同的碱基序列的结果,而这些操作,使用python+selenium是方便的。
4.不断向NCBI服务器发起比对请求,并使用多线程的方式处理不同比对RID的结果,提高程序效率。
三、实际操作
环境:
windows 10+python 3.6.5(推荐搭配PyCharm)+phantomjs +Mysql
用到的python第三方扩展包:
- selenium
- pymysql
- openpyxl
预处理:
- Mysql中新建table2表存放碱基序列,结构为
| n | id | dna |
|---|
其中n为自增的主键,id为碱基序列的名称,dna为碱基序列。
2.Mysql中新建submit表存放Request ID,结构为
| RID | TTime | len |
|---|
其中TTime为碱基序列提交的时间,len为本次提交的总长度。
将待处理碱基序列导入表table2中。可以参考下列python代码
import pymysql
def sp(st):#分离序列的ID和序列b=st.split('\n')ID=b[0]DNAs=''for i in b:if b.index(i)==0:continueDNAs=DNAs+ireturn ID, DNAs
def DOSQLin(n,idL,DNAs):
#生成插入SQL语句id=idL.split(' ')ID=id[0]sql = "insert into table2 (n,id,dna) values (%s,'%s','%s');"%(n,ID,DNAs)return sql
f = open('C:\\*.fasta','r') #
All=f.read()
a=All.split('>')#每一条序列都是>开头的
del a[0]#删除第一个空元素
#链接数据库
conn = pymysql.connect(host='127.0.0.1', port=3306, user='root', passwd='luanchen',db='blast')
cur = conn.cursor()
n=1
for i in a:ID,DNA=sp(i)sql=DOSQLin(n, ID, DNA)try:cur.execute(sql)except:prio
cur.close()
conn.cmmit()#提交改变结果
conn.close()
四、提交碱基序列得到Request ID
过程分为下列步骤:
- 从mysql表中得到若干条待比对的碱基序列,再将这若干条从mysql表中删除;
- 将1中得到的序列生成一个本地文件(*.fasta) ;
- 通过selenium的方式打开在线BLAST网页,上传2得到的文件;
- 得到上传文件后的Request ID,并保存;
- 重复1~4直至mysql table2表中没有序列。
python代码(尽量加了很多注释,实际写得很乱…)
这篇关于python blast在线比对 每天最多数万条的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!