本文主要是介绍【论文笔记】RA-UNet: A Hybrid DeepAttention-Aware Network to ExtractLiver and Tumor in CT Scans,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
RA-UNet:用于在CT扫描中提取肝脏和肿瘤的一种混合深度注意力感知网络
论文地址:https://arxiv.org/abs/1811.01328
代码地址:https://github.com/RanSuLab/RAUNet-tumor-segmentation
目录
文章结构
问题动机
贡献
创新点
方法
网络总体结构
数据的预处理
RA-UNet 的结构
以U-Net作为基本结构
残差学习机制
注意力残差学习机制
损失函数
阶段一:使用 RA-UNet-I 进行肝脏定位
阶段二:使用 RA-UNet-II 进行肝脏分割
阶段三:基于RA-UNet-II的肿瘤提取
优化器
总结
文章结构
-
问题的背景与文章的贡献。
-
简要回顾了最新的自动化肝肿瘤分割方法。
-
详细介绍了这些方法,包括数据集、预处理策略、混合深度学习结构和训练过程。
-
对提出的方法进行了评估,报告了实验结果,并与其他一些方法进行了比较,并将方法扩展到了其他医学分割任务中。
-
给出了结论和下一步的工作。
问题动机
由于肝脏和肿瘤的异质和扩散形状,从CT图像中分割它们是具有挑战性的。
通常肝脏和肿瘤提取方法可以分为三类:手动分割、半自动分割和自动分割。手动分割是一种主观性强、可重复性差且耗时的方法。它在很大程度上依赖于人手工提取的可识别特征,并且需要具有高水平技术技能的人员来完成。半自动分割需要初始的人工干预,这可能导致偏差和错误。
为了加速和促进诊断、治疗计划和监测,并最终帮助外科医生切除肿瘤,有必要开发一种自动和精确的方法来从CT图像中分割肿瘤。
困难:大规模的空间和结构变化、肝脏与肿瘤区域之间的对比度低、噪声、部分体积效应、3D空间肿瘤特征的复杂性、附近器官之间的相似性。3D网络通常不像相应的2D网络那样有效和灵活,3D卷积的高计算成本和低效率,这使得2D网络扩展到3D网络是不切实际的。
为了解决这些问题,文章提出了一种混合残差注意力感知的肝脏和肿瘤提取神经网络RA-UNet,旨在以端到端的方式有效地从CT图像中提取肝脏和肿瘤的3D体积上下文特征。所提出的网络集成了残差U-Net架构和注意力残差学习机制,使得能够在深度网络上进行优化和性能改进。
贡献
-
通过堆叠注意力模块可以实现不同类型的注意力,从而使注意力感知特征可以自适应地变化。
-
使用 3D U-Net 作为基本架构来捕获多尺度注意力信息,并将低级特征与高级特征相结合。
-
RA-UNET可以直接从3D数据中分割肝脏和肿瘤。
-
模型不依赖于任何预先训练的模型或常用的后处理技术。
创新点
-
大多数最新的CNN架构,都使用来自3D图像的2D切片来执行分割任务。没有考虑器官的空间结构组织,没有充分利用体积信息。RA-UNet直接使用3D图像进行分割。
-
首次在医学影响分割任务中实现注意力残差机制
方法
网络总体结构
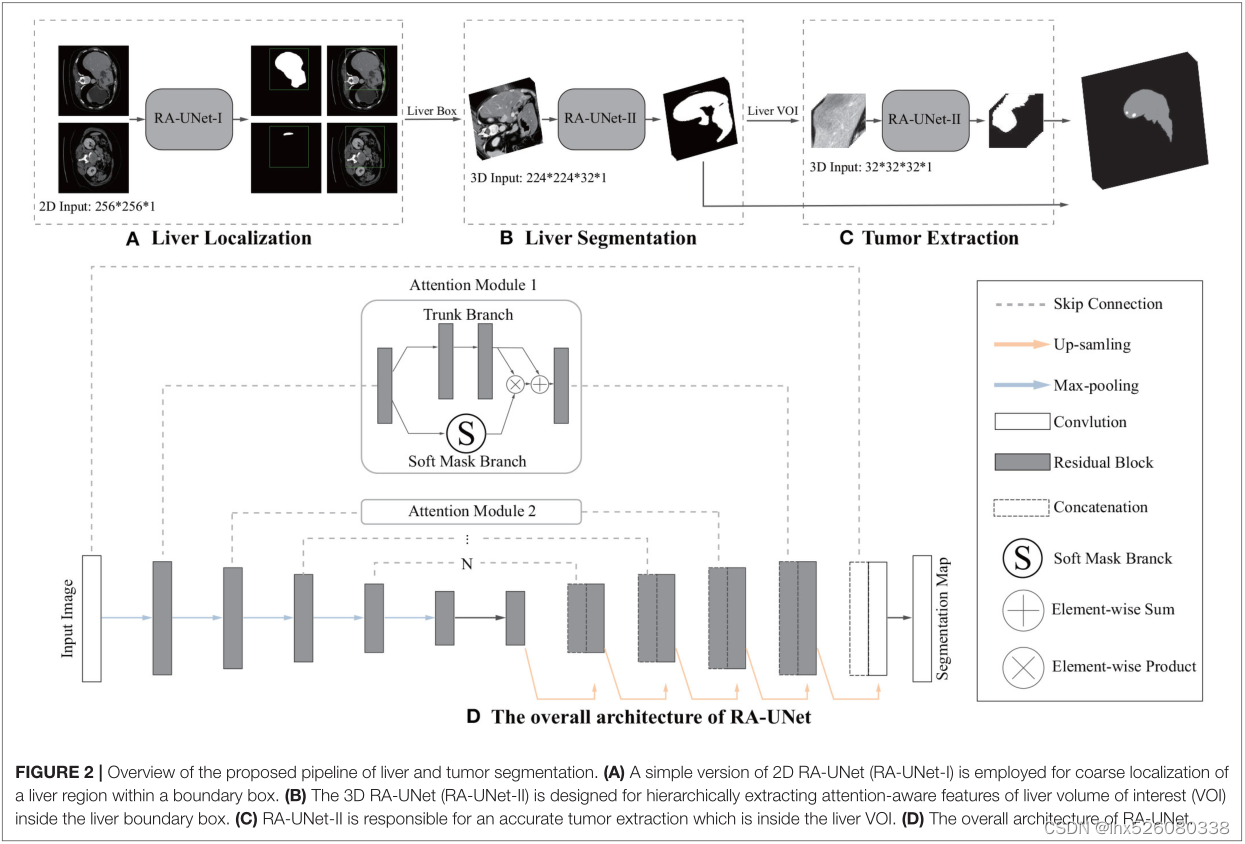
包括三个主要阶段,依次提取肝脏和肿瘤。
-
为了减少整体计算时间,使用名为RA-UNET-I的2D剩余注意力感知U-Net(RA-UNET-I)来获得粗略的肝脏边界框。
-
对3D RA-UNET进行训练,以获得精确的感兴趣区域(volume of interest , VOI)。
-
将得到的肝脏VOI送到第二个RA-UNET-II来提取肿瘤区域。
数据的预处理
HU值设置为-100~200,对加窗后的数据进行了零均值归一化和最小最大归一化。
RA-UNet 的结构
以U-Net作为基本结构
包含编码器和解码器两部分。编码器提取不同层次的特征,解码器接收具有不同复杂性的特征,并以从粗到细的方式重建特征。通过编码器部分和相应的译码部分引入了远程连接,使得编码器的不同层次特征可以合并到译码部分,从而使网络更加精确和可扩展。
残差学习机制
在反向传播中,残差网络的梯度是累加,避免了梯度消失的问题。
每个残差块由批归一化(BN)层、激活(RELU)层和卷积层组合而成。除了第一和最后一层(图2D)之外,其余的块被堆叠起来,以释放深度神经网络的能力。
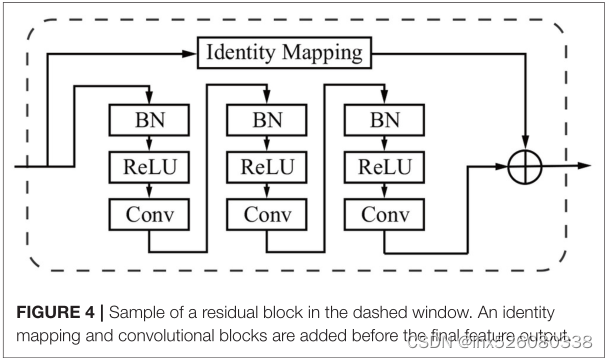
残差单元可以直接将特征从早期卷积传播到晚期卷积,从而提高模型的性能。
注意力残差学习机制
注意力残差机制将注意力模块分为主干分支和软掩码分支,主干分支用于处理原始特征,软掩码分支用于构造恒等映射。
软掩码分支中的编码器包含最大池操作、残差块和连接到相应解码器的远程残差块,其中在残差块和上采样操作之后执行逐元素求和。在软掩码的编码器和解码器部分之后,添加了两个卷积层和一个 Sigmoid 层以对输出进行归一化。
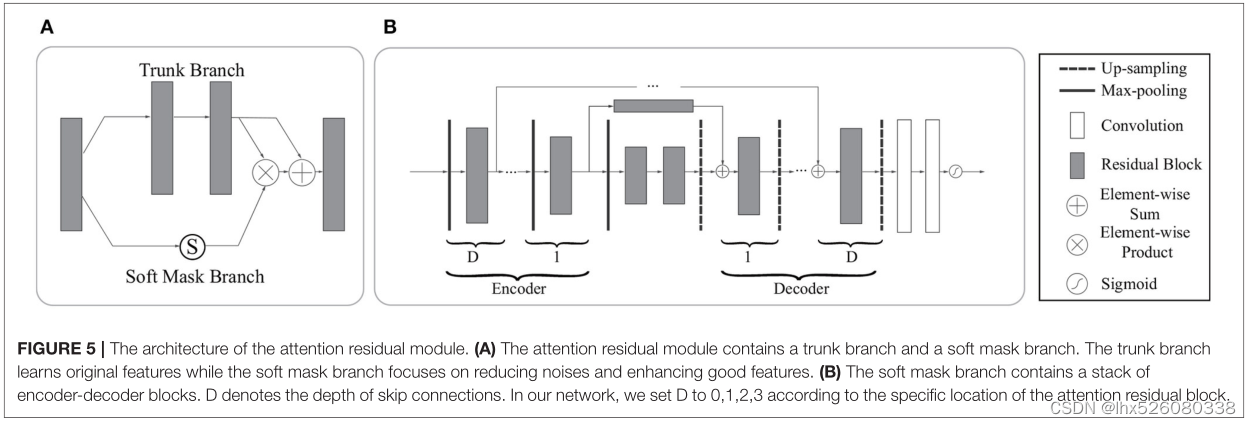
在注意力残留机制中,它旨在增强良好的特征并减少来自主干分支的噪声。
一般来说,注意力残差机制可以通过主干分支保留原始特征信息,通过软掩模分支关注那些肝肿瘤特征。
损失函数
采用Dice损失函数。
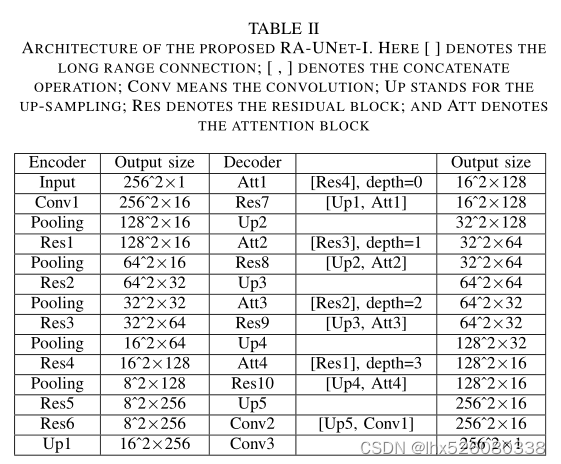
阶段一:使用 RA-UNet-I 进行肝脏定位
目的:降低通过 RA-UNet-II 精确分割肝脏和肿瘤的计算成本。
将切片下采样到256 * 256,使用2D版本的RA-UNet初步分割得到每个切片上的肝脏边界框,再使用3D连通域标记方法标记,取最大的区域,并在xyz轴方向上扩展了 10 个像素(为确保包括整个肝脏区域),获得肝脏的3D边界框,最后将图像插值到512 * 512。
RA-UNet-I网络结构:
阶段二:使用 RA-UNet-II 进行肝脏分割
将 x-y 平面中的肝脏边界框插值到 224×224,并在 z 方向连续随机选取 32 个切片以形成训练补图像块。在每个 CT 块上使用 RA-UNet-II 以按顺序生成 3D 肝脏概率块。然后,我们对这些概率块进行插值和堆叠,以恢复到边界框的原始大小。
RA-UNet-II网络结构:
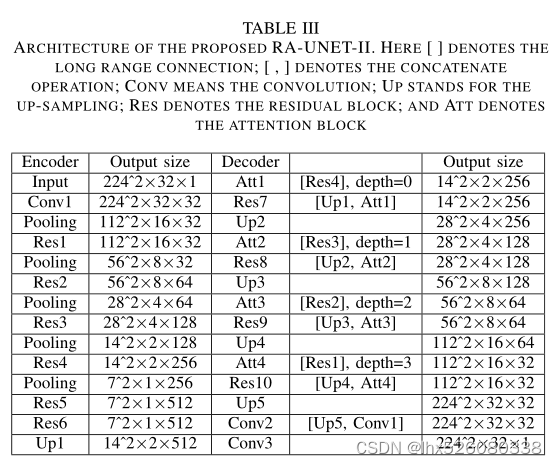
阶段三:基于RA-UNet-II的肿瘤提取
未进行插值和大小调整,以免丢失小病灶。为了确定patch size,将patch size分别设置为32×32×32、64×64×32和128×128×32来测试肿瘤分割的性能。结果表明,128×128×32 块大小的数据实现了最佳的肿瘤分割性能。在肿瘤以及周围的非肿瘤区域都选取图像块进行训练,以解决数据不平衡问题并学习更有效的肿瘤特征。在合并的 VOI 上使用投票策略以产生最终的肿瘤分割。最后,过滤掉不在肝脏区域的体素。
优化器
使用Adam优化器,初始LR为 0.001,β1=0.9,β2=0.999。
总结
文章提出一种名为RA-UNet的3D混合残差注意感知的分割方法,以精确提取感兴趣的肝脏体积(VOI)并从肝脏VOI中分割肿瘤。RA-UNet以3-Unet作为基本体系结构,然后结合不同层次的特征图提取上下文信息。利用残差和注意力相结合的分割网络-RA-UNet,包括三个阶段:使用RA-UNet-I 进行肝脏定位、使用RA-UNet-II进行肝脏的精确分割和 使用RA-UNet-II 分割肿瘤部分。堆叠注意力模块解决了梯度消失的问题,使得网络能够变得更深,通过残差学习使得注意力特征图自适应变化。得到的分割模型泛化能力较强,在另一个肿瘤分割数据集中也有很好的表现。
这篇关于【论文笔记】RA-UNet: A Hybrid DeepAttention-Aware Network to ExtractLiver and Tumor in CT Scans的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







