本文主要是介绍论文解读《TransUNet: Transformers Make Strong Encoders for Medical Image Segmentation》,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
论文解读《TransUNet: Transformers Make Strong Encoders for Medical Image Segmentation》
TransUNet:用于医学图像分割的变压器强编码器
发表期刊:CVPR2021
代码:代码链接
论文:论文链接
一、摘要:
在各种医学图像分割任务上,u形架构(也称为U-Net)已成为事实上的标准并取得了巨大的成功。但是,由于卷积运算的内在局限性,U-Net通常在显式建模远程依赖关系方面显示出局限性。专为序列到序列预测而设计的具有全局自我关注机制的Transformer已经成为替代架构,但是由于缺乏low-level的细节,其结果可能导致定位能力受限。在本文中提出的TransUNet同时具有Transformers和U-Net的优点,是医学图像分割的有力替代方案。transformer将CNN特征图编码为上下文序列,解码器对编码的特征上采样,再与高分辨率特征图结合实现精准定位
结合U-Net增强细节,可以将Transformers用作医疗图像分割任务的强大编码器。TransUNet在多器官分割和心脏分割等不同医学应用上与不同竞争方法相比取得了更优越的性能。
二、介绍
2.1
在U-Net的不同变体中,由具有(跳过连接)skip-connection的对称编解码器网络以增强细节保留,已成为事实上的选择。基于这种方法,在广泛的医疗应用中取得了巨大的成功,例如来自CMR的心脏分割和来自CT的器官分割等等。
Unet:一种典型的编码-解码结构,编码器部分利用池化层进行逐级下采样,解码器部分利用反卷积进行逐级上采样,原始输入图像中的空间信息与图像中的边缘信息会被逐渐恢复,由此,低分辨率的特征图最终会被映射为像素级的分割结果图。而为了进一步弥补编码阶段下采样丢失的信息,在网络的编码器与解码器之间,U-Net算法利用Concat拼接层来融合两个过程中对应位置上的特征图,使得解码器在进行上采样时能够获取到更多的高分辨率信息,进而更完善地恢复原始图像中的细节信息,提高分割精度。
而增加了跳过连接(skip connection)结构的U-Net,网络能够保留更多高层特征图蕴含的高分辨率细节信息,从而提高了图像分割精度。
(跳过连接)skip-connection:通常用于残差网络中,
它的作用是:在比较深的网络中,解决在训练的过程中梯度爆炸和梯度消失问题。采用skip-connection的好处是,把对应尺度上的特征信息引入到上采样或反卷积过程,为后期图像分割提供多尺度多层次的信息,由此可以得到更精细的分割效果。
探讨:
单一地使用Transformer对标记好的图像块进行编码,直接将隐藏的特征用上采样为完整分辨率进行输出,无法产生令人满意的结果。
本文提出TransUNet,Transformer采用自注意力self-attention机制将来自卷积神经网络(CNN)特征图的标记化图像块编码为提取全局上下文的输入序列。然后为了弥补Transformers特征解析(encoder)带来的损失,TransUNet采用混合CNN-Transformer架构,解码器对编码的特征上采样然后与从编码路径中跳过的不同高分辨率CNN特征图组合,以实现精确定位。实验结果表明基于transformer的比先前CNN的医学图像分割效果要好。

注:CNN特别是全卷积神经在医学图像分割领域主导地位,在一些变体中,像UNet用跳过连接(skip-connection)方式的网络增强了细节上特征的保留,成功应用在医学图像处理领域。但是在质地形状等特征上的保留仍有局限性,因此,基于CNN特征提出self-attention机制,完全免除卷积运算符,完全依靠注意力机制,此前广泛应用于NLP,最近应用在图像识别上也颇有成效。
三、 方法
3.1 图像序列化
论文目标是预测相应像素大小为H*W的标签图。最常见的方法:CNN(例如U-Net)。
注:一般操作就是CNN编码成高维特征图,再解码成完整的空间分辨率,而我们的方法使用transformer将self-attention机制加入编码阶段。
首先通过将输入x属于R(H X W X C),给定图像其空间分辨率为HxW,通道数为C。用PXP大小的切片去分割图片可以得到N个切片(N= HW/P^2 是图像切片的数量,即输入序列长度),那么每个切片的尺寸就是PXPXC,形成二维的序列,转化为向量,将N个切片重组后向量连接就可以得到NXPXPXC(总的输入变换)的二维矩阵。
3.2 (切片嵌入法)Patch Embedding
Patch Embedding:
HW/(PP)<H*W,因此采用混合CNN的transformer以及级联上采样(a cascaded upsampler)策略,以能获得精准定位。
(1)CNN-Transformer编码器以及级联上采样
作者发现混合CNN-Transformer编码器比简单地使用Transformer作为编码器表现更好。
将切片Xp通过线性投影( linear projection)映射到D维的嵌入空间,切片嵌入表示如下面公式所示(1):

注:E是切片嵌入投影,Epos是position embedding。transformer层包含L层Multihead Self-Attention(MSA)和Multi-Layer Perceptron (MLP)模块
变压器编码器由L层多头自注意(MSA)和多层感知器(MLP)块(等式)组成(2)(3))

级联上采样:(CUP)
(CUP)包含多个上采样层,提取特征生成特征图,用CUP解码输出隐藏特征,在隐藏层重新修改尺寸之后,再联级多个上采样块实例化CUP(每个块都包含两个上采样运算,一个33卷积层,一个ReLU激活层)将整个分辨率HW/(PP) 的转化成HW。其中上采样保留了skip-connection(跳跃连接),使得能以不同的分辨率级别进行特征聚合。
嵌入的结构示意图:
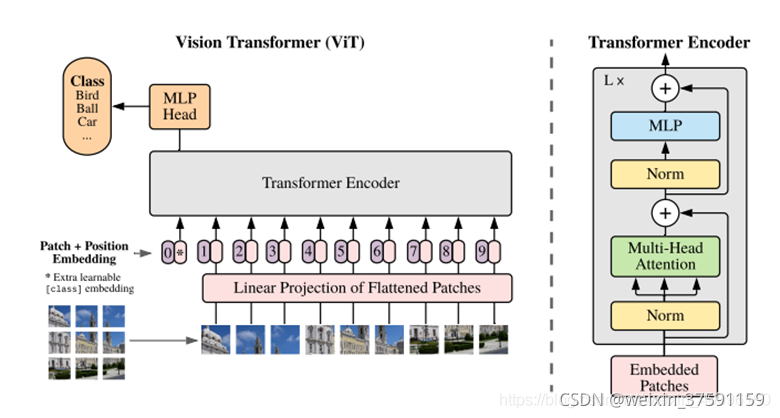
(1)将图像转化为序列化数据
如下图所示。首先将图像切片,每个切片重整成一个向量,得到铺平切片。
如果图片是HWC 维的,用PP大小的切片去分割图片可以得到 N个切片,那么每个切片的尺寸就是 PPC,转化为向量后就是 P^2C维的向量,将N 个切片重组后的向量连在一起就得到了一个 N*(PPC) 的二维矩阵。

(2)位置嵌入

(3)学习性嵌入(Learnable embedding)

这个是一个学习型嵌入(记作 Xclass ),其作用类似于BERT中的[class] token。在BERT中,[class]编码经过编码后对应的结果作为整个句子的表示;类似地,这里 Xclass经过编码后对应的结果也作为整个图的表示。
(4)Transformer 编码
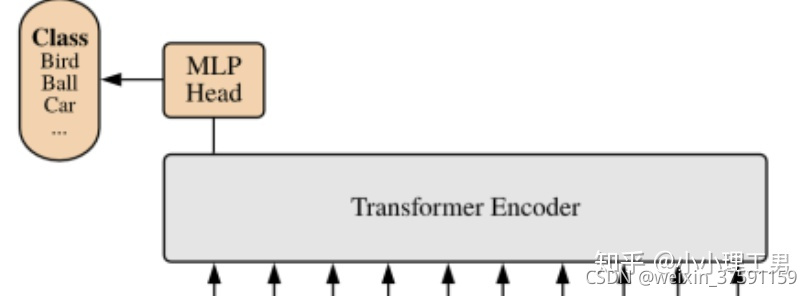
Transformer Encoder的结构如下图所示:

对于Encoder的第 L 层,记其输入为Zl-1 ,输出为 Zl,则计算过程为:

四、TransUNet总体结构
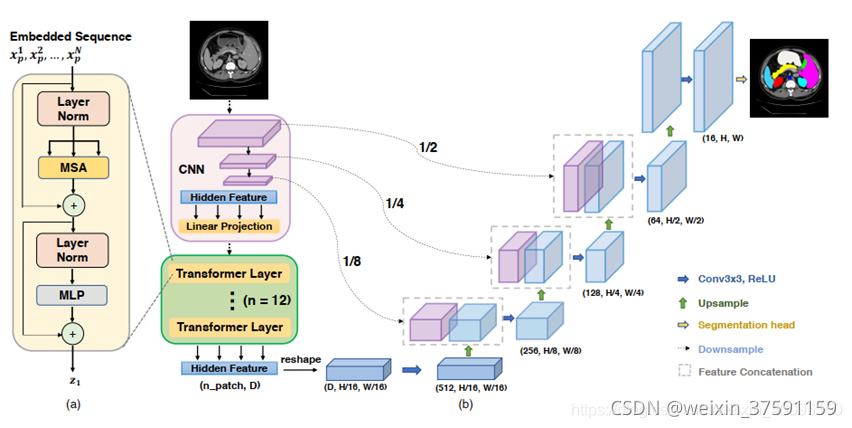
解码器:就是常规的转置卷积上采样恢复图像像素。同时从编码器的CNN下采样对应过来同层分辨率的级联。
级联在计算机科学里指多个对象之间的映射关系,建立数据之间的级联关系提高管理效率。
五、对比实验
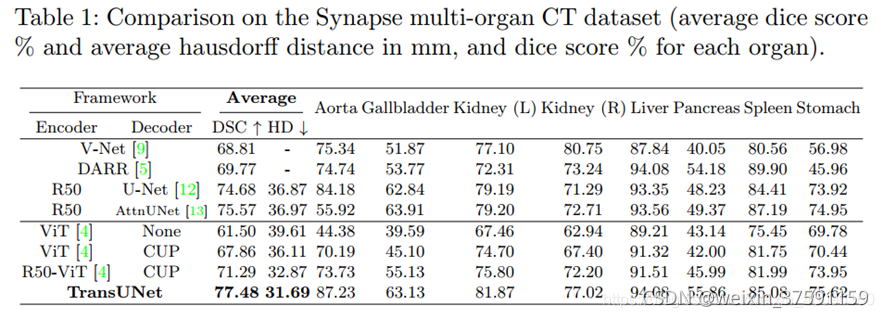
医疗图像分割评价标准:Dice系数(主要是用来计算两个集合的相似性的(也可以度量字符串的相似性))和豪斯多夫距离(描述两组点集之间相似程度的一种量度)
Aorta:主动脉;gallbladder:胆囊 ;kidney:肾 ;liver:肝脏;pancreas:胰腺;spleen:脾脏;stomach:胃
六、网络模型主要应用及结果
6.1 实验中使用的分割数据集
①Synapse multi-organ segmentation dataset(Synapse多器官分割数据集)
腹部CT扫描 (30次腹部CT扫描 总共有3779张轴向增强腹部临床CT图像)
每个CT体由512 *512像素的85198切片组成,体素空间分辨率([0.540.54]×[0.980.98]×[2.55.0]) mm^3。报告了8个腹部器官(主动脉、胆囊、脾、左肾、右肾、肝、胰腺、脾、胃)的平均Dice和平均豪斯多夫距离(HD),随机分为18个训练病例(2212个轴向切片)和12个验证病例。
②Automated cardiac diagnosis challenge
心脏CMR(心脏核磁)
一系列短轴切片从左心室底部到顶部覆盖心脏,切片厚度为5至8毫米。短轴平面内空间分辨率从0.83到1.75 mm^2/pixel。每个患者扫描都用手工标注了左心室(LV)、右心室(RV)和心肌(MYO)。报告了平均Dice,随机分为70个训练病例(1930个轴向切片),10个用于验证,20个用于测试。
path="E:/lhf\TransUNet-main\project_TransUNet\project_TransUNet\data\Synapse/train_npz/case0005_slice050.npz"
data=np.load(path)
x_train=data["image"]*255
la_train=data["label"]*255
plt.subplot(121)
plt.imshow(x_train)
plt.subplot(122)
plt.imshow(la_train)
plt.show()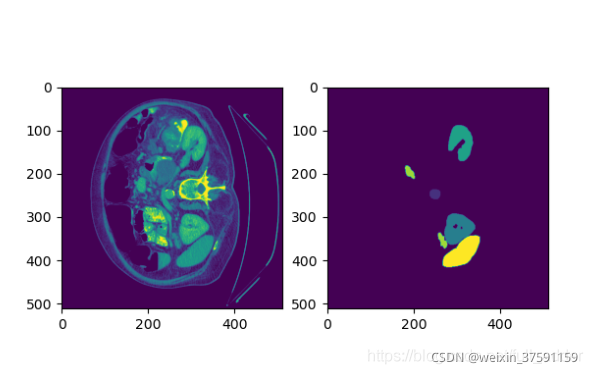
6.2 主要结果
作者分别在Synapse多器官分割数据集和ACDC (自动化心脏诊断挑战赛)上实验了TransUNet的效果。具体地,对于混合编码器,论文中使用ResNet-50和ViT分别作为CNN和Transformer的backbone,并且都经过了ImageNet的预训练处理。
在Synapse多器官CT数据集(腹部)上的比较(平均dice分数%和平均豪斯多夫距离,单位为毫米,以及每个器官dice分数%)
表1是TransUNet与VNet等模型的效果对比

6.3 视觉比较

七、 结论
TransUNet是率先将Transformer结构用于医学图像分割工作的研究。TransUNet将重视全局信息的Transformer结构和底层图像特征的CNN一起进行混合编码,能够更大程度上提升UNet的分割效果。
Transformer是一种天生具有强大自我注意机制的结构。在这篇论文中,作者研究Transformer在一般医学图像分割中的应用。为了充分利用Transformer的力量,提出了TransUNet,它不仅将图像特征作为序列来编码强全局上下文,还通过Unet混合网络设计来很好地利用低层CNN特征。TransUNet可作为一种替代框架用于医学图像分割,其性能优于各种竞争方法,包括基于cnn的自我注意方法。
本文为了完整的应用transformer,提出了TransUNet, 不仅通过将图像以序列处理编码全局上下文信息,也通过使用U型结构将低层次CNN特征利用上,作为基于FCN的主流医学图像分割方法的替代框架,在医学图像分割上(包括多器官分割和心脏分割)上均比各种竞争方法(像基于CNN的自注意方法)具有更优的表现。
这篇关于论文解读《TransUNet: Transformers Make Strong Encoders for Medical Image Segmentation》的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!











