本文主要是介绍群体遗传学基础学习,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
Introduction
群体遗传学(Population Genetics)是研究在群体水平上基因频率的变化及其影响因素的学科。它结合了遗传学、进化生物学和统计学的方法来分析基因在群体中的分布和变化。
群体(Population)指的是一组同一物种的个体,它们生活在同一地理区域内,并能够相互交配和繁殖。这个群体的个体共享一个共同的基因库(gene pool),即所有个体的基因及其等位基因的集合。
群体有几个特征:
-
物种一致性:群体中的所有个体属于同一个物种,意味着它们在生物学上能够相互交配并产生具有生殖能力的后代。
-
地理相对性:群体通常指在特定地理区域内的个体集合,虽然不同群体之间可能会有基因交流(如迁移),但通常被认为是相对隔离的。
-
基因库:群体内的个体共享一个基因库,这意味着在群体内部进行的所有交配,都会影响该群体的基因频率和遗传结构。
-
交配能力:群体中的个体能够进行随机或非随机的交配,产生后代,从而使得群体的基因频率在世代之间发生变化。
在群体遗传学研究中,群体的概念是基础,因为研究的主要目标是理解在不同条件下(如自然选择、基因漂变、基因流动、突变等),一个群体的基因频率和基因型频率是如何变化的。
群体遗传学的研究帮助科学家理解进化过程、物种形成、群体间的遗传差异及其对环境变化的适应能力等。
Concepts
以下是群体遗传学中一些相关概念的简要介绍:
-
Locus(基因座):指基因或DNA片段在染色体上的特定位置。基因座可以是一个特定基因的位置,也可以是任意DNA序列的位置。
-
Variation/Mutation(变异/突变):指基因或DNA序列中发生的变化。突变是引起变异的过程,是基因组中遗传信息的永久性改变。
-
Indel(插入/缺失):指DNA序列中发生的短片段插入或缺失变异。Indel可导致基因功能的变化,尤其是在编码区发生时。
-
SNP(单核苷酸多态性):指基因组中单个核苷酸的位置上的变异,是一种常见的遗传变异形式。
-
Alleles(等位基因):指位于同一基因座的不同版本或变体。一个基因座可以有多个等位基因。
-
SV(结构变异,Structural Variation):指基因组中涉及大片段DNA的变异,通常定义为长度大于50个碱基对的变异。SV包括多种类型的变异,如:
- 缺失(Deletion):基因组中一段DNA片段的丢失。
- 重复(Duplication):一段DNA片段的重复。
- 倒位(Inversion):一段DNA片段在基因组中的方向发生了反转。
- 易位(Translocation):一段DNA片段从一个位置移动到另一个位置。
- 插入(Insertion):一段新的DNA片段插入到基因组中。
-
CNV(拷贝数变异,Copy Number Variation):属于结构变异的一种,指基因组中某段DNA序列的拷贝数发生了变化,导致这段序列的拷贝数多于或少于通常的两个拷贝。CNV可以包括:
- 基因扩增(Gene Amplification):某段DNA的拷贝数增加。
- 基因缺失(Gene Deletion):某段DNA的拷贝数减少。
-
Haplotypes(单倍型):指一组位于同一染色体上的紧密连锁的基因或遗传标记的组合,通常作为一个整体进行遗传。
-
Selection(选择):自然选择的过程,通过优先传递适应环境的基因,影响群体中基因频率的变化。
- Positive selection(正选择):有利于有益突变的传播和保持。
- Negative selection(负选择):排除有害突变,维持群体基因组的稳定性。
-
Genetic drift(基因漂变):群体中的等位基因频率由于随机抽样效应而发生的变化,尤其在小群体中更为显著。
-
核苷酸多样性 (π) :测量群体中任意两个个体的基因序列之间平均差异的程度。反映了群体的遗传多样性。
-
非同义突变率与同义突变率的比值 (dN/dS) : 非同义突变率(dN)是引起氨基酸序列变化的突变率,同义突变率(dS)是不引起氨基酸序列变化的突变率。dN/dS比值用于判断是否存在选择压力。
- dN/dS = 1:指非同义替代和同义替代发生的概率相等,通常表示该基因区域处于中性进化状态,即没有选择压力,仅靠随机突变率演化。
- dN/dS < 1:指非同义替代的发生率低于同义替代,通常表示负选择(或净化选择,purifying selection)。在这种情况下,非同义突变可能对生物体不利或有害,因此被自然选择淘汰。
- dN/dS > 1:指非同义替代的发生率高于同义替代,通常表示正选择(positive selection)。在这种情况下,非同义突变可能带来有利的适应性优势,因此被自然选择保留下来。
-
LD(连锁不平衡,Linkage Disequilibrium):指在一个群体中,两个或多个位点上的等位基因频率之间存在统计学上的非随机关联。换句话说,在一个群体中,某些等位基因组合比随机预期的频率出现得更多或更少。
- 完全连锁平衡(LD = 0):两个等位基因之间的组合是随机的,意味着它们独立遗传。
- 完全连锁不平衡(LD = 1):两个等位基因总是一起遗传,意味着它们之间存在强烈的关联。
-
Tajima’s D:是一个统计量,用于检测群体中核苷酸多样性和序列分化的异常,进而判断是否存在自然选择或其他进化力(如人口扩张、收缩等)。
- Tajima’s D = 0:表示核苷酸多样性和序列分化之间的一致性,通常意味着该群体处于中性进化状态,即没有自然选择作用。
- Tajima’s D > 0:表示观测到的核苷酸多样性较高,序列间的差异较少,可能表明存在平衡选择或群体结构(如两个或多个亚群体的混合)。
- Tajima’s D < 0:表示观测到的核苷酸多样性较低,序列间的差异较大,可能表明存在正选择(有利突变的快速扩散)或人口扩张。
-
D’(D-系数):衡量两个遗传位点间连锁不平衡(LD)程度的指标,取值范围从0到1。D’ = 1 表示完全连锁,D’ = 0 表示无连锁。
-
重组率 (1-D’) : 重组率用于衡量两个位点之间的重组频率。1-D’表示在连锁不平衡(LD)中,反映了重组事件的相对频率。
-
Divergence(分歧):描述不同种群或物种之间的基因差异程度,反映了进化过程中累积的遗传变化。
-
Molecular clock(分子钟):假设特定DNA序列的突变速率恒定,用于估计不同物种或种群间分歧时间的工具。
-
Founder effect(奠基者效应):小群体从大群体中分离出来形成新的群体时,因最初几个创始者的基因组成差异而导致的新群体基因频率变化。
-
Bottleneck effect(瓶颈效应):指群体经历了急剧的数量减少后,存活个体的基因组成对整个群体基因频率产生显著影响,通常伴随遗传多样性的减少。
Hardy-Weinberg定律
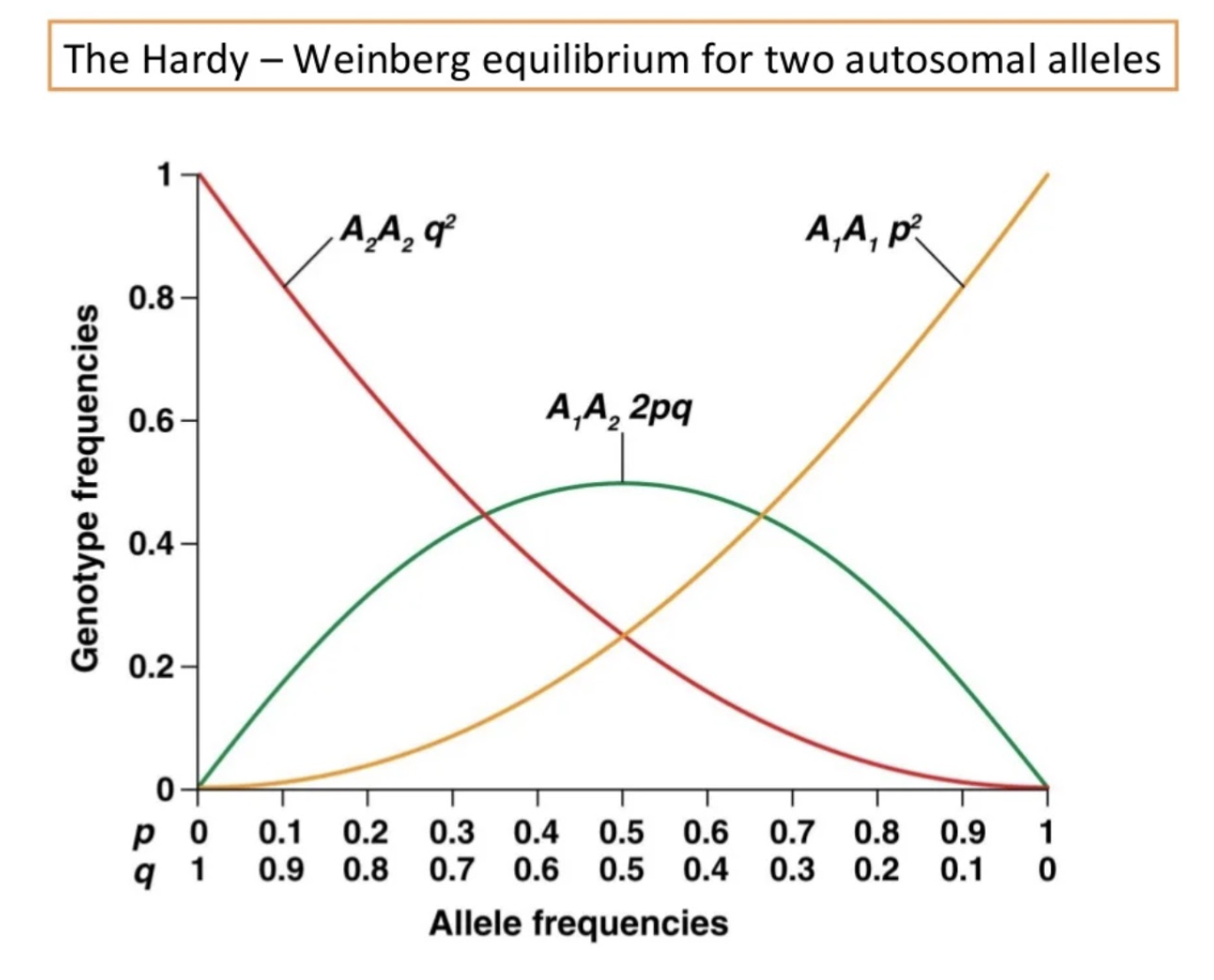
Hardy-Weinberg定律(Hardy-Weinberg Law),又称为哈迪-温伯格平衡定律,是群体遗传学中的一个基本原则。它描述了在一个理想的随机交配的群体中,等位基因和基因型的频率在世代之间保持恒定的状态。这一定律是由英国数学家G.H. Hardy和德国医生Wilhelm Weinberg在1908年独立提出的。
- Hardy-Weinberg定律的条件
Hardy-Weinberg定律成立的前提条件是:
- 无突变:没有新的等位基因通过突变加入到群体中。
- 无自然选择:所有基因型的生存和繁殖成功率相等,没有选择压力。
- 无限大的群体:群体的大小足够大,以至于基因频率的变化不受随机基因漂变的影响。
- 随机交配:群体中的个体是随机交配的,没有任何形式的非随机交配(如近亲交配或择偶偏好)。
- 无基因流:没有来自其他群体的基因引入,也没有个体迁出群体。
- Hardy-Weinberg定律的数学表达
假设在一个二倍体群体中,一个基因座有两个等位基因:A和a。我们定义:
- p:等位基因A的频率(取值范围为0到1)。
- q:等位基因a的频率(取值范围为0到1)。
根据等位基因频率的定义,有 p + q = 1 p + q = 1 p+q=1。
根据Hardy-Weinberg定律,基因型频率(AA、Aa和aa)的平衡状态可以用等位基因频率来表示:
- AA的频率: p 2 p^2 p2
- Aa的频率: 2 p q 2pq 2pq
- aa的频率: q 2 q^2 q2
这些频率之和也等于1:
$ p^2 + 2pq + q^2 = 1 $
- Hardy-Weinberg平衡的应用
-
检测选择压力:通过比较观测到的基因型频率和预期的Hardy-Weinberg平衡频率,可以检测是否存在自然选择或其他进化因素的作用。
-
估算等位基因频率:利用基因型频率来估算等位基因频率,尤其在研究隐性遗传病的频率时。
-
基因库的稳定性:在一定条件下,如果一个群体的基因库符合Hardy-Weinberg平衡,则可以认为该群体的基因库在无外部干扰情况下是稳定的。
- 实际应用中的偏离
在现实中,许多群体不完全符合Hardy-Weinberg平衡条件,可能因为:
- 自然选择:不同基因型具有不同的适应性。
- 小群体效应:基因漂变在小群体中更为显著。
- 非随机交配:如近亲交配或选择性交配。
- 基因流:来自其他群体的基因引入。
偏离Hardy-Weinberg平衡可以揭示关于群体演化过程的有价值的信息。
VCF
VCF(Variant Call Format 是一种用于存储和描述基因组变异(如SNP、插入、缺失等)的文本文件格式。VCF文件广泛用于基因组学研究,尤其是在分析和存储从基因组测序数据中识别出的变异时。
VCF文件由**头部(Header)和数据区(Data section)**两部分组成。
- 头部(Header)
头部部分以“#”开头,包含文件的元信息和描述变异的相关注释。常见的头部信息包括:
-
文件格式版本:例如,
##fileformat=VCFv4.2。 -
样本信息:例如,
##SAMPLE=<ID=Sample1,Description="Sample description">。 -
参考基因组信息:例如,
##reference=ftp://ftp.1000genomes.ebi.ac.uk/...。 -
信息字段的描述:例如,
##INFO=<ID=DP,Number=1,Type=Integer,Description="Total Depth">,描述变异的附加信息。 -
格式字段的描述:例如,
##FORMAT=<ID=GT,Number=1,Type=String,Description="Genotype">,描述样本格式信息。
- 数据区(Data section)
数据区包含实际的变异信息,每行表示一个变异。数据区以一行以“#CHROM”开头的字段名称作为标题行,常见的字段有:
-
CHROM:染色体号,表示变异所在的染色体。
-
POS:位置,表示变异在染色体上的位置(1-based)。
-
ID:变异的标识符(若无信息则为“
.”)。 -
REF:参考碱基或参考序列,表示参考基因组中该位置的碱基。
-
ALT:替代碱基或替代序列,表示变异后的碱基或序列。
-
QUAL:变异的质量得分,通常为Phred质量分数。
-
FILTER:变异的过滤信息,描述该变异是否通过了特定的质量过滤标准(如“PASS”表示通过)。
-
INFO:附加信息字段,包含与变异相关的各类注释和统计信息。
-
FORMAT:样本格式字段,描述以下各列样本数据的格式。
-
样本数据:每个样本的变异信息,根据FORMAT字段中的描述显示具体数据,如基因型、覆盖度等。
-
例子
下面是一个简单的VCF文件示例:
##fileformat=VCFv4.2
##source=Example
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT Sample1
1 123456 rs123456 G A 50 PASS DP=100;AF=0.5 GT:DP 0/1:35
1 789101 . T C,G 99 PASS DP=200;AF=0.25,0.75 GT:DP:AD 1/2:60:10,20
在这个例子中:
-
第一个变异在1号染色体的123456位点,参考碱基是G,替代为A,质量得分50,通过了过滤,附加信息显示总深度(DP)为100,等位基因频率(AF)为0.5,样本1的基因型(GT)是0/1,覆盖度(DP)是35。
-
第二个变异在1号染色体的789101位点,参考碱基是T,替代为C和G(多等位基因变异),质量得分99,通过了过滤,附加信息显示总深度(DP)为200,等位基因频率(AF)分别为0.25和0.75,样本1的基因型(GT)是1/2,覆盖度(DP)是60,等位基因深度(AD)分别为10和20。
-
使用场景
VCF文件主要用于: - 基因组变异分析:存储和分析个体或群体的基因变异。 - 临床基因组学:用于个体的基因组测序数据分析,帮助检测可能的致病突变。 - 进化研究:比较不同物种或个体之间的基因组差异。 - 群体遗传学:研究群体中等位基因的分布和频率。
VCF格式标准化且易于扩展,适合存储大规模基因组数据,并支持丰富的注释信息,是基因组学研究中的重要工具。

关注公众号 ‘bio llbug’,获取最新推送。
这篇关于群体遗传学基础学习的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!