本文主要是介绍Phybers:脑纤维束分析软件包,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
摘要
本研究提供了一个用于分析脑纤维束数据的Python库(Phybers)。纤维束数据集包含由表示主要白质通路的3D点组成的流线(也称为纤维束)。目前已经提出了一些算法来分析这些数据,包括聚类、分割和可视化方法。由于流线的几何复杂性、文件格式和数据集的大小(可能包含数百万条纤维束),对纤维束数据的处理并不简单。因此,本研究收集和整理了最新的脑纤维束分析方法,并将其集成到Python库中,以整合和共享脑纤维束示踪分析工具。由于计算要求较高,最具挑战性的模块需要用C/c++实现。可用的功能包括纤维束分割(FiberSeg)、分层纤维聚类(HClust)、快速纤维聚类(FFClust)、归一化到参考坐标系、纤维采样、计算大脑纤维集合之间的交点、聚类过滤工具、从聚类中计算测量值以及纤维束可视化。该库分为四个主要模块:分割(Segmentation)、聚类(Clustering)、实用工具(Utils)和可视化(Fibervis)。Phybers可以在GitHub上免费获得,并提供了样本数据和大量文档。此外,该库可以通过pip库轻松地在Windows和Ubuntu操作系统上安装。
引言
大脑结构连通性可以通过扩散磁共振成像(dMRI)进行研究。这是一种无创的在体技术,通过测量脑组织中水分子的运动,提供关于脑白质(WM)的微观尺度信息。利用dMRI数据上的扩散局部模型重建和纤维束示踪算法,可以计算出3D WM束的主要轨迹。为了简便起见,这些流线也被称为“纤维束”,尽管它们并不代表单个轴突。
多年来,随着MRI设备和重建以及纤维束成像算法的改进,用于分析纤维束成像数据集的工具也在不断发展。如今,大脑纤维束数据相当复杂,包含长短不一的纤维,以及噪声和复杂的几何结构。此外,这些数据集可能包含数百万条纤维用于概率性纤维追踪,从而产生额外的计算需求,尤其是在执行多被试分析时。这就是为什么有许多示踪数据分析算法旨在对这些数据进行聚类、识别模式、分割、过滤、可视化和计算测量值的原因。由于示踪数据具有复杂性,这些算法通常难以使用,并且需要对文件格式、输入参数和结果有深入的了解。因此,为了简化和推广dMRI的使用,一些研究小组创建了用于处理dMRI图像的软件包。这些工具提供了dMRI数据处理各步骤所需的算法,从图像失真校正到示踪分析。最终目标是开发出一套处理纤维束示踪数据的方法,以更好地描述基于高质量数据的WM纤维,并研究健康被试和病理性大脑的WM微结构。
可用于处理dMRI数据的工具有许多。表1、2总结了影像学研究社区常用的软件包。表中列出了这些工具的主要特性和功能,如编程语言、操作系统(OS)、软件许可证、dMRI格式、纤维追踪格式、扩散加权(DW)模型重建、纤维追踪、纤维聚类、纤维束分割、可视化以及纤维测量计算等。主要软件包括:BrainSUITE、Camino、Diffusion toolkit、ExploreDTI、FSL、MRtrix、Freesurfer、DSI Studio、Dipy、DiffusionKit和SlicerDMRI。
表1.dMRIs研究使用的主要软件。
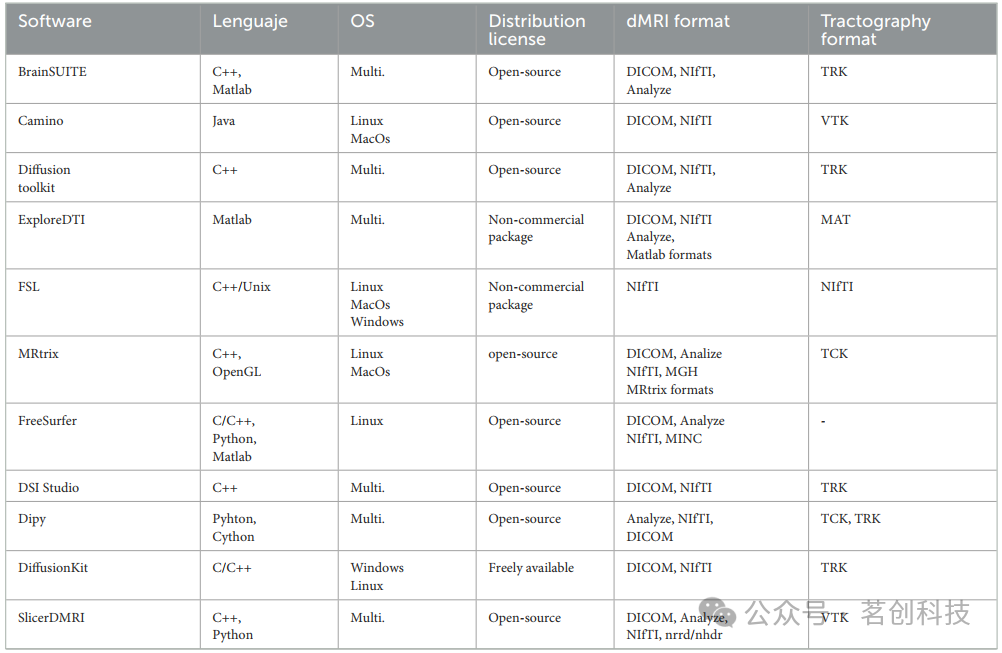
表2.dMRIs研究使用的主要软件。
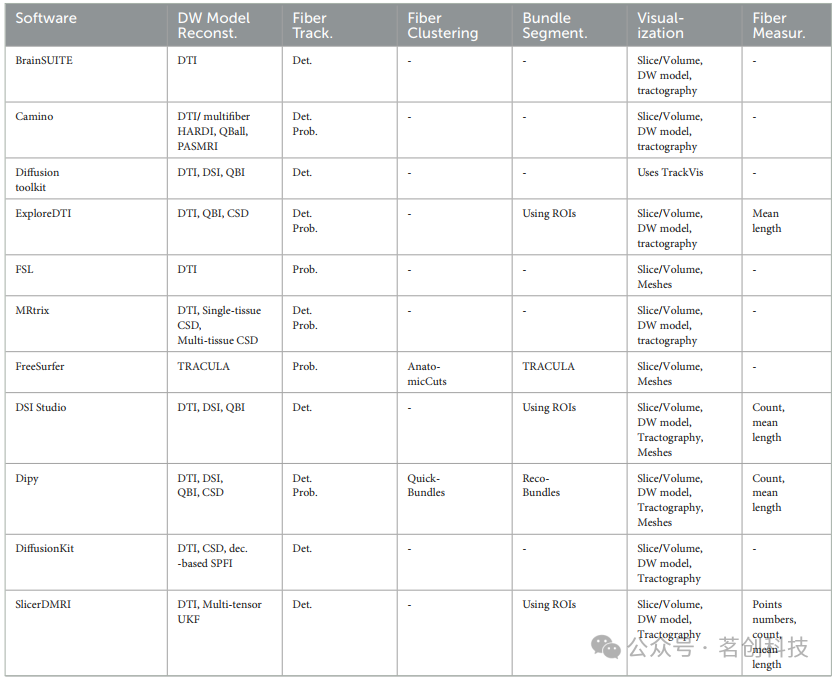
如表1、2所示,这些工具包具有不同的功能。其中一些更侧重于dMRI预处理、模型重建和纤维束成像,而其他一些则包括示踪分析方法。用户通常会使用多个软件来实现其处理流程,但需要注意文件格式、参考坐标系和三维空间。尽管有一些较为全面的工具包,但没有一个工具包涵盖所有现有的算法。
目前很少有专门用于分析示踪数据的软件包,例如纤维聚类和分割,以及纤维束过滤。因此,本研究提出了一种用于分析大脑纤维示踪数据的工具包。该工具包结合了多种纤维示踪分析工具(由本研究小组开发),其中包括使用脑纤维图谱优化的纤维束分割算法,基于K-Means的分层纤维聚类和快速纤维聚类(FFClust)算法。由于缺乏统一的代码、编程语言的多样性、多个库依赖以及缺乏示例代码/数据和文档,这些工具对外部用户而言难以应用。为此,本研究开发了一个名为Phybers的开源库,它集成了所有这些算法以及其他纤维聚类分析和可视化工具。
Phybers
Phybers包含四个模块,其中包括用于不同预处理阶段的算法。Utils套件包含用于纤维分割或聚类之前对纤维示踪数据进行预处理的工具,例如使用变形场(NIfTI格式)将纤维(bundles格式)转换到另一个空间。分析模块包括基于脑纤维束图谱的纤维束分割算法,以及快速纤维聚类(FFClust)和分层聚类(HClust)两种聚类算法。此外,还提供了一套用于分析纤维束分割和纤维聚类算法结果(bundles格式)的后处理工具。可视化模块支持不同类型的数据,例如体积(NIfTI)、网格(mesh和GIfTI格式)、纤维束(TRK、TCK和bundles)。此外,它集成了一个交互式图形用户界面(GUI),允许用户实时操纵3D对象。例如,可以通过定位两个或多个3D区域来执行脑纤维束的手动分割。
Phybers是基于Python开发的,以便在PyPI存储库中发布和更新。本研究使用C/C++和Python 3.9实现了这些算法,其中使用了numpy、nibabel、pandas和subprocess等Python依赖项。然而,所有依赖项都会随着软件包的安装而自动安装。可以使用命令$ pip install phybers来执行库安装,并且该软件发行版包含所有支持功能的示例数据和代码示例。Phybers兼容Python 3.9以上的版本,并支持Jupyter Notebook和Spyder等Python平台,为满足每个用户的特定需求提供了强大的灵活性。此外,它可以在Ubuntu和Windows系统上无缝运行,也可以通过虚拟机在macOS上使用。最后,使用Sphinx生成库文档。Phybers库包含四个模块(图1),分别定义为Segmentation、Clustering、Utils和Visualization。

图1.四个模块:(A)分割,(B)聚类,(C)实用工具和(D)可视化。
Phybers层次结构
分割模块
该模块包括基于多被试图谱的白质纤维束分割算法(图2)。该方法使用相应点之间的最大欧氏距离(dME)作为纤维对之间相似性的度量,定义为:
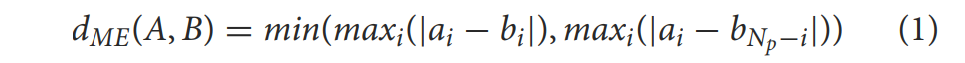
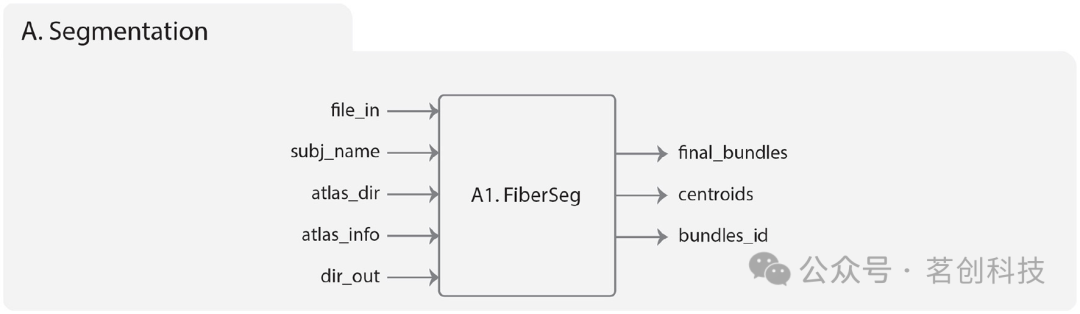
图2.分割模块图示。
其中ai和bi分别表示纤维A和B中点的三维坐标,两者具有相等数量的点(Np),按直接顺序列出。这里纤维A的点依次遍历为ai=[a1,a2,…,aNp],而纤维B的点同样定义为bi=[b1,b2,…,bNp]。因此,纤维B的逆序表示为bNp−i=[bNp,bNp−1,…,b1]。
Phybers分割模块中的FiberSeg具有以下输入(图2):
1.file_in:被试的全脑纤维束成像数据集文件。这些纤维束必须与所使用的纤维束图谱位于同一参考系中,并以bundles格式表示。
2.subj_name:被试名称,用于标记结果。
3.atlas_dir:bundle图谱文件夹,其中每个bundle都存放在单独的文件中,并以21个等距点进行采样。
4.atlas_info:与所用图谱相关联的文本文件,其中存储了应用分割算法所需的信息,即图谱束的列表,包含名称、分割阈值(mm)和每个束的大小。请注意,分割阈值可以根据要使用的数据库进行调整。
5.dir_out:存储算法生成的所有结果的目录名。
FiberSeg的输出是:
1.final_bundles:存储分割后纤维束的目录,即从被试纤维束数据集中提取的图谱束,这些束被标记并以bundles式保存在单独的文件中。
2.centroids:包含每个分割束质心的目录,以bundles格式保存在单个文件中。
3.bundles_id:一个文本文件,该文件记录了每个分割束的索引。
图3显示了使用DWM束图谱对来自HCP数据库中的一名被试进行纤维束分割的结果。显示的分割束包括丘脑辐射(B)、胼胝体节段(C)、弓状束(D)、扣带束(E)、下纵束、下额枕束、钩状束、皮质脊髓束和穹窿束(F)。图4显示了使用SWM束图谱的分割结果。该图谱包括93个束,根据从Desikan-Killiany图谱中提取的解剖ROIs进行标记。四组短联合纤维束为:尾侧额中束(B)、喙侧额中束(C)、枕外侧束(D)和缘上束(E)。
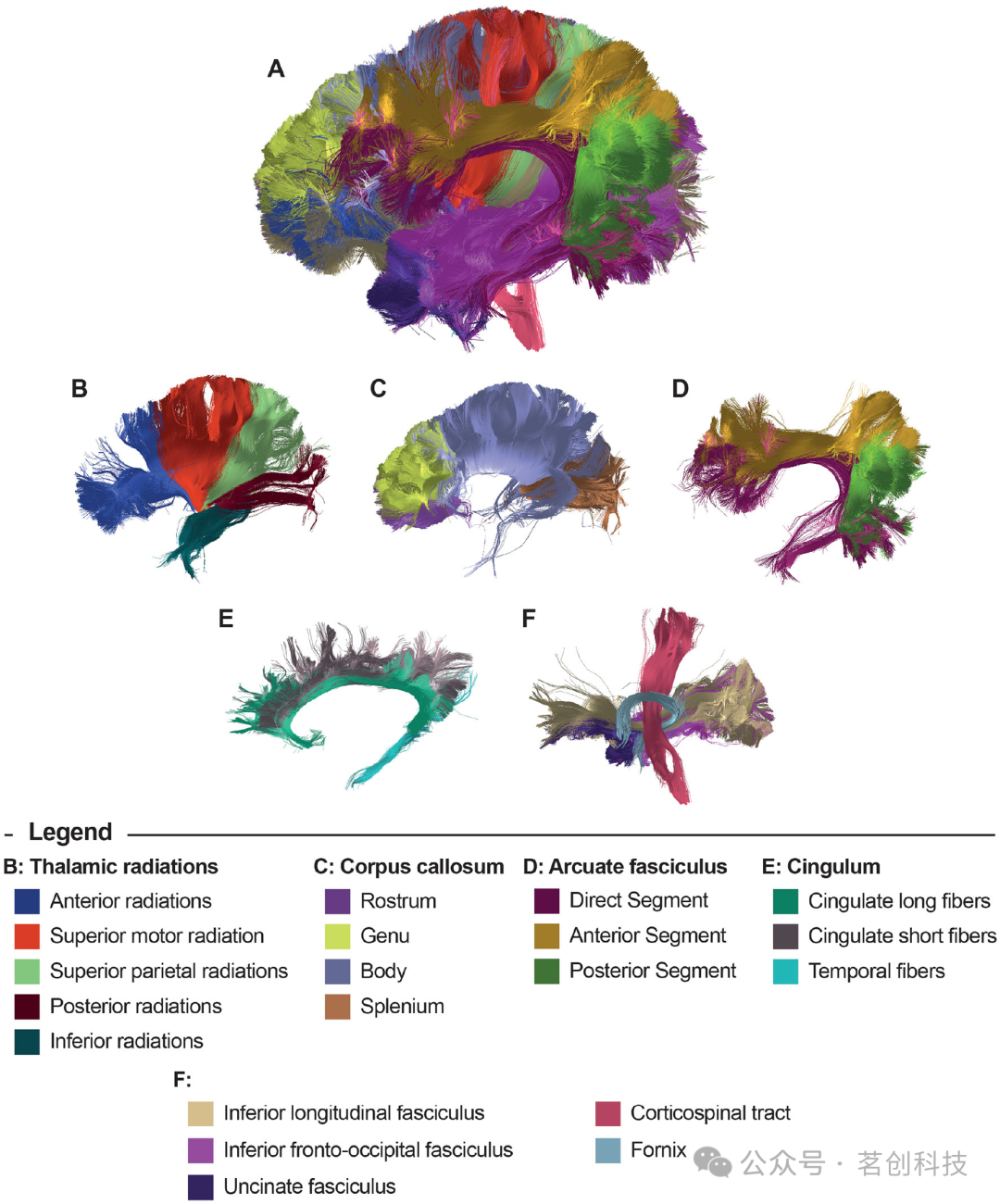
图3.使用DWM束图谱进行分割的结果。
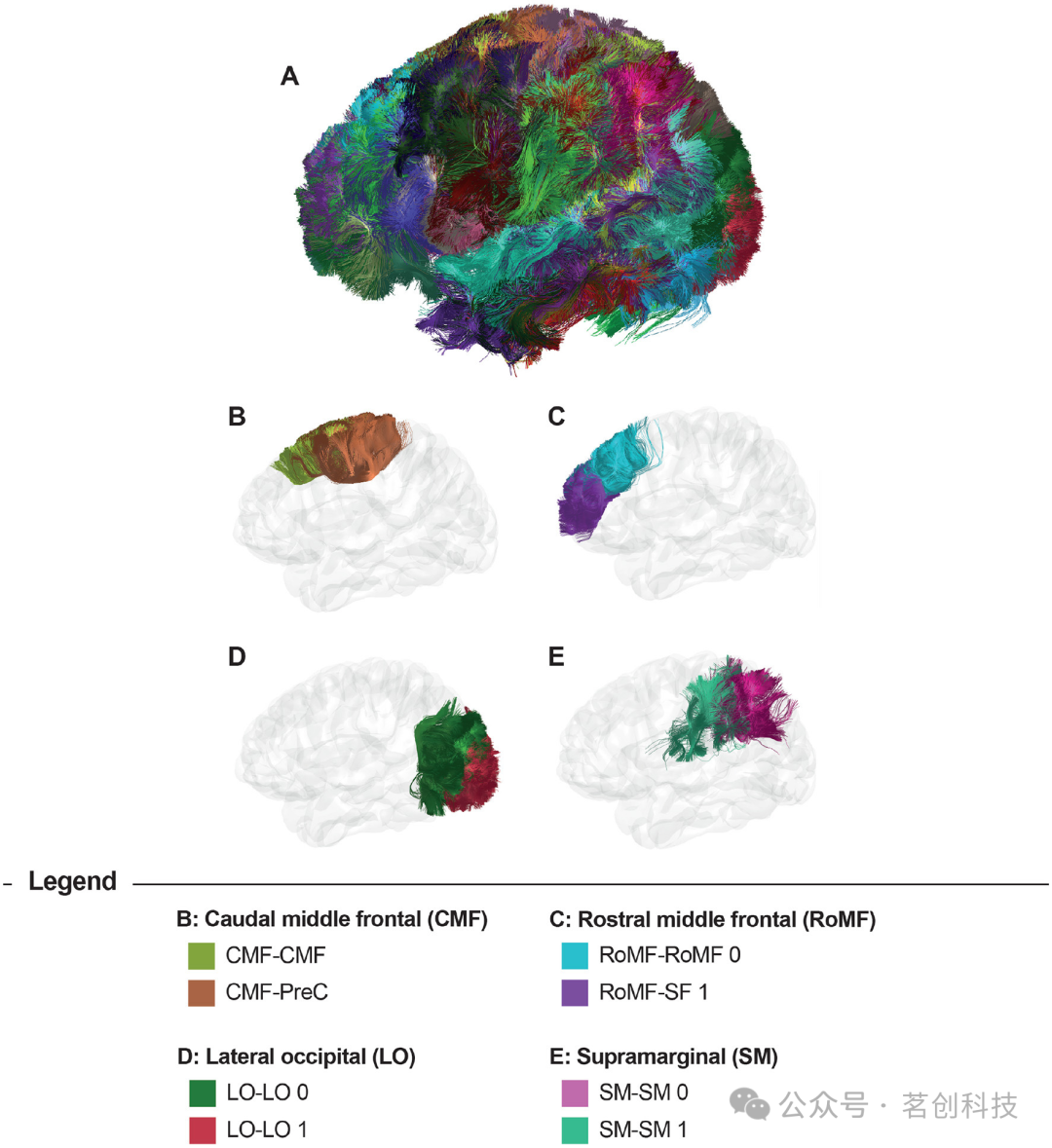
图4.使用SWM束图谱进行分割的结果。
聚类模块
①HClust子模块
HClust是一种分层聚类算法,它基于成对纤维之间的距离测量来创建束。该算法通过使用纤维点之间的最大欧氏距离(公式1)计算了纤维束数据集中所有纤维对之间的距离矩阵(dij)。然后,对于欧式距离小于最大距离阈值(fiber_thr)(mm)的纤维对,在dij矩阵上计算亲和图。亲和度由公式(2)计算得到。
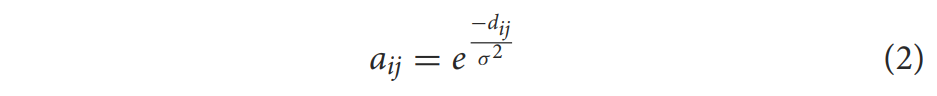
其中dij是元素i和j之间的距离,σ是定义相似度的参数(单位为mm)。
HClust的输入如下(图5B1):
1.file_in:输入的示踪数据文件。
2.dir_out:存储算法生成的所有结果的目录。
3.fiber_thr:最大距离阈值(单位:mm),默认为30mm。
4.partition_thr:自适应分割阈值(单位:mm),默认为40mm。
5.variance:相似度(mm),默认60mm。
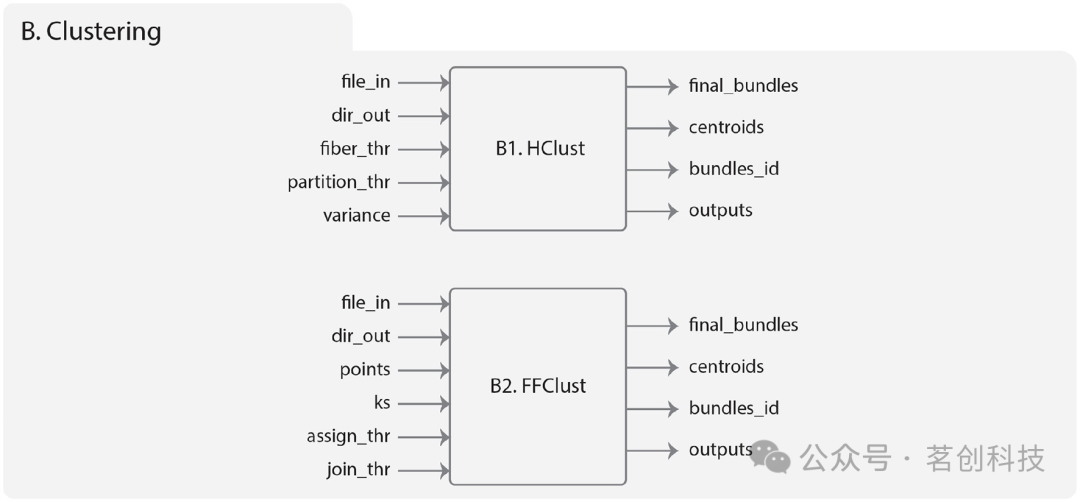
图5.聚类模块图示。
HClust输出为:
1.final_bundles:将所有生成的带有簇编号标签的纤维聚类存储在目录中,并以bundle格式保存在单独的文件中。
2.centroids:包含每个已创建聚类的质心的目录,以bundle格式保存在单个文件中。
3.bundles_id:一个文本文件,存储了被试纤维示踪数据集中每个聚类纤维的索引。
4.outputs:临时目录。
图6展示了将HClust算法应用于包含4000条纤维的示踪数据集的结果。左图显示了在聚类之前以蓝色呈现的4000条纤维的示踪情况,右图显示了手动选择并使用随机颜色调色板的八个纤维簇。本研究建议将HClust算法应用于最多包含40000条纤维的示踪数据集。如果希望将其应用于整个大脑并使用更大的数据集,则可以考虑首先利用FFClust的被试内聚类,然后将HClust应用于FFClust质心的策略。

图6.使用HClust计算中央后区域纤维示踪的结果。
②FFClust子模块
FFClust(快速纤维聚类)是一种被试内聚类算法,旨在识别大型纤维示踪数据集上紧凑且均匀的纤维簇。该算法包括四个阶段。首先,它在五个特定的纤维点上应用小批量K-Means聚类(阶段1),并合并共享相同点簇的纤维(阶段2)。然后,将小簇重新分配给较大的簇(阶段3),依次考虑纤维的正反序距离。最后,该算法将共享中心点的簇分组并合并由其质心表示的相近簇(阶段4)。纤维之间的距离被定义为对应纤维点之间的最大欧氏距离。该算法支持使用OpenMP进行顺序和并行执行。图5B2显示了模块的层次结构。其输入包括:
1.file_in:输入示踪数据集文件。
2.dir_out:存储算法生成的所有结果的目录。
3.points:点聚类(阶段1)中使用的点的索引,默认值:0、3、10、17、20。
4.ks:使用K-Means(阶段1)计算每个点的聚类数,默认值:300、200、200、200、300。
5.assing_thr:簇重新分配的最大距离阈值(单位:mm)(阶段3),默认值:6.0mm。
6.join_tht:簇合并的最大距离阈值(单位:mm)(阶段4),默认值:6.0mm。
FFClust的输出结构类似于HClust模块。
图7显示了将FFClust应用于全脑示踪数据集的结果(包含150万条流线)。使用Utils模块的PostProcessing子模块对检测到的簇进行筛选,以简化结果可视化。左图显示了尺寸大于150且长度在50-60mm之间的簇,而右图显示了尺寸大于100且长度大于150mm的簇。
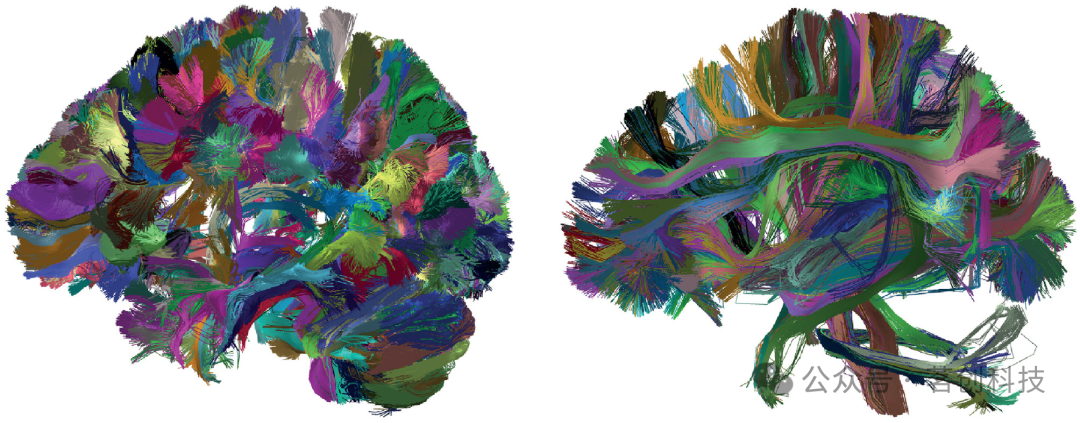
图7.使用FFClust算法获得的结果。
Utils模块
Utils模块是一组用于示踪数据集预处理以及大脑纤维聚类和分割结果分析的工具。该模块包括用于读写大脑纤维文件(以bundles格式)、将纤维转换到基于变形场的参考坐标系中、在一定数量的等距点上对纤维进行采样、计算脑纤维集之间的交点,以及提取测量值和过滤纤维簇或分割束的工具。本研究考虑了每个簇(或束)的大小、平均长度(mm)以及纤维之间的距离(mm)等指标。
①Deform子模块
Deform子模块(图8C1)使用非线性变换文件将示踪数据集文件转换到另一个空间。这些图必须以NIfTI格式存储,其中包含要应用于每个体素3D空间位置的变换信息。Deform子模块将变换应用于纤维点的3D坐标。Deform模块需要的输入数据包括形变图、待变换纤维的文件路径以及变换后的输出文件路径。
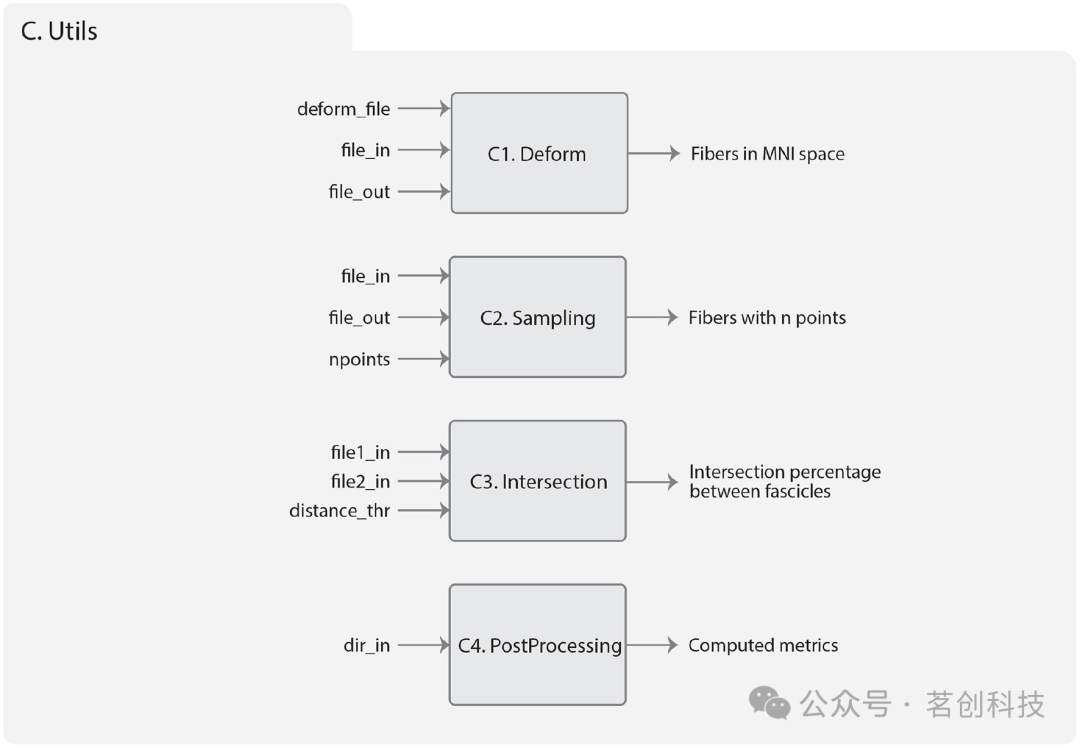
图8.Utils模块图示。
图9显示了在示踪数据集上应用变换函数的结果,使用解剖图像作为可视化参考。左图显示了在应用变换之前的结果,而右图显示了将示踪数据集变换到MNI空间后的结果。在左图中可以看到明显的错位,而右图则是进行校正后的结果。
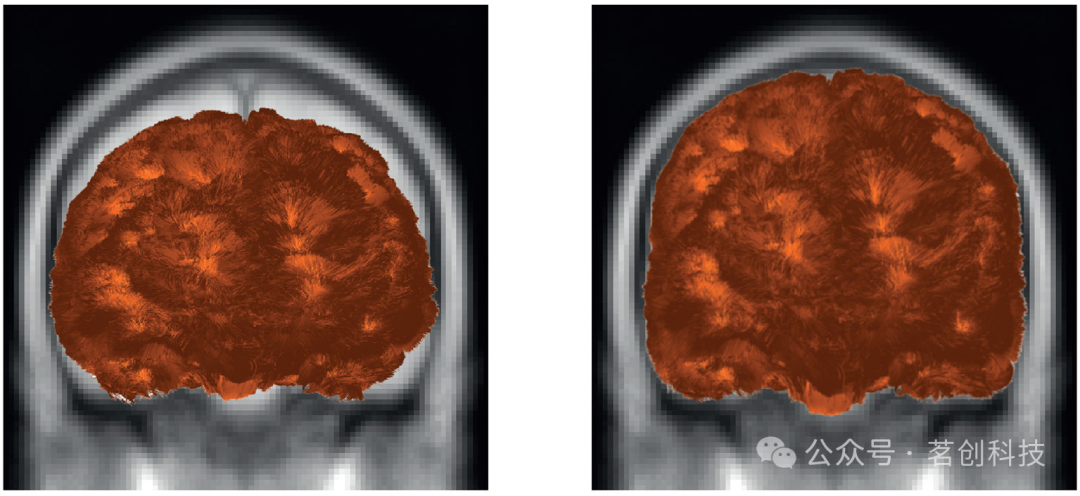
图9.使用Deform子模块进行纤维变换的示例。
②Sampling子模块
示踪数据集通常由大量具有可变数量点的三维折线组成。Sampling子模块(图8C2)对纤维进行采样,使用一定数量的等距点重新计算它们的点。该算法的输入数据包括待采样的示踪数据集文件路径,具有n个点的纤维束输出文件,以及点数(npoints)。Sampling子模块用于分割和聚类算法的预处理阶段。
③Intersection子模块
Intersection子模块(图8C3)计算两组脑纤维之间的相似度,它使用最大距离阈值(mm)来判断两种纤维是否相似。两组纤维必须处于同一空间。首先,计算两组纤维之间的欧氏距离矩阵。然后计算一组纤维与另一组纤维相似的纤维数量。相似性度量值的取值范围为0-100%,即输出的是相似性百分比。
④PostProcessing子模块
PostProcessing子模块(图8C4)包含一组可用于对聚类和分割算法生成的结果进行进一步处理和分析的工具。该工具构建了一个Pandas库对象(Dataframe),其中每个键对应于纤维集的名称(簇或分割束),其次是在纤维集上定义的度量指标,如纤维数量(大小)、纤维束内距离(mm)和平均长度(mm)。它可用于对聚类或分割结果执行单个或多个特征筛选。
可视化模块
可视化模块能够呈现多种类型的3D对象,包括纤维示踪数据集、网格和MRI扫描层或体积。对于每个对象,都定义了一组可以通过图形用户界面(GUI)访问的功能。GUI支持同时可视化多个对象,并对脑纤维示踪数据集应用线性变换等。图10说明了可视化模块的流程图。
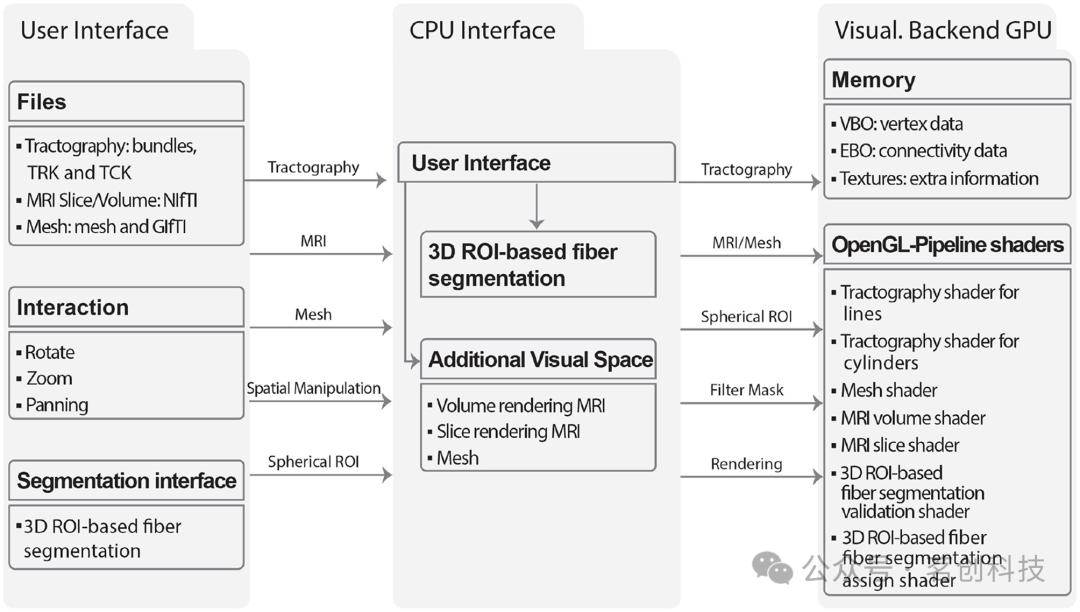
图10.可视化模块图示。
①可视化算法
纤维示踪数据集文件可以用线条或圆柱形式呈现。在线条情况下,软件加载流线,并为每个顶点定义一个固定的法线,该法线对应于流线特定段的归一化方向。此外,顶点着色器中的Phong光照算法用于计算流线的颜色。MRI数据使用特定的着色器进行层可视化和体绘制。网格可以使用点、线框或阴影三角形来显示。
②基于ROI的交互式3D纤维束分割
该功能允许用户使用球形感兴趣区域(ROI)交互式地提取纤维束。在内部,它创建了一个基于点的数据结构(Octree)用于快速查询。当一个节点被填满并且添加了一个新点时,该节点将其边界框细分为八个新的非重叠节点,并将点移动到新节点中。
在查询过程中,不同的3D对象会检查节点是否与边界框发生冲突或位于边界框内。在第一种情况下,算法会递归地遍历分支节点,直到到达叶节点,在该叶节点中,会测试点并将其添加到验证器缓冲区中。在后一种情况下,所有包含在子节点中的点都会被转换为相应的纤维,并在纤维验证器缓冲区中标记为已选。每个对象的所选纤维可以用于逻辑数学运算(AND、OR、XOR、NOT)。这样可以使用多个ROI来寻找连接特定区域的纤维,同时排除其他区域的纤维。图11展示了选择与两个ROI(绿色和紫色)相交的纤维,同时排除了与蓝色ROI相交的纤维。
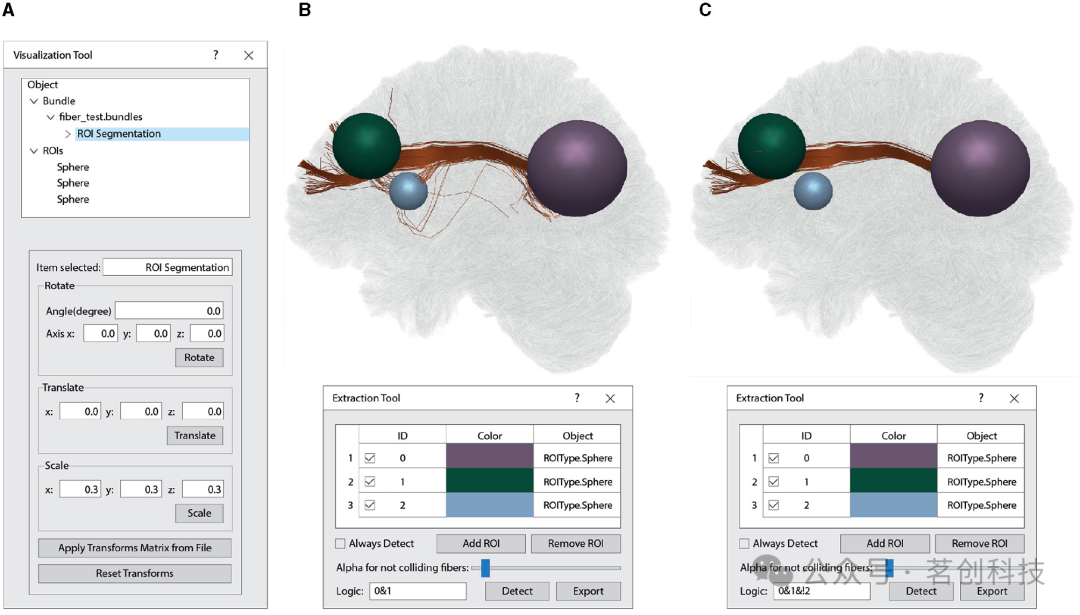
图11.使用可视化模块进行基于ROI的交互式3D纤维分割。
结论
本研究提出了一个名为Phybers的软件库,其中包含用于分析脑纤维束的最先进工具,旨在为科学界提供便利。Phybers集成了诸如纤维束分割、纤维聚类和可视化算法等工具。此外,还集成了用于对纤维示踪数据集进行采样和转换的实用工具,以及用于计算纤维束之间交集并对纤维集进行后处理的工具。该库提供了示例数据和详尽的文档,并且在开发时考虑到了可扩展性,因此可以集成其他现有的最先进算法。总的来说,Phybers有助于促进这些算法的运用,并实现最先进工具的更好共享。
参考文献:González Rodríguez LL, Osorio I, Cofre G. A, Hernandez Larzabal H, Román C, Poupon C, Mangin J-F, Hernández C and Guevara P (2024) Phybers: a package for brain tractography analysis. Front. Neurosci. 18:1333243. doi: 10.3389/fnins.2024.1333243
小伙伴们关注茗创科技,将第一时间收到精彩内容推送哦~
这篇关于Phybers:脑纤维束分析软件包的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!






