本文主要是介绍基于pytorch实现的DenseUnet医学图像分割(腹部多脏器),希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
1、前言
本章将介绍将densenet的主干网络引入unet中

官方实现的代码:kits19-challenge/network at master · nitsaick/kits19-challenge (github.com)
本章实现的项目目录如下:
主要代码有train、evaluate、predict脚本

2、代码介绍
数据预处理脚本
数据的预处理放在dataset脚本中,这里参考sam模型的预处理。利用numpy和cv进行归一化、翻转、图像增强等等,而非torch中的transform
主要如下:
红色框的部分为windowing窗口化拉伸对比度,因为大多数医学数据都是CT格式,对比度很差,如果原数据对比度还行的话,可以注释掉
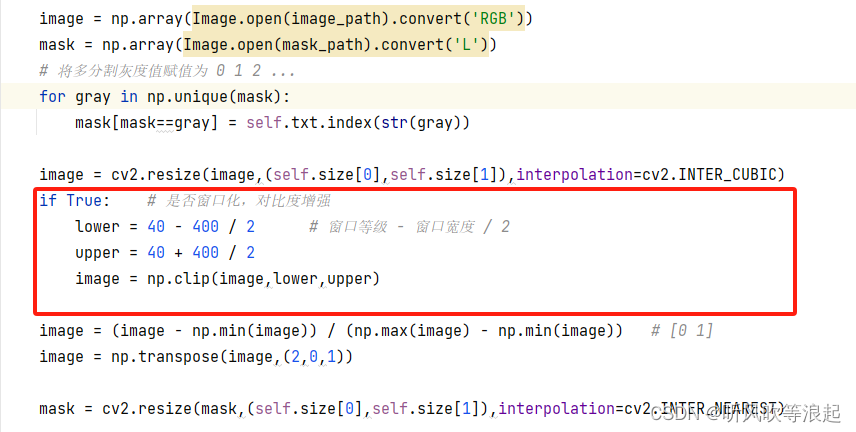
数据增强采用了水平和垂直翻转:

train 训练脚本
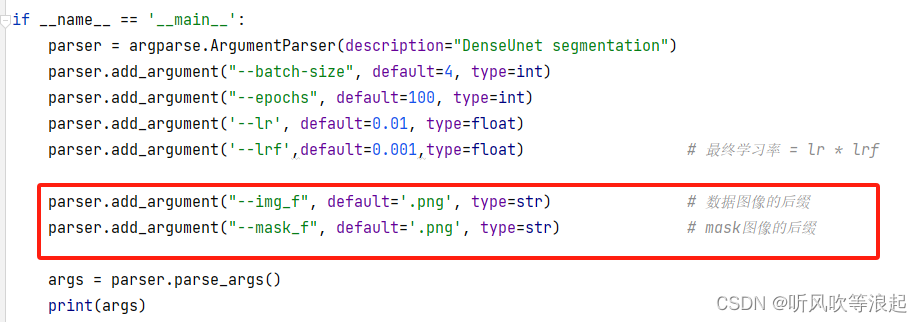
参数如下,如果image和mask的后缀格式不同,需要更改这里
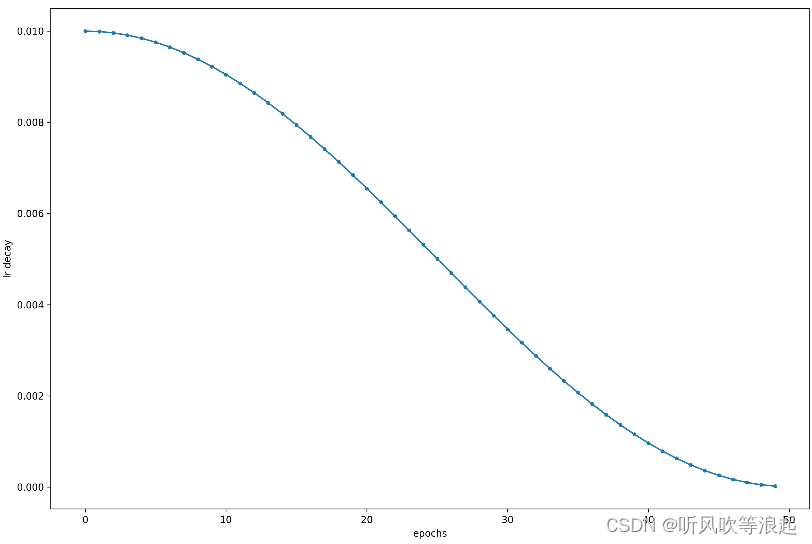
使用的优化器是Adam、损失是多类别的交叉熵、学习率衰减是cos余弦退火算法
evaluate 评估模型
默认采用训练过程中生成的最好的权重
代码会在测试集上进行评估,计算mean iou、recall、precision、全局pixel准确度等等
3、项目使用
测试用的数据集为腹部多脏器的五分割:
项目下载:基于DenseUnet对腹部多脏器5类的分割实战【包含代码+数据集+训练结果】资源-CSDN文库
3.1 数据集摆放
数据集摆放如下:

--data--train---images 训练集的图像 --data--train---masks 训练集的图像标签 --data--val---images 验证集的图像 --data--val---masks 验证集的图像标签 --data--test---images 测试集的图像(如果有的话) --data--test---masks 测试集的图像标签(如果有的话)
训练集用于训练网络、验证集用于验证模型调整超参数、测试集用于评估模型精度
3.2 训练
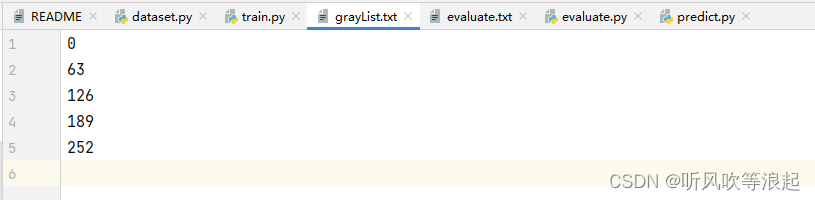
摆放好数据,直接运行train脚本即可,代码会计算mask的像素值,然后自动设定denseunet的输出类别个数
训练完成,会将所有结果保存在runs目录下:
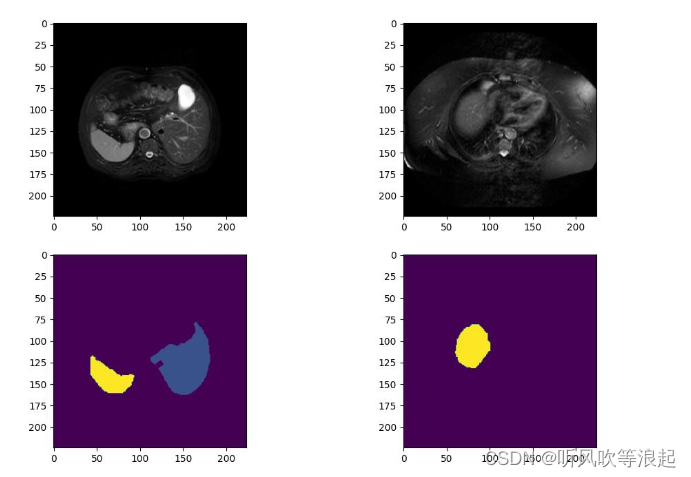
预处理可视化:
因为原图是MRI格式的,所有windowing方法增强效果不明显
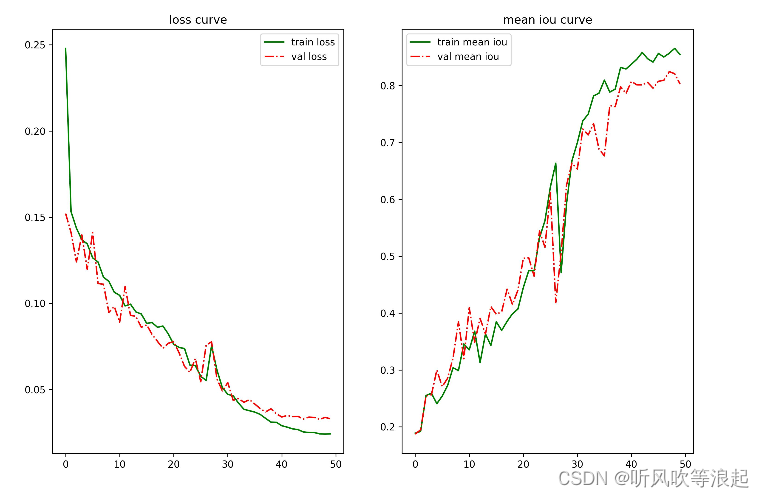
训练日志:
依次为epoch、train loss、train iou、val loss、val iou

学习率衰减:
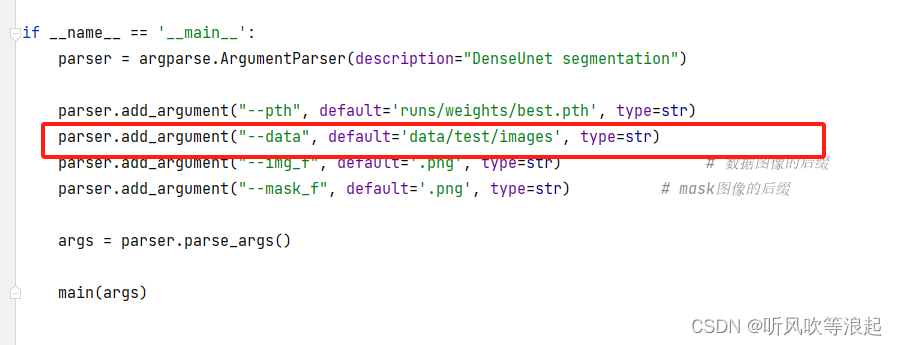
3.3 评估模型
脚本是evaluate代码,这里填写测试集路径即可
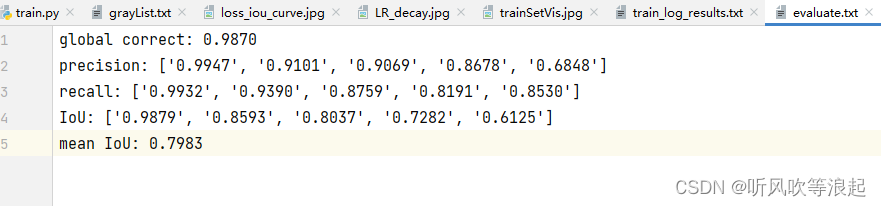
代码会计算测试集的精度,保存在txt文本中(runs目录)
列表的值,是不同类别的recall、iou等
3.4 推理代码
predict 脚本
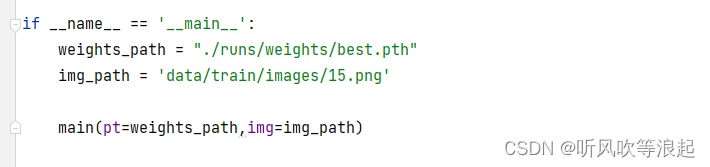
效果如下,会生成gt图以及image+gt的掩膜图
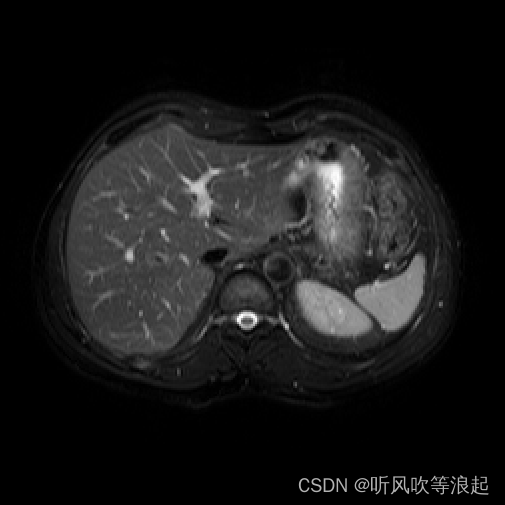
输入图像:
gt图:

掩膜图:

这篇关于基于pytorch实现的DenseUnet医学图像分割(腹部多脏器)的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







