wgdi专题
WGDI之深入理解blockinfo输出结果
blockinfo模块输出文件以csv格式进行存放,共23列,可以用EXCEL直接打开。 block info 其中16列非常容易裂解,描述如下 id 即共线性的结果的唯一标识chr1,start1,end1 即参考基因组(点图的左边)的共线性范围(对应GFF1的位置)chr2,start2,end2 即参考基因组(点图的上边)的共线性范围(对应GFF2的位置)pvalue 即共
如何用WGDI进行共线性分析(二)
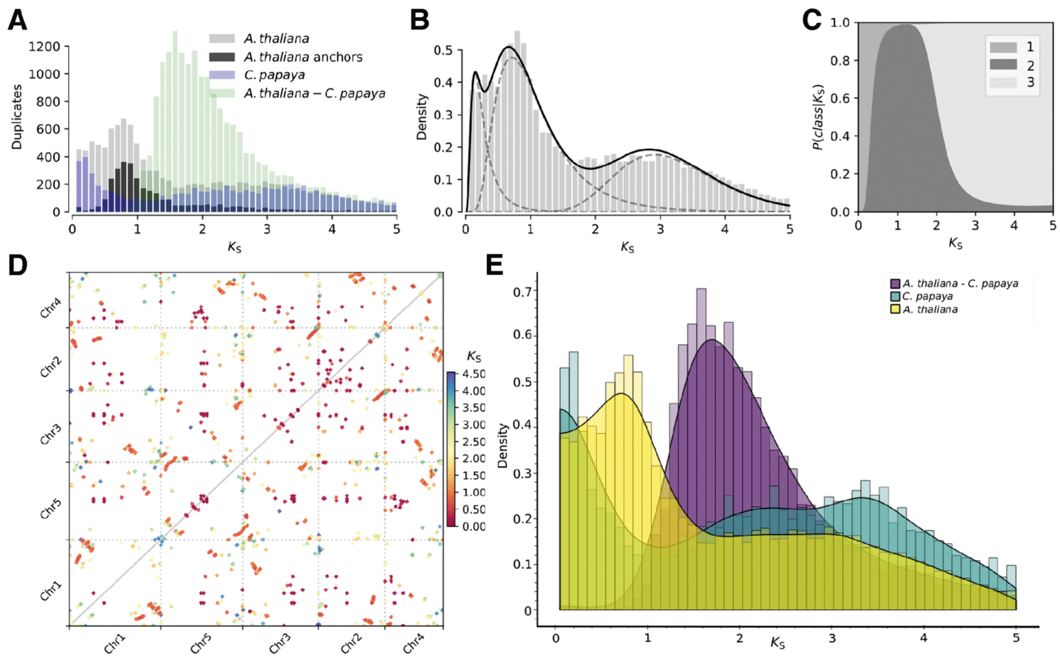
Ks可视化 我们在上一篇如何用WGDI进行共线性分析(一)得到共线性分析结果和Ks值输出结果的整合表格文件后,我们就可以绘制Ks点阵图和Ks频率分布图对共线性区的Ks进行探索性分析,从而确定可能的Ks峰值,用于后续分析。 Ks点阵图 最初绘制的点图信息量很大,基本上涵盖了历史上发生的加倍事件所产生的共线性区。我们能大致的判断片段是否存在复制以及复制了多少次,至于这些片段是否来自于同一次加倍事件则
如何用WGDI进行共线性分析(上)
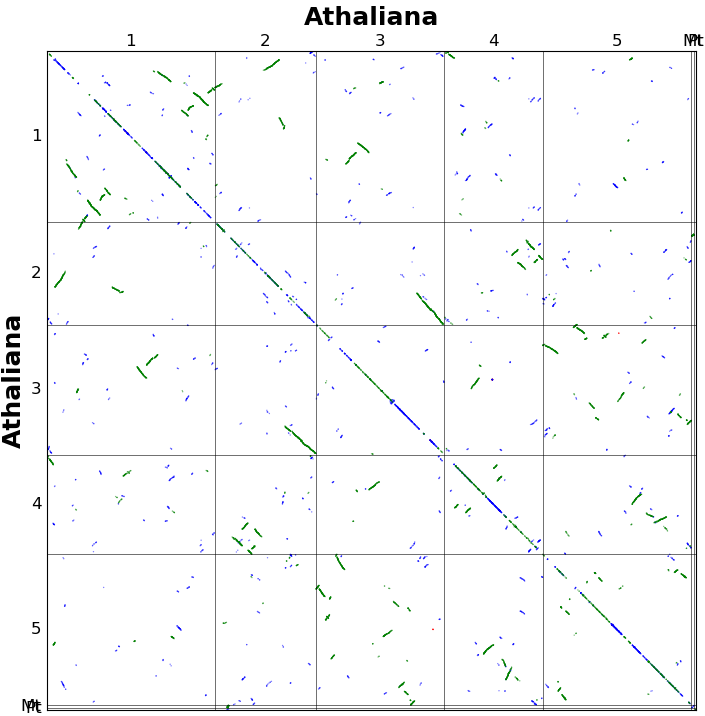
多倍化以及后续的基因丢失和二倍化现象存在于大部分的物种中, 是物种进化的重要动力。如果一个物种在演化过程中发生过多倍化,那么在基因组上就会存在一些共线性区域(即两个区域之间的基因是旁系同源基因,其基因的排布顺序基本一致)。 例如拟南芥经历了3次古多倍化,包括2次二倍化,一次3倍化 (Tang. et.al 2008 Science). ..For example, Arabidopsis th
WGDI之深入理解blockinfo输出结果
blockinfo模块输出文件以csv格式进行存放,共23列,可以用EXCEL直接打开。 block info 其中16列非常容易裂解,描述如下 id 即共线性的结果的唯一标识chr1,start1,end1 即参考基因组(点图的左边)的共线性范围(对应GFF1的位置)chr2,start2,end2 即参考基因组(点图的上边)的共线性范围(对应GFF2的位置)pvalue 即共