scanpy专题
Scanpy(2)多种可视化
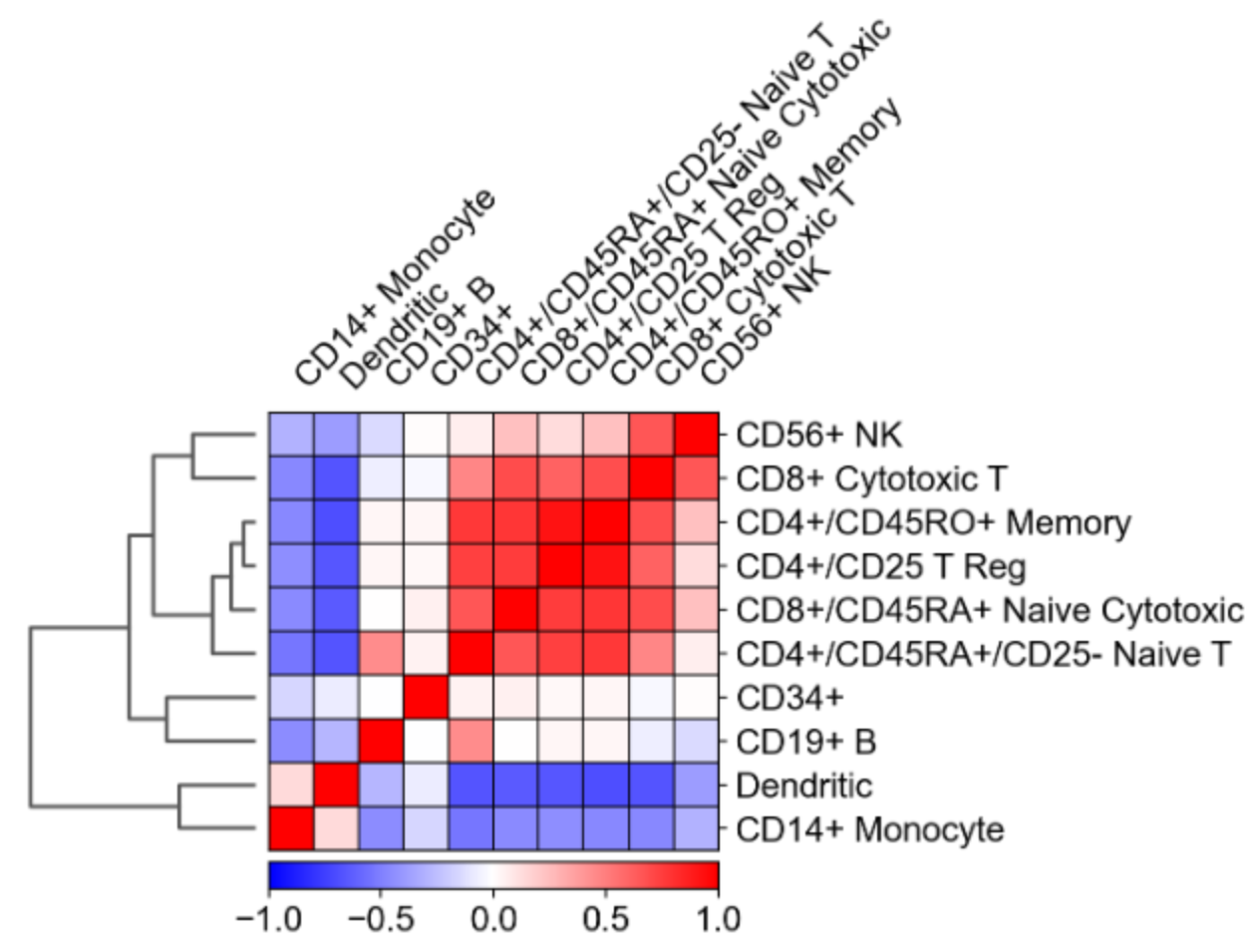
本篇内容为scanpy的可视化方法,可以分为三部分: embedding的散点图;用已知marker genes的聚类识别(Identification of clusters);可视化基因的差异表达; 我们使用10x的PBMC数据集(包含68k个细胞)。Scanpy在其发行版中包含了这个数据集的缩减版,该数据集只包含700个细胞和765个高变基因。此数据集已经过预处理和UMAP计算。 在本
R的seurat和python的scanpy对比学习
现在的单细胞分析,往往避免不了scanpy的使用,我们可以通过对比seurat来学习scanpy 今天的格式怎么都改不了。。。手机阅读有点费劲,,推荐电脑阅读。 单细胞数据分析概览 单细胞分析,总流程 python教程 seurat教程 seurat中与scanpy对等的函数操作 数据预处理 Seurat (R) Creat
【Scanpy】单细胞转录组分析思路之细胞分类
本文将详细讲述单细胞转录组分析的步骤。 一、文章信息 题目:ClusterMap for multi-scale clustering analysis of spatial gene expression 链接:https://doi.org/10.1038/s41467-021-26044-x 期刊:Nature 二、数据集 数据集:小鼠初级
python小白入门scanpy,我的学习之路
第一天 :dIntroduction · HonKithttps://python.swaroopch.com/ 这本书重点看看数据结构就行了 简单入门 第二天GitHub - huangsam/ultimate-python: Ultimate Python study guide for newcomers and professionals alike.Ultimate
scanpy赋值问题
今天发现一个很奇怪的bug import numpy as npimport pandas as pdimport anndata as adfrom scipy.sparse import csr_matrixprint(ad.__version__)counts = csr_matrix(np.random.poisson(1, size=(100, 2000)), dtype=n
scanpy赋值问题
今天发现一个很奇怪的bug import numpy as npimport pandas as pdimport anndata as adfrom scipy.sparse import csr_matrixprint(ad.__version__)counts = csr_matrix(np.random.poisson(1, size=(100, 2000)), dtype=n