nii专题
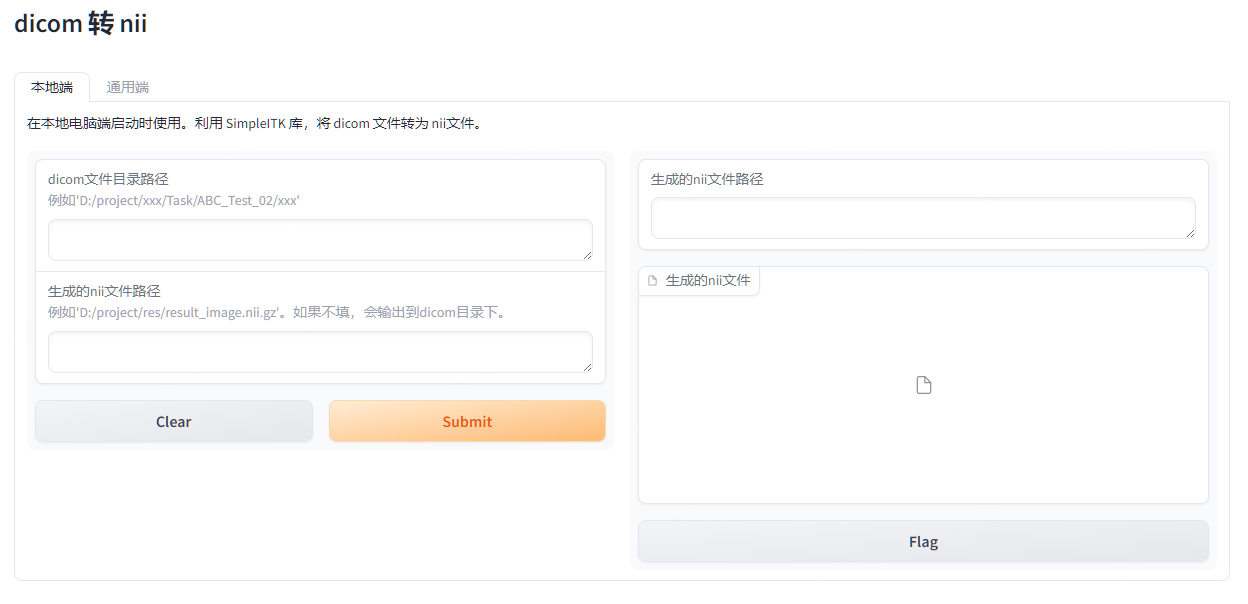
Gradio 案例——将 dicom 文件转为 nii文件
文章目录 Gradio 案例——将 dicom 文件转为 nii文件界面截图依赖安装项目目录结构代码 Gradio 案例——将 dicom 文件转为 nii文件 利用 SimpleITK 库,将 dicom 文件转为 nii文件更完整、丰富的示例项目见 GitHub - AlionSSS/dcm2niix-webui: The web UI for dcm2niix (DICO
python使用nibabel和sitk读取保存nii.gz文件
nii.gz格式是医学图像常用的压缩格式,python中可用nibabel和sitk来读取保存。 使用nibabel 由于使用nibabel图像会旋转90度,所以读取保存的时候还得保存映射信息,3维图像格式为(z, x, y) 读取nii.gz文件 img = nib.load('xxxxx.nii.gz')img_affine = img.affineimg = img.get_d
matlab 2018b查看nii图像
1.下载Tools for NIfTI (ANALYZE) MR image工具包: 谷歌第一个:https://www.mathworks.com/matlabcentral/fileexchange/8797-tools-for-nifti-and-analyze-image 下载后将解压后的文件夹放入MATLAB安装路径中的toolbox中 2.在MATLAB中加载工具包路径;
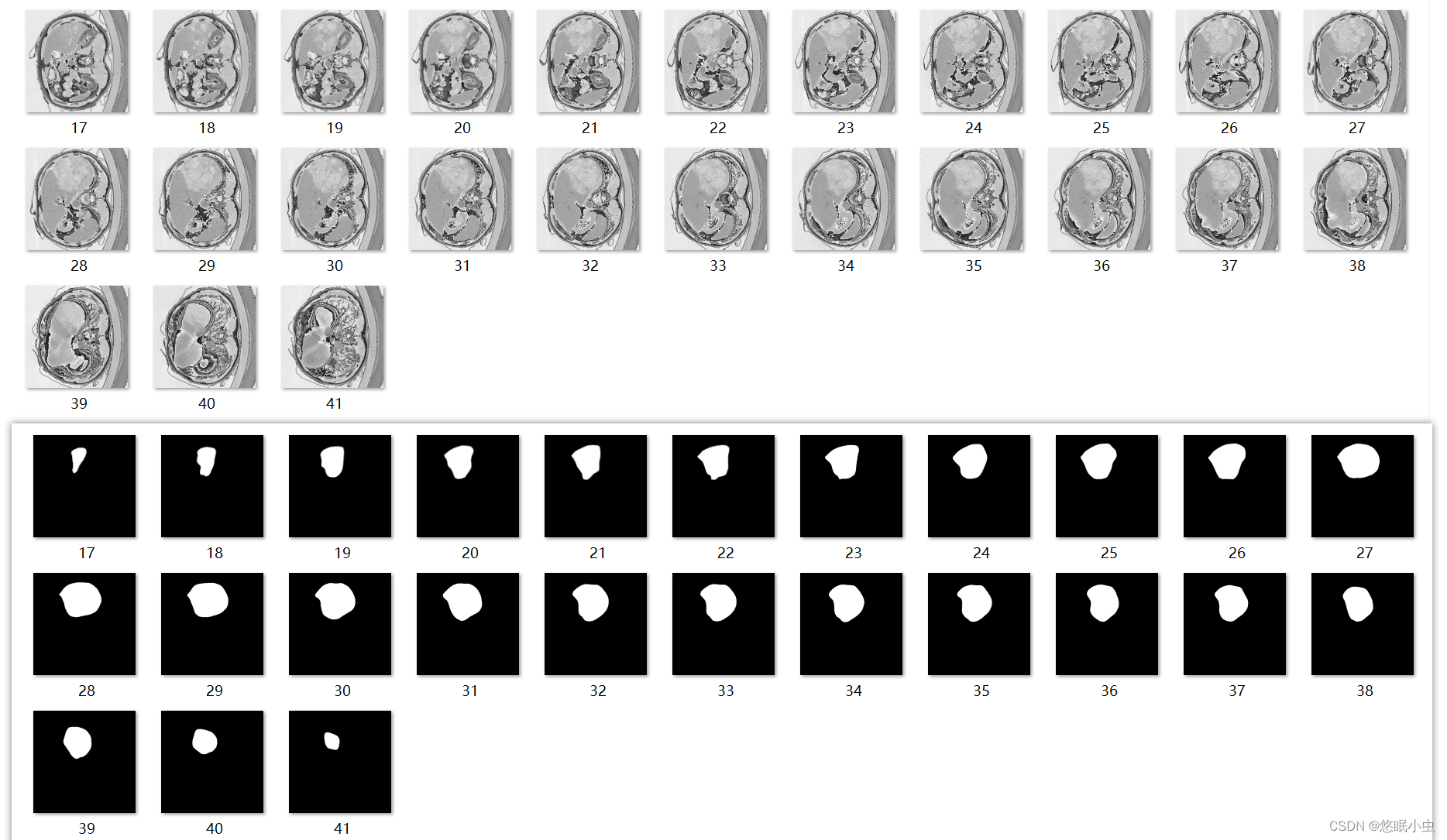
医学图像处理:nii格式转换(3D切片为2D)
目录 NIFTI文件结构 读取NII文件 ITK-SNAP安装 使用方法 NII转PNG NIFTI文件结构 NIFTI 格式,是一种用于存储和交换医学成像数据的文件格式,特别适用于神经影像学领域。NIFTI文件通常有两个扩展名:.nii(用于图像数据)和 .hdr(用于头文件,包含有关图像的元数据)。这两个文件是成对出现的,.hdr 文件包含有关图像数据的详细信息,
nii格式mask映射为新的mask
在DCE图像上勾画的ROI转换为nii格式形成的mask,形状和DCE的原始图像一致,用于特征提取,形状不一致不能特征提取; DCE图像和T2图像的形状不一致,所以mask不能用于T2的特征提取; 这个脚本根据DCE勾画的mask,和T2的图像,生成T2的mask,形状和T2一致,用于特征提取; import globimport osimport shutilimport numpy a
医学图像预处理(一)——读取图片(dcm,nii,PIL,skimage,cv2)
总结一下目前遇到的一些医学图像预处理步骤,但才开始自学两周,所以很多东西或许不对或者不全面,以后争取每两周回顾以前的文章,将内容完善,并提高质量。(2019-3-5) 由于篇幅原因,将预处理的步骤分开来,这一篇将总结读取图片的方法。 2019/7/7更新,写在最前:关于图片读取出来宽、高顺序问题 (文中nii读取图片那节也有提到图片读取出来的w,h先后问题。) 有这样的疑问是看得多了之后发现有时候
【matlab】如何将.mat文件与.nii文件互转
【matlab】如何将.mat文件与.nii文件互转 .mat转为.nii文件 有时候代码需要读取的是.nii文件,但是如何现有的数据是.mat格式,需要将.mata转化为.nii文件 1、先加载.mat文件 % 加载.mat文件load('your_mat_file.mat'); % 请将'your_mat_file.mat'替换为实际的.mat文件名 2、将.mat转为.nii
如何处理.nii文件
最近读了一篇论文[1],是利用深度学习进行MRI图像重建的,作者在github[2]上提供给我们的他的实现代码,他使用的一个MRI分割比赛的数据集[3],但是将数据集下载下来发现全部都是.nii格式的文件,用普通的方法也是打不开的,经过一个星期的摸索,也是大概了解了如何读取以及处理.nii文件。 1. NIFTI出现原因 .nii文件是NIFTI格式的文件,出现的原因是原来一种图像格式是
nii convert to 2D image【python】
可以自己精简,我的label是二分类 import SimpleITK as sitkimport cv2from PIL import Imageimport numpy as npimport nibabel as nib # nii格式一般都会用到这个包import imageio # 转换成图像import osimport numpy as npfrom scipy.
从rookie到基佬~014:Python代码 DICOM(.dcm)数据转化为NIFTI(.nii)数据
一天一个变弯小技巧 今日份洗脑: DICOM(.dcm)数据转化为NIFTI(.nii)数据 结论:SimpleITK:他好,我也好,汇源肾宝 医学图像的存储格式 1、NIFTI(.nii)是Neuroimaging−Informatics−Technology−Initiative即神经影像信息技术,NIFTI格式被认为ANALYZE7.5格式的替代品。NIFTI最初是用于神经成像的,但
几种读nii图像方法的轴序比较
读 .nii / .nii.gz 图像并转成 numpy 可用 medpy.io、nibabel、itk、SimpleITK 几种方法,然而几种方法读出来的轴序有出入,本篇比较此几种方法。 Datum 所用数据来自 verse,经 iTomxy/data/verse/preprocess.py 预处理,朝向和轴序是 RAI。三视图(可视化:show_nii): 其中最短的是 LR = 35,
医学图像 ABIDE 等数据集 .nii.gz Python格式化显示
nii.gz 文件 .nii.gz 文件通常是医学影像数据的一种常见格式,比如神经影像(如脑部MRI)。这种文件格式通常是经过gzip压缩的NIfTI格式(Neuroimaging Informatics Technology Initiative)。 要在Python中查看.nii.gz文件,你可以使用一些专门的库,如Nibabel(NiBabel是用于读取和写入神经影像数据的Python库
读取nii数据,转为volume.dat二进制数据
代码 #include<iostream>#include<itkImage.h>#include"functions.h"#include<exception>#include<itkRescaleIntensityImageFilter.h>using GrayPixelType = unsigned char;using OrgPixelType = short;using Or
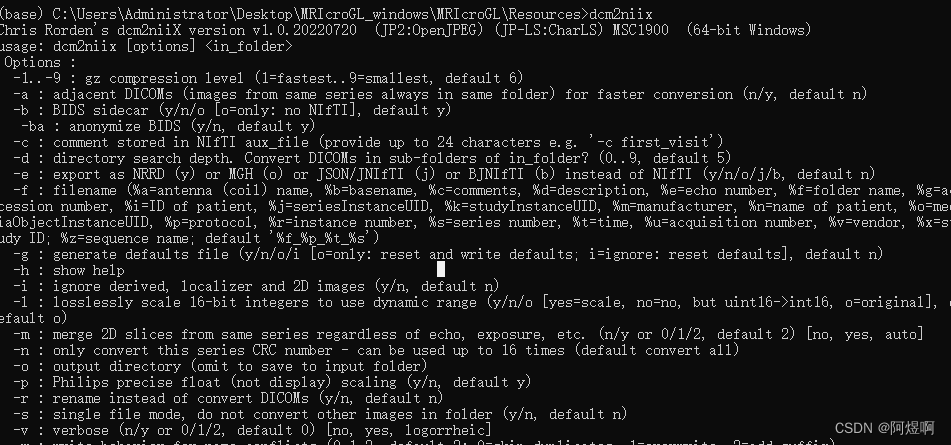
批量dicom格式转nii(dcm2nii),目前网上最全总结与最强转换方法!!
提示:文章写完后,目录可以自动生成,如何生成可参考右边的帮助文档 文章目录 Dcm2nii各方法目前存在的问题又快又稳定的批处理方法!总结 Dcm2nii各方法目前存在的问题 使用mricron的dcm2niigui.exe转换,此方法虽然可以成功转换,但存在的问题如下:①输出的nii可能不止一个;②只能输出到同级目录,不能自定义输出文件深度。使用matlab中的sp
【ACDC数据集】:预处理ACDC心脏3D MRI影像数据集到VOC数据集格式,nii转为jpg,label转为png
【Segment Anything Model】做分割的专栏链接,欢迎来学习。 【博主微信】cvxiaoyixiao 本专栏为公开数据集的介绍和预处理,持续更新中。 文章目录 1️⃣ ACDC数据集介绍2️⃣ ACDC数据集样例 3️⃣ 预处理ACDC目标 4️⃣ 处理结果样图 5️⃣ 代码 6️⃣ 划分测试集和训练集 1️⃣ ACDC数据集介绍 他是一个多类别的心脏3D MR
matlab 读写磁共振影像.nii 数据
问题描述: 参考LEiDA方法和周昌松老师的large-scale network 的分离整合的划分[1],脑区划分结束后需要把脑区的结果用脑图的形式呈现出来,但是因为我划分脑区的结果都是保存在mat文件里,Brainnet Viewer 则需要使用 .nii 类型的文件。因此,研究了一下如何将数据写入.nii文件。 加载BrainNet Viewer的样例数据 nifti_data
Matlab R2016a软件读取、显示和保存nii图像
最近在做医学图像处理,就会遇到通过软件读取、显示和保存.nii图像,网上查了好多资料,都感觉很模糊,好不容易找到了解决办法。故整理于此,方便再次学习。 第一步,需要下载Tools for NIfTI (ANALYZE) MR image工具包 下载完成后,解压文件放到Matlab R2016a的安装路径的toolbox中。 第二步,在MATLAB中加载工具包路径。 在“主页”