mer专题
GenomeScope——jellyfish k-mer分析的下游分析
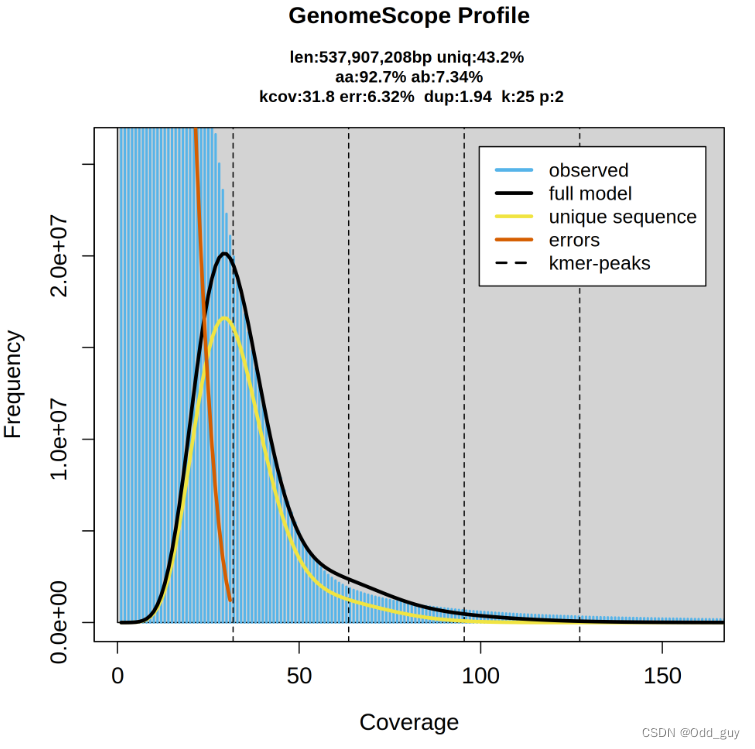
GenomeScope 2.0是一个可对多倍体基因组数据进行概括性分析的有效工具,其可以将jellyfish或KMC等k-mer分析软件的结果作为输入,并拟合混合模型,对基因组数据进行更深入的挖掘。 一、安装 GenomeScope 2.0的工作依赖于R,且需要安装其依赖包:argparse、minpack.lm。可以使用conda工具进行安装: #安装依赖包conda install -
DNA特征提取之序列组装K-mer算法
基因就像一本天书,里面的字都是有A,T,C,G组成的。我们测序出来的大于几百bp的小的序列,我们叫做reads。我们测序的结果中有上百万条的reads,而这些reads的位置我们又不知道,我们只能根据他们重叠的部分来尽量还原他的原型。 k-mer是指将reads分成包含k个碱基的字符串,一般长短为m的reads可以分成m-k+1个k-mers。举个例子吧:为了简化,有这么个reads(当然实
使用社区OBS编译xbmc(for mer)
首先是在社区注册账户https://build.pub.meego.com/user/register_user,当然了, 和负责管理的人员有两个邮件来回,账号就被批准可用了。 接下来的,登录创建工程,添加包,设置Project Config < project name = "home:licl" > < title >licl's Home Project</ tit