本文主要是介绍在pharmit里匹配药效团,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
我把400个无活性的小分子(decoys)提交到pharmit里。
命名为decoyset00~decoyset08,查找时,按这个找。
1、导入药效团配体:
进入药效团筛选界面:
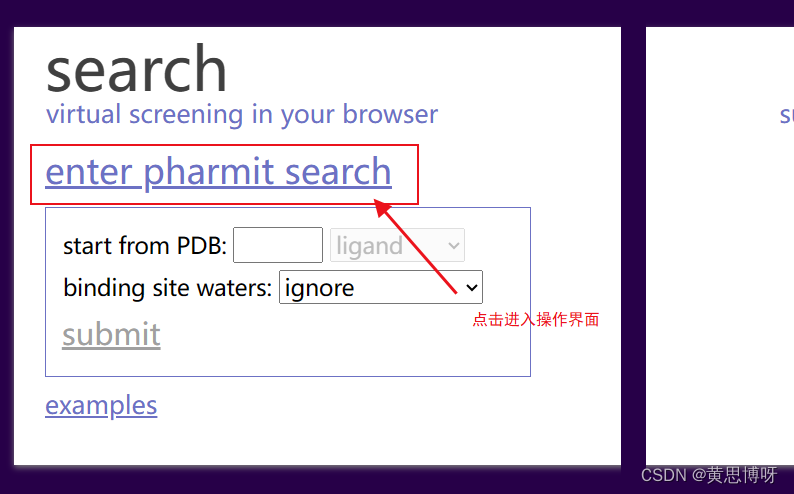
导入代表药效团模型的活性肽构象:
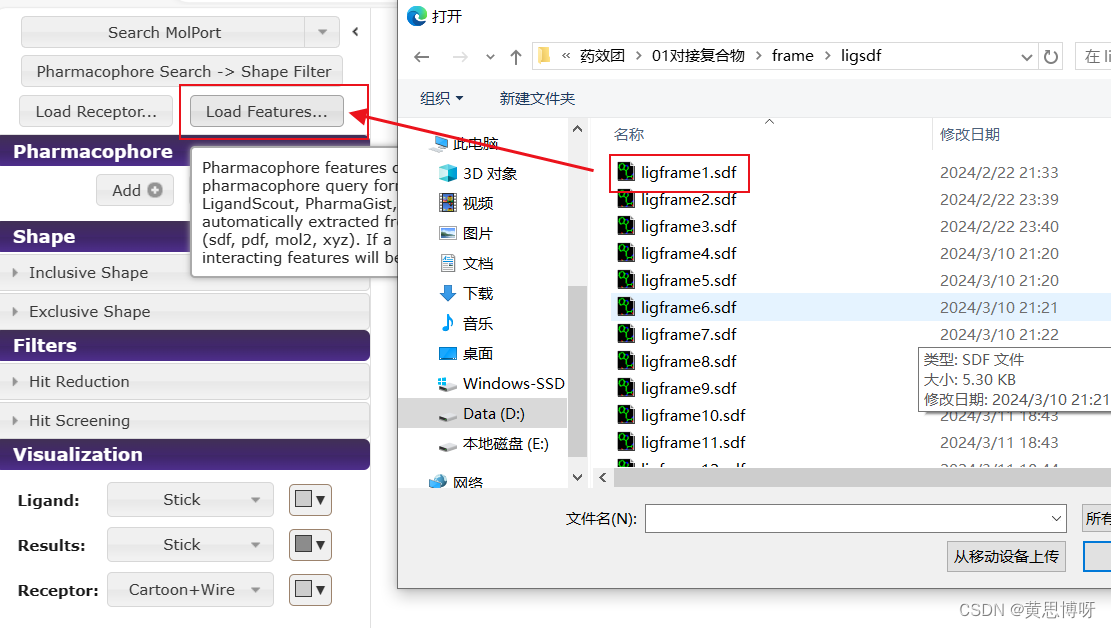
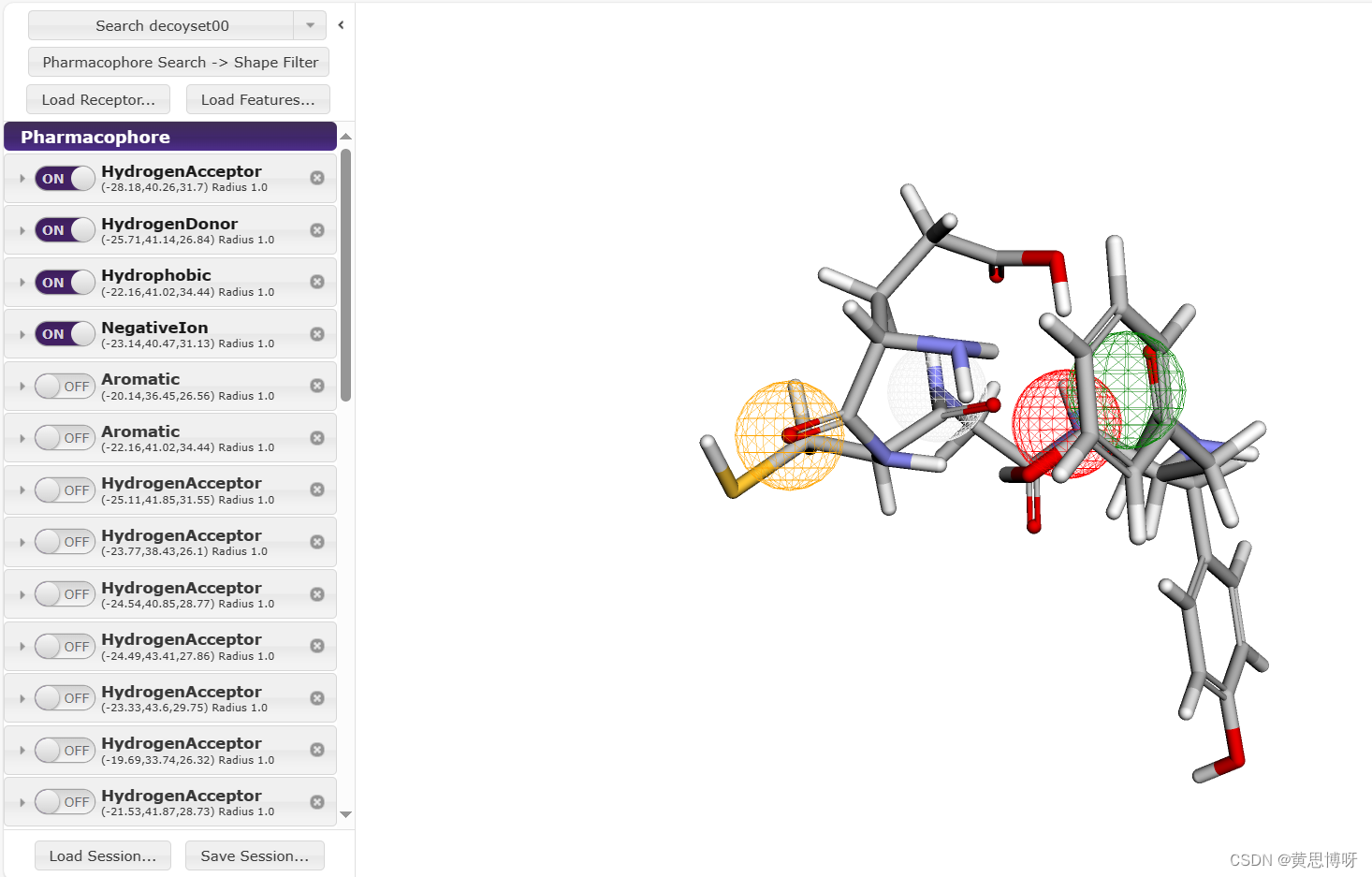
2、选择预先设置好的药效团特征空间排列:
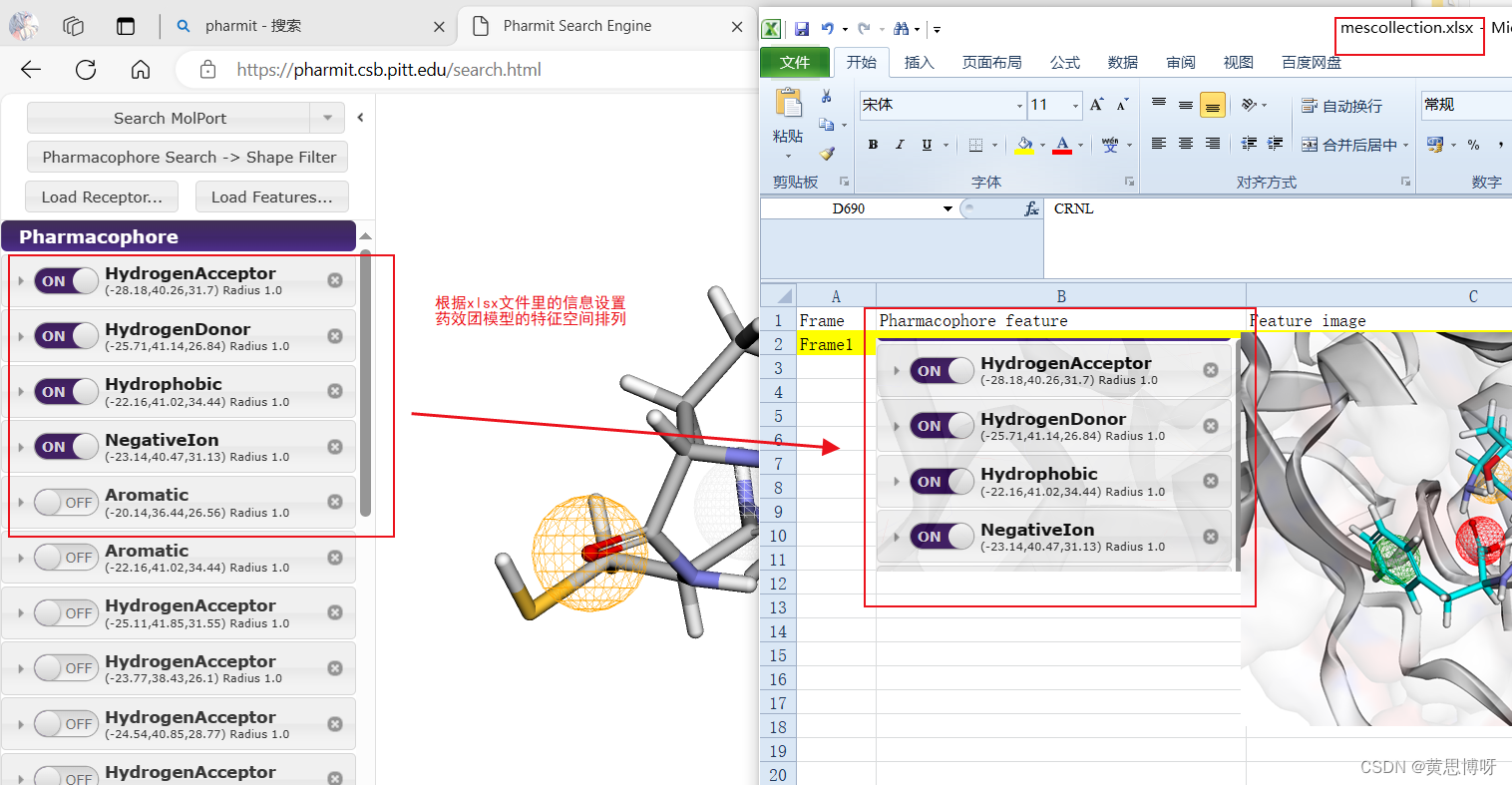
打开mescollection.xlsx文件,根据每个frame里记录的药效团特征信息,恢复pharmit网站里的药效团模型:
3、对诱导剂分子数据集进行匹配:
点击Search CHEMBL32按钮,在Contributed Libraries里搜索对应的诱导剂数据集,例如这里是decoyset00:

点击Search decoyset00,等待右侧的匹配结果 出现:
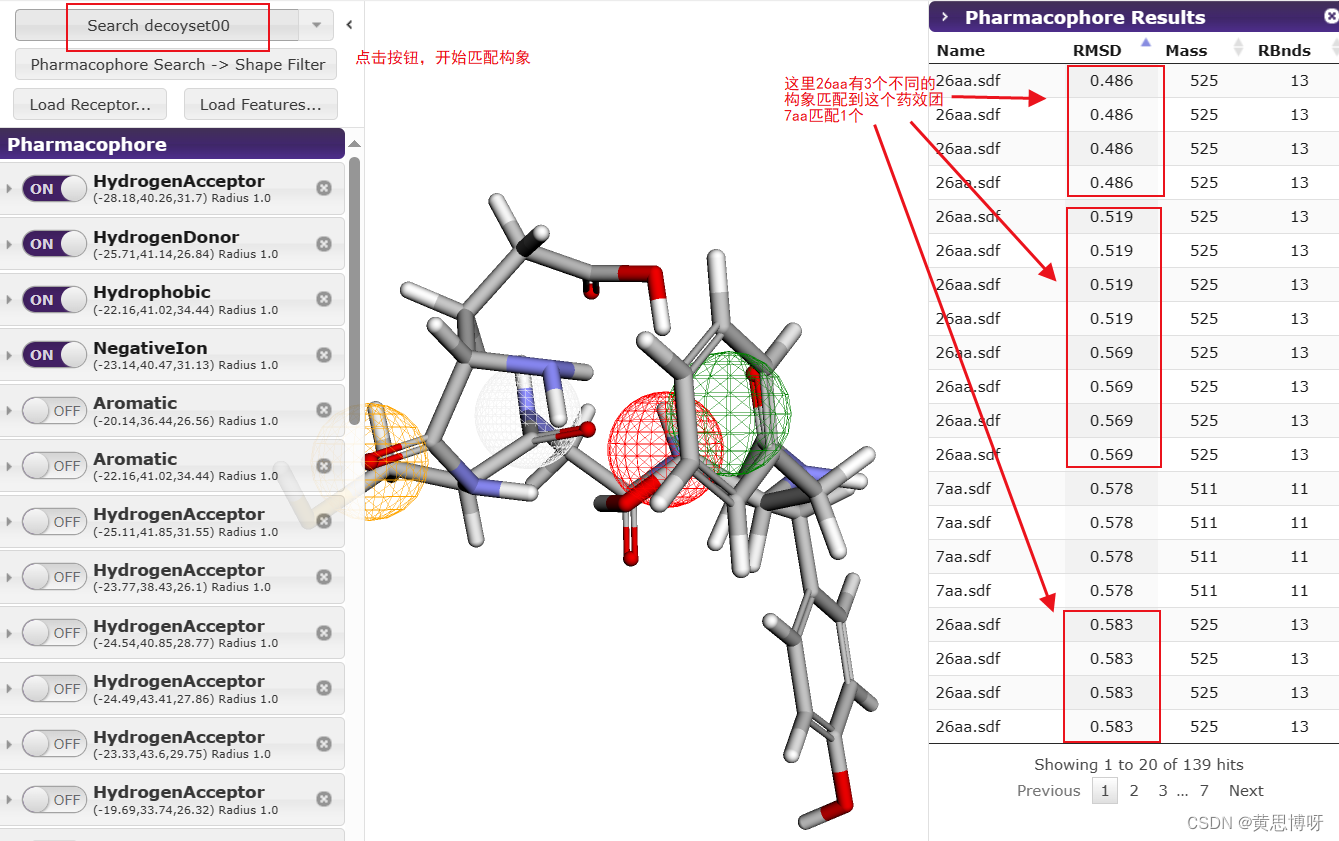
在第1页表格里26aa构象里匹配到4个,7aa匹配到1个构象。
4、记录匹配到的结果:
可以先将第1页表格的数据记录到excel里:
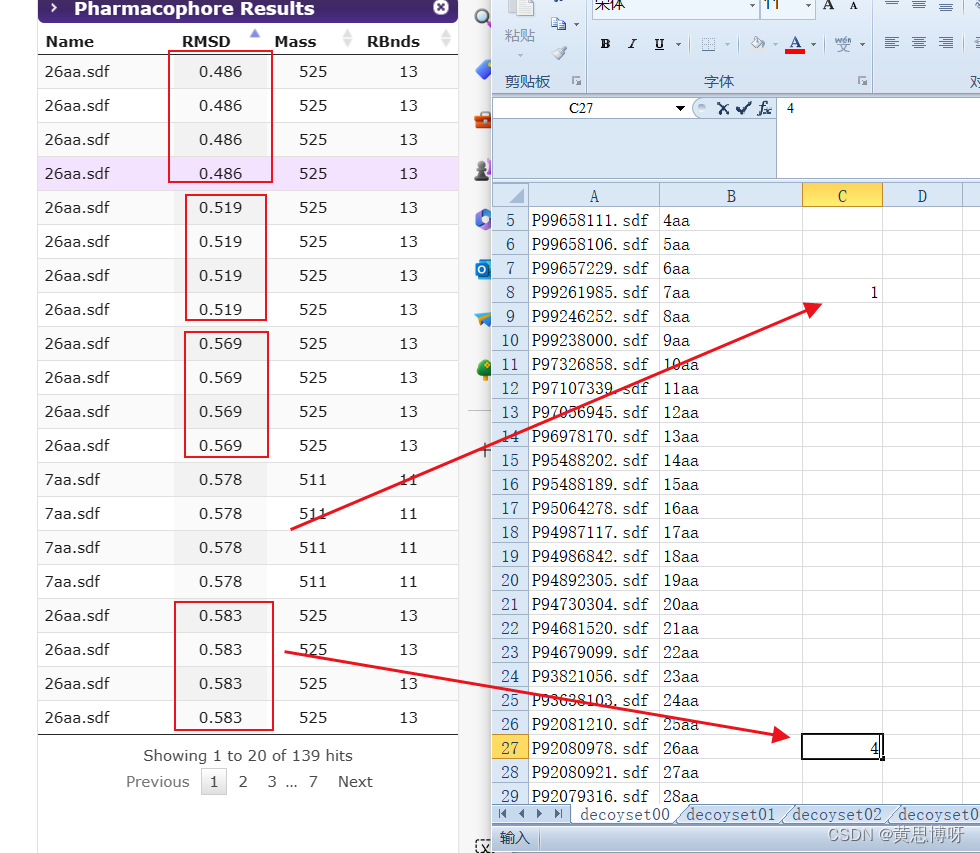
逐页更新表格的内容,例如第2页表格里26aa又出现3个构象成功匹配模型,就将原先4加上3变成7:
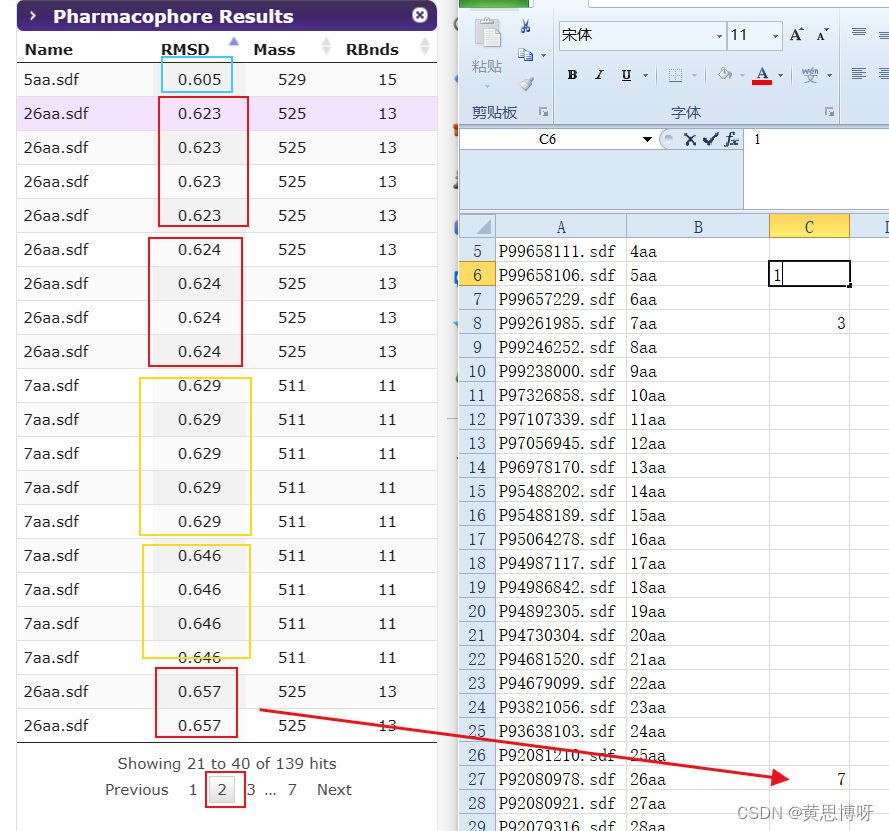
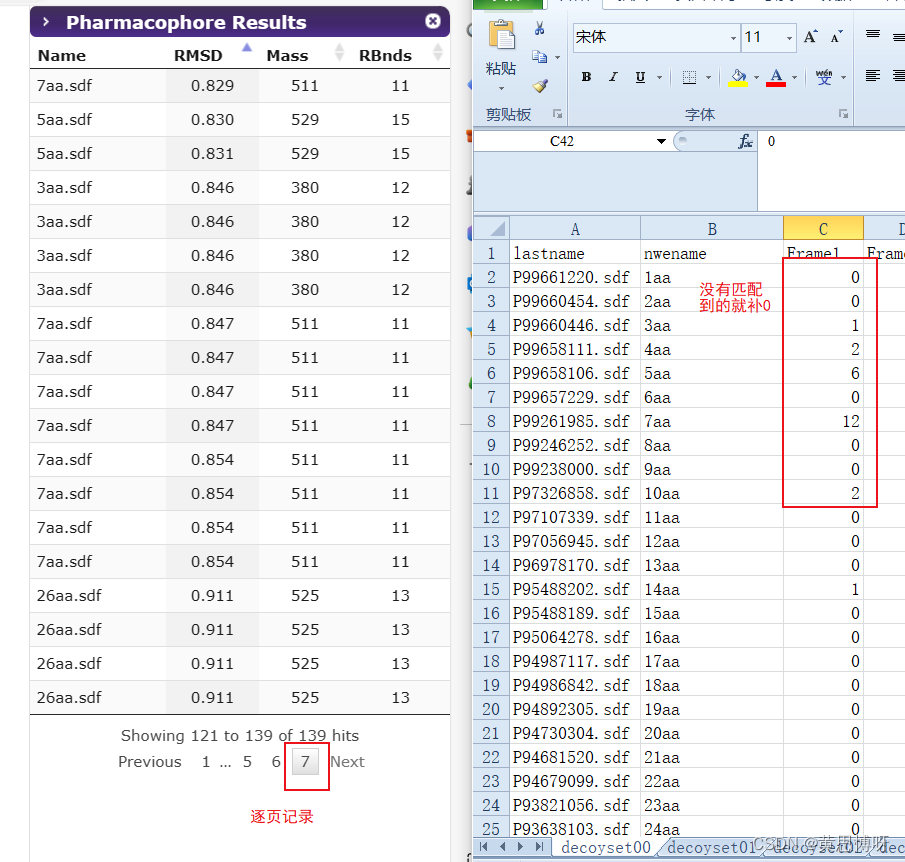
将每页表格中出现的构象逐一增加到xlsx表格里,最后对没有匹配到的构象赋值0,完成这一帧模型和这50个诱导剂化合物数据集的药效团检索:
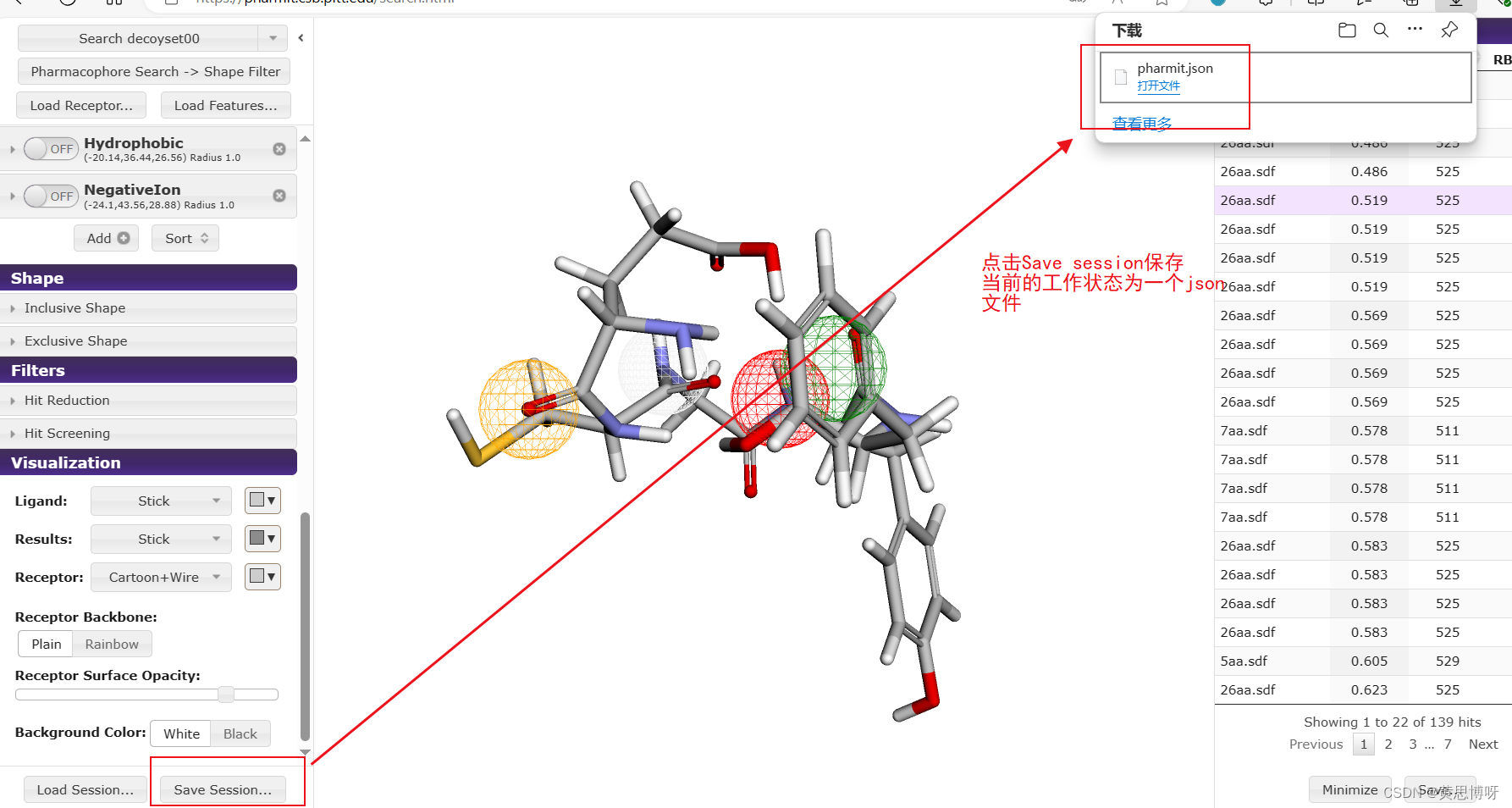
5、保存当前的记录(存档):
点击Save Session记录当前的状态为一个json文件:
打开一个空白的pharmit操作页面,点击Load Session导入之前保存的json文件,就会回到存档位置:
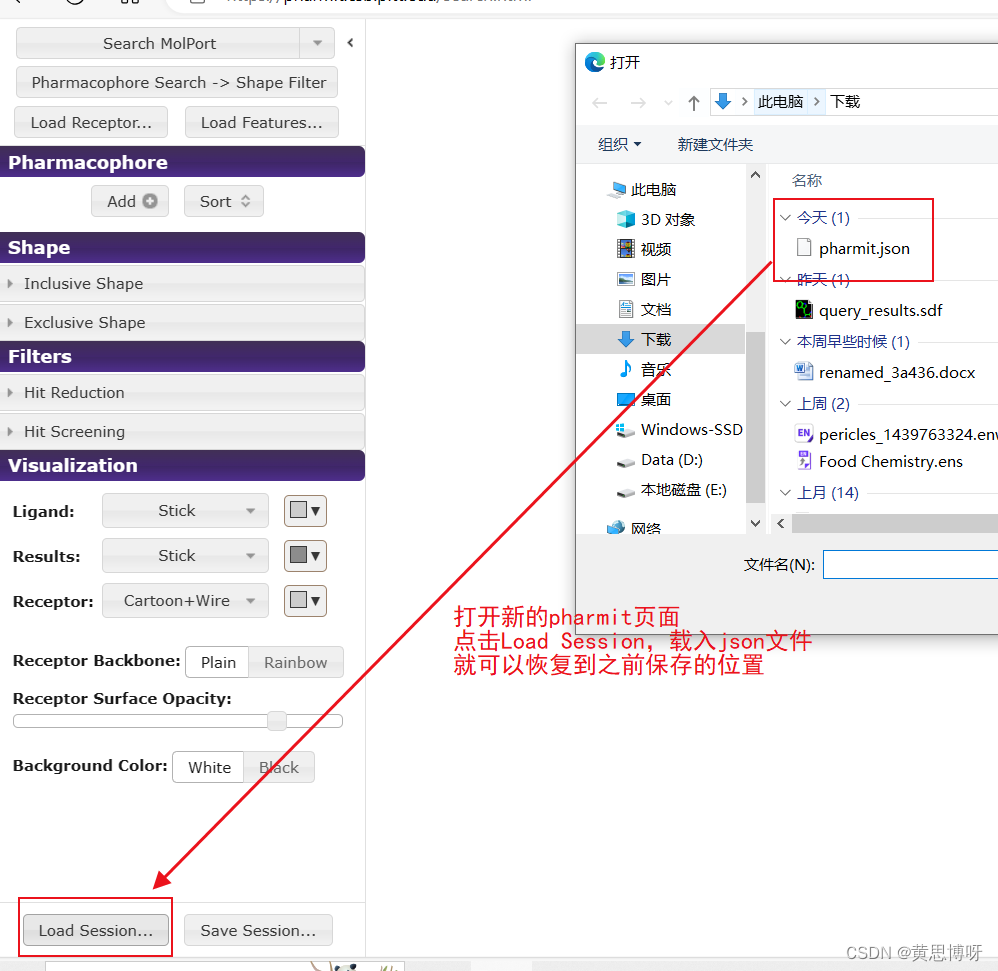
结果如下图,需点击Search decoyset00,重新开始匹配:
这篇关于在pharmit里匹配药效团的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







