本文主要是介绍Cell Biolabs丨艾美捷丨细胞迁移试验研究,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
Cell Biolabs/CytoSelect™ 24孔细胞迁移试验,12µm细胞迁移是一个高度整合的多步骤过程,协调胚胎形态发生,组织修复和再生。它在癌症、精神疾病和精神疾病的进展中起着关键作用发育迟缓、动脉粥样硬化和关节炎。细胞对迁移促进剂的初始反应是向引诱剂的方向J化和延伸突起;这些突起可以由大而宽的片状足或穗状丝状足。
在这两种情况下,这些突起都是由肌动蛋白驱动的聚合反应可以通过细胞外基质(ECM)粘附或细胞间相互作用来稳定(通过跨膜受体)。
细胞生物实验室的细胞选择™ 细胞迁移分析试剂盒采用聚碳酸酯膜插入物(12µm孔径)来测定细胞的迁移特性。试剂盒中含有足够的试剂,可用于对12个样本进行评估。12µm的孔径最适合研究缓慢移动的细胞或具有大的,如原代星形胶质细胞。
Cell Biolabs:细胞学试剂品牌,为生物技术和研究类客户提供基于细胞功能研究以及疾病等方面的技术和服务。主要涵盖细胞研究、细胞信号通路和蛋白质生物学、代谢研究、病原体和毒素、干细胞研究等领域。

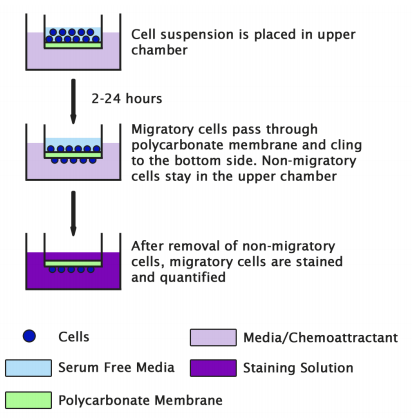
Cell Biolabs该实验化验原理:
细胞选择™ 细胞迁移分析试剂盒包含聚碳酸酯膜插入物(12µm孔尺寸)在24孔板中。该膜作为一个屏障,用于区分迁移细胞和非迁移细胞。迁移细胞能够将突起延伸至化学吸引物(通过肌动蛋白)细胞骨架重组)并最终通过聚碳酸酯膜的孔。最后,将细胞从膜顶部移除,并对迁移细胞进行染色和染色量化。
Cell Biolabs 相关研究方案:
1. CBA-107: CytoSelect™ 24-Well Cell Migration Assay (12µm, Fluorometric)
2. CBA-100-C: CytoSelect™ 24-Well Cell Migration and Invasion Assay (8µm, Colorimetric)
3. CBA-100-COL: CytoSelect™ 24-Well Cell Haptotaxis Assay (Collagen I, Colorimetric)
4. CBA-100-FN: CytoSelect™ 24-Well Cell Haptotaxis Assay (Fibronectin, Colorimetric)
5. CBA-100: CytoSelect™ 24-Well Cell Migration Assay (8µm, Colorimetric)
6. CBA-101: CytoSelect™ 24-Well Cell Migration Assay (8µm, Fluorometric)
7. CBA-102: CytoSelect™ 24-Well Cell Migration Assay (5µm, Fluorometric)
8. CBA-103: CytoSelect™ 24-Well Cell Migration Assay (3µm, Fluorometric)
9. CBA-106: CytoSelect™ 96-Well Cell Migration Assay (8µm, Fluorometric)
10. CBA-110: CytoSelect™ 24-Well Cell Invasion Assay (Basement Membrane, Colorimetric)
11. CBA-120: CytoSelect™ 24-Well Wound Healing Assay (Light Microscopy)
12. CBA-125: Radius™ 24-Well Cell Migration Assay (Microscopy)
13. CBA-130: CytoSelect™ 96-Well Cell Transformation Assay (Soft Agar Colony Formation)
这篇关于Cell Biolabs丨艾美捷丨细胞迁移试验研究的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!






