本文主要是介绍Ebba Biotech 艾美捷Carbotrace:检测碳水化合物结构,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
Carbotrace:
一种用于检测碳水化合物结构的荧光示踪分子,为植物中碳水化合物结构的解剖定位、生物基材料和生物燃料中多糖的非还原性成分的分析提供了方法,同时,Ebba Biotech 艾美捷Carbotrace也可区分碳水化合物中不同类型的糖苷键。
Ebba Biotech 艾美捷 Carbotrace 630–用于碳水化合物光谱成像的橙色光示踪分子光学追踪可以直接识别碳水化合物。为植物中碳水化合物结构的解剖学绘图和生物基材料和生物燃料中多糖的非破坏性成分分析提供了手段。
Ebba Biotech 艾美捷 Carbotrace 630使用示例:
样品中纤维素含量的测量
植物组织中纤维素的解剖学定位
要激发Carbotrace 630的荧光,从而识别样品中的结构,请使用任何类型的荧光显微镜设备(共焦或荧光显微镜)或具有荧光检测能力的分光光度计。
可使用标准PI(碘化丙啶)、Cy3、TxRed、mCherry或Cy3在630nm处检测Carbotrace 630的发射。5.滤波器组和激励使用488或514 nm激光线实现。光谱允许在600-650 nm的检测范围和458-514 nm的激发范围内进行自定义设置。
Ebba Biotech Carbotrace相关研究方案:
名称 cat# 荧光 规格
Carbotrace Mix&Try C5-M&T 以下5种荧光染料测试装,每支10ul 50 uL
Carbotrace 480 C480-50 Ex: 405-458 nm, Em: 470–550 nm, Emax = 480 nm 50 uL
Carbotrace 480 C480-100 Ex: 405-458 nm, Em: 470–550 nm, Emax = 480 nm 100 uL
Carbotrace 480 C480-150 Ex: 405-458 nm, Em: 470–550 nm, Emax = 480 nm 150 uL
Carbotrace 480 C480-200 Ex: 405-458 nm, Em: 470–550 nm, Emax = 480 nm 200 uL
Carbotrace 520 C520-50 Ex: 405-488 nm, Em: 500–600 nm, Emax = 520 nm 50 uL
Carbotrace 520 C520-100 Ex: 405-488 nm, Em: 500–600 nm, Emax = 520 nm 100 uL
Carbotrace 520 C520-150 Ex: 405-488 nm, Em: 500–600 nm, Emax = 520 nm 150 uL
Carbotrace 520 C520-200 Ex: 405-488 nm, Em: 500–600 nm, Emax = 520 nm 200 uL
Carbotrace 540 C540-50 Ex: 430-500 nm, Em: 530-600 nm, Emax = 540 nm 50 uL
Carbotrace 540 C540-100 Ex: 430-500 nm, Em: 530-600 nm, Emax = 540 nm 100 uL
Carbotrace 540 C540-150 Ex: 430-500 nm, Em: 530-600 nm, Emax = 540 nm 150 uL
Carbotrace 540 C540-200 Ex: 430-500 nm, Em: 530-600 nm, Emax = 540 nm 200 uL
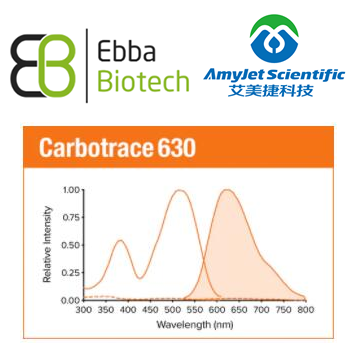
Carbotrace 630 C630-50 Ex: 458-514 nm, Em: 600-650 nm, Emax = 630 nm 50 uL
Carbotrace 630 C630-100 Ex: 458-514 nm, Em: 600-650 nm, Emax = 630 nm 100 uL
Carbotrace 630 C630-150 Ex: 458-514 nm, Em: 600-650 nm, Emax = 630 nm 150 uL
Carbotrace 630 C630-200 Ex: 458-514 nm, Em: 600-650 nm, Emax = 630 nm 200 uL
Carbotrace 680 C680-50 Ex: 530-565 nm, Em: 600-800 nm, Emax = 680 nm 50 uL
Carbotrace 680 C680-100 Ex: 530-565 nm, Em: 600-800 nm, Emax = 680 nm 100 uL
Carbotrace 680 C680-150 Ex: 530-565 nm, Em: 600-800 nm, Emax = 680 nm 150 uL
Carbotrace 680 C680-200 Ex: 530-565 nm, Em: 600-800 nm, Emax = 680 nm 200 uL
这篇关于Ebba Biotech 艾美捷Carbotrace:检测碳水化合物结构的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







