本文主要是介绍iMeta | 西悉尼大学刘洪伟/张海洋-温暖和干燥环境选择基因组较小的细菌,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
点击蓝字 关注我们
温暖和干燥环境选择基因组较小的细菌

原文链接DOI:https://doi.org/10.1002/imt2.70
研究论文
●2023年1月3日,澳大利亚西悉尼大学刘洪伟和张海洋组在iMeta在线发表了题为“Warmer and drier ecosystems select for smaller bacterial genomes in global soils”的文章。
● 本研究使用联合物种分布模型来量化环境对物种发生的影响。研究表明,增加更温暖和更干燥的生态系统的覆盖可能会通过减少基因组大小来简化细菌基因组。
● 第一作者:刘洪伟、张海洋
● 通讯作者:张海洋 (haiyang.zhang@westernsydney.edu.au)
● 合作作者:Jeff Powell、Manuel Delgado-Baquerizo、王军涛、Brajesh Singh
● 主要单位:澳大利亚西悉尼大学霍克斯伯里农学院、河北大学生命科学院、西班牙塞维利亚自然资源和农业生物学研究所 (IRNAS)、西班牙巴勃罗·德·奥拉维德大学、澳大利亚西悉尼大学全球陆地创新中心
亮 点
● 联合物种分布模型用于量化环境对细菌物种出现的影响
● 温暖干燥的环境有利于基因组较小的细菌
● 较高的土壤肥力(例如,磷)有利于基因组较小的细菌
摘 要
细菌基因组大小反映了细菌进化过程和代谢生活方式,对微生物群落组装和生态系统功能具有影响。然而,为了了解基因组介导的微生物对环境选择的反应程度,我们需要研究观察基因组大小沿着代表土壤细菌通常遇到的不同条件的环境梯度分布。在这项研究中,我们使用从全球237个地点收集的表层土壤,并使用细菌群落分析分析了环境条件(例如土壤碳和养分、干旱、pH值和温度)如何影响群落水平的土壤细菌发生和基因组大小。我们使用联合物种分布模型来量化环境对物种发生的影响,发现干旱是基因组大小的主要调节因子,温暖和干燥的环境选择基因组较小的细菌。干旱引起的对细菌生长的生理限制(例如,细胞代谢缺水)可能导致了这些相关性。这一发现表明,未来随着生态系统变的更加温暖和干旱,基因组较小的细菌将具有相对更好的生存力。
视频解读
Bilibili:https://www.bilibili.com/video/BV1Ax4y1G78v/
Youtube:https://youtu.be/8WBau3UusIo
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science
全文解读
正文
基于功能性状的研究深刻改变了生态学,并为我们对许多分类群的生态学理解(例如,群体组装)提供了可证明的价值。然而,关于微生物特性重要性的知识落后于其他生物(例如植物)。特别是,我们对全球微生物特征的生物地理模式以及它们在环境选择下的程度知之甚少。在微生物特征中,基因组大小是预测微生物生理学、它们与其他微生物和环境的生态关系及其进化历史的基础。这是因为 (i) 基因组大小与其他与生命史策略相关的细菌特征有关,例如生长速度和对极端条件的耐受性,(ii) 微生物类群中基因组大小的变化可以反映进化事件,例如作为可能与新物种、功能或生活方式的出现相关的基因组扩展和减少,以及 (iii) 具有大基因组的生物需要更多的资源来生长和繁殖,并且可能具有更受限制的生态分布(大基因组约束假设)。换句话说,较小的细菌基因组需要较少数量的AGCT,并且它们在营养有限(例如,N和P)环境中更有优势。因此,环境中基因组特征的分布可能表现出反映营养状况的独特模式。
土壤是一个复杂且异质的生态系统,涉及动态相互作用的细菌群落,这些细菌群落在调节陆地养分循环和植物生产力方面发挥着关键作用。它的功能严格取决于水、光和生物多样性等关键因素,这些因素为人类提供了生计。最近的研究调查了微生物基因组大小对生态系统类型、营养策略和氧气使用的响应,但对土壤微生物基因组及其对大空间梯度环境因素的响应的研究仍然有限。目前土壤微生物基因组大小的生物地理学不确定性主要有两个原因。首先,调查通常关注单一环境因素与基因组大小之间的关系,但要确定最重要的环境因素则需要进行多因素调查。例如,最近的研究仅调查了微生物基因组大小与环境的相互作用,以对变暖和养分可用性的响应。其次,这些研究仅调查了这些环境梯度的一小部分,并且缺乏对涵盖全球范围内气候、土壤和/或植被的生态系统的研究。理论上,在扩大/全球范围的环境调查中可以存在权衡(影响发生在环境梯度的两端)和单向模式(影响发生在梯度的一端)。这种对微生物物种发生的环境影响可以是积极的、消极的或中性的(图 1A),并且影响的程度因细菌物种而异。为此,我们提出了环境影响与细菌基因组大小之间的四种关系:(i)权衡(图 1B),(ii-iii)单向影响(图 1C,D),以及(iv)无相关性(图 1E)。在这项研究中,我们旨在研究基因组大小是否可以调节环境梯度对全球范围内细菌出现的影响。我们假设条件较差的环境,如较干燥的土壤(图 1F)、较温暖的气候(图 1G)和土壤养分的可用性降低(图 1H),有利于基因组较小的细菌物种的出现。这些假设基于大基因组约束假设,以及之前的发现,即营养限制/干旱引起的生长生理限制(例如细胞代谢缺水)可能导致基因组精简(例如基因丢失)。
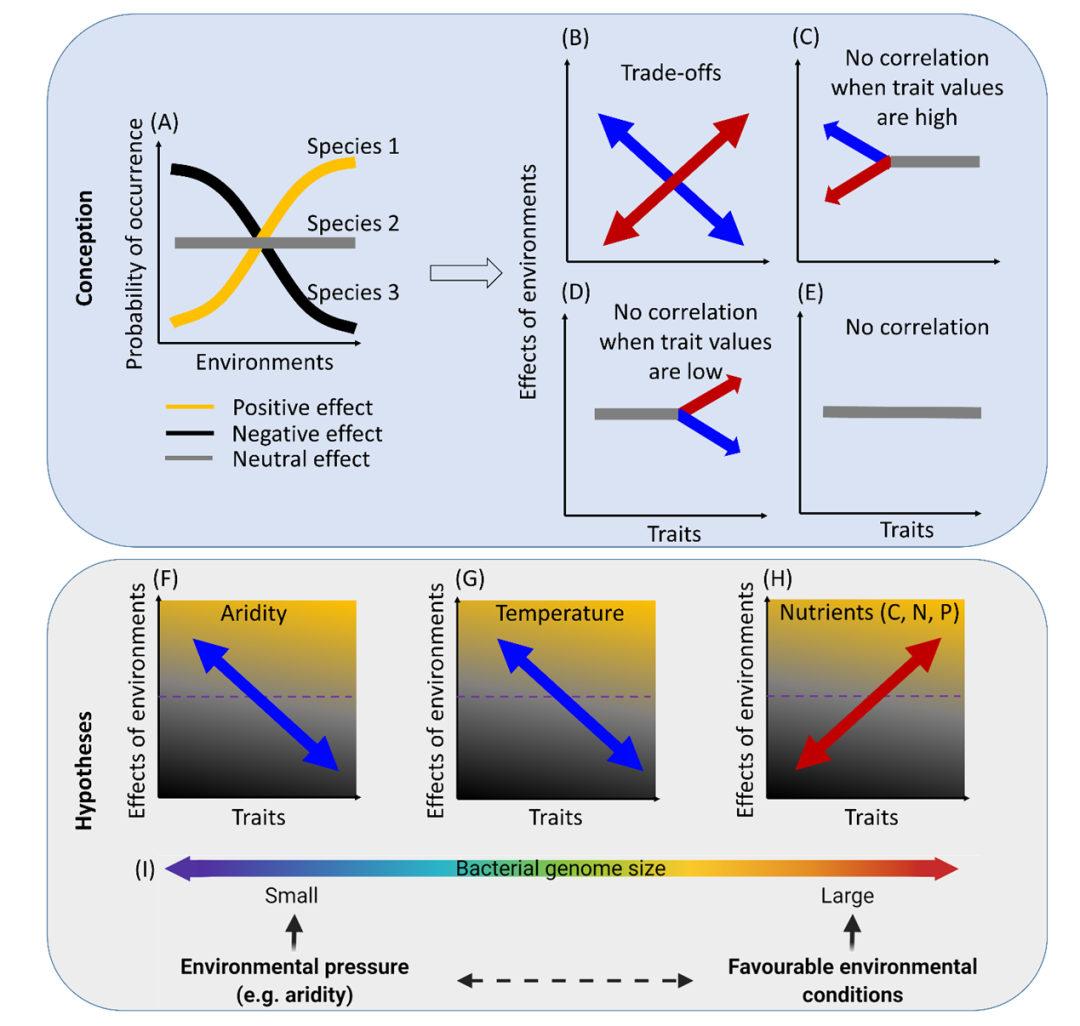
图 1. 概念和假设
细菌出现概率对环境梯度的生态响应:正相关(黄色)、负相关(黑色)和中性(灰色)(A)。正(或负)效应大小表明当特定环境条件增加时,细菌倾向于以更高(或更低)的概率出现。效应大小是细菌出现概率与环境条件之间关系的斜率,由联合物种分布模型的β参数计算。我们提供了四种微生物特征与环境影响大小之间的相关性。这些是 (i) 性状影响是双向的权衡:环境梯度对物种出现概率的影响正向(红色)或负向(蓝色)沿着性状梯度(B)移动,(ii &iii)性状仅在环境梯度的一端表现出效果,当性状值低(C)或高(D)时仅具有单向效果;(iv) 所有性状水平 (E) 都没有环境影响。我们假设从有利的土壤条件转变为压力环境(即更温暖和更干燥的气候)有利于土壤中具有小 GS 的细菌。具体而言,我们假设营养物质(碳、氮和磷)与细菌基因组大小 (F) 呈正相关,而年最高温度 (MAXT) (G) 和干旱 (H) 与细菌基因组大小呈负相关。总体而言,我们假设压力条件会选择基因组大小较小的细菌 (I)。
为了检验上述假设,我们分析了从全球237个地点收集的土壤样本的细菌群落(图 S1),涵盖不同的生态系统类型(森林、草地和灌木丛)和不同的环境梯度,如土壤干旱、养分和气温。土壤性质变化范围大,土壤pH值从4.04到9.21,土壤C从0.15%到34.77%,土壤N从0.02%到1.57%,土壤P从75.10到4111.04 mg P kg- 1土。这些采样点的年平均温度梯度为 -11.4 至 26.5ºC,年平均降水量为67至3,085毫米。使用16SrRNA基因扩增子测序检测到总共135,175个土壤细菌特征(2,315个属,QIIME2软件分析的序列变异,http://qiime2.org/),并提取了在属水平注释的细菌特征(参见补充方法,图S2、S3和S4)。从属特征表中删除那些未识别和较少识别的细菌属,例如细菌培养克隆。为了评估在属水平上汇总这些数据是否合适,我们调查了细菌性状数据库中的细菌基因组大小,其中包括这些属的可培养和不可培养细菌物种的数据。我们发现大多数 (95%) 细菌属 (n = 2,238) 中的物种具有较低的变异系数 (CV) (< 20%)(图 S2,CV 定义为标准偏差与平均值的比率对于某个属)。进一步的阈值敏感分析表明,对于 CV 低于 11% 的细菌属,细菌基因组大小估计是稳健的(该阈值是根据敏感性分析选择的,详情见图 S3)。因此,我们将一个属内的观察结果的 CV 阈值设置为 11%,以选择细菌属以纳入后续分析,其中保留了1,237个细菌属。然后,我们将我们的全球土壤微生物组数据与这些细菌属相匹配,得到143个细菌属,其基因组大小值从1.58 Mbp(立克次体)到16.04 Mbp(小囊藻)(图 S4)。我们选择的属包括丰富的和稀有的属(图 S5)。
然后,我们通过将数据集与联合物种分布模型 - 物种群落分层建模 (HMSC)(图 S6)相匹配,研究了环境梯度与细菌基因组大小之间的关系。在模型拟合和评估之后,我们提取了效应大小,即细菌出现概率与环境条件之间关系的斜率,这是来自 HMSC的beta参数,后验概率至少为95%。对于基因组大小与环境对细菌出现概率的影响程度之间的相关性,我们进行了系统发育最小二乘回归以解释属间的系统发育自相关,因为细菌基因组大小与其进化历史有关。我们发现距赤道的距离(绝对纬度)对土壤中的基因组大小分布有很大影响(图 2A,R2 = 0.22,P < 0.00001),因为具有小基因组的细菌更有可能与纬度增加,而具有大基因组的细菌表现出相反的情况。这一结果与先前报道的地理模式一致,其中细菌群落的平均基因组大小随着距赤道距离的增加而减小。
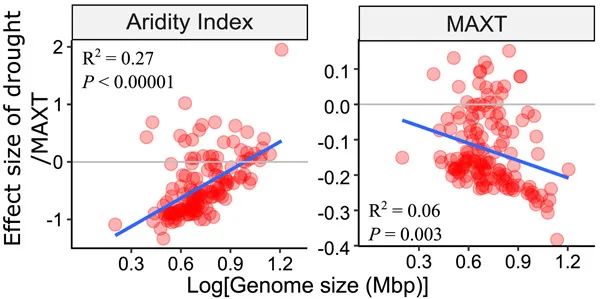
重要的是,我们发现细菌基因组大小与干旱显着相关(图 2B,R2 = 0.27,P < 0.00001),因为具有小基因组的细菌对增加的干旱表现出积极反应(对增加的干旱指数的消极反应)而细菌属大型基因组往往对干旱没有任何反应(与这些细菌相关的效应量接近于零)(图 2B)。这种具有小基因组的细菌选择趋势在不同的分类水平(例如目和门水平)上是一致的(图 S7)。我们的进一步分析表明,大型基因组细菌物种包含更多的基因(图 S6A,R2 = 0.97,P < 0.00001)(图 S8A),这表明干旱生态系统中基因组减少可能导致细菌基因组简化.一致地,最近的一项研究还报告了细菌群落重量平均基因组大小与平均年降水量之间的正相关关系。然而,在他们的研究中,尚不清楚这种正相关是由增加可用水量正向选择基因组较大的细菌,还是因增加可用水量而排除基因组大小较小的细菌,或两者兼而有之。我们的结果支持第二种情况,即基因组较小的细菌物种比那些基因组较大的物种对增加的水可用性有更多的负面反应。同样,温暖的气候,如较高的平均最高温度 (MAXT) 所示,通过排除基因组的大细菌,有利于基因组小的细菌的出现(图 2C,R2 = 0.06,P = 0.003)。这一结果与之前报告中的发现一致,并得到我们进一步分析的支持,该分析表明大型基因组大小的细菌物种具有较低的最适温度(图 S8B,R2 = 0.07,P = 0.001)。这意味着与基因组小的细菌相比,升高的温度可以对基因组大的细菌产生更高的选择压力。
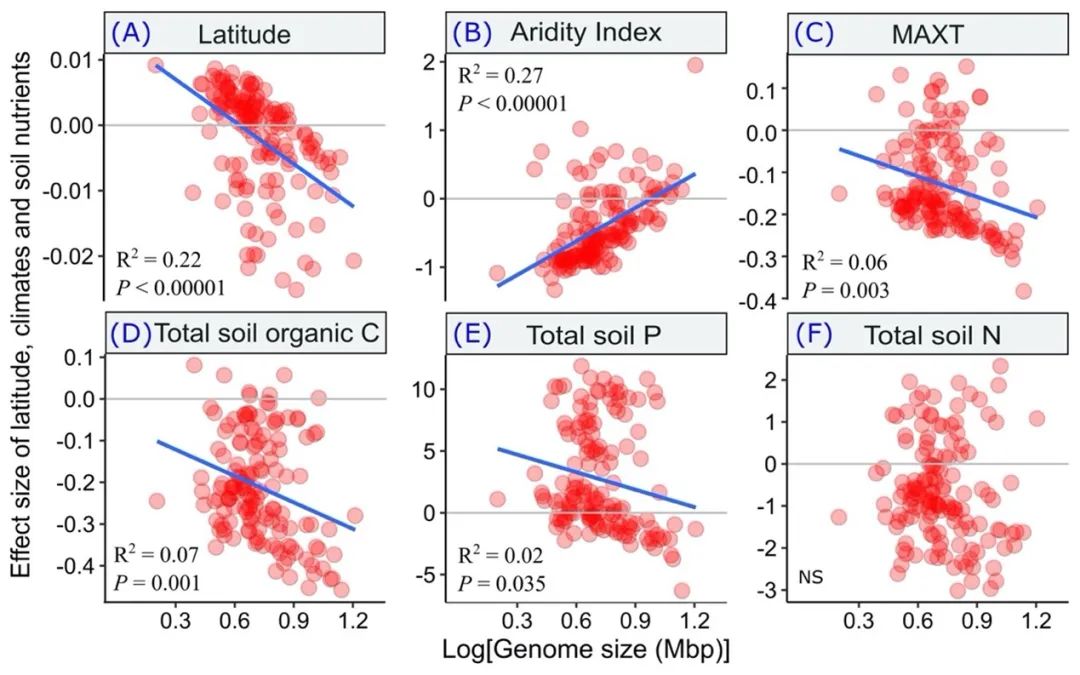
图 2. 细菌基因组大小(对数转换;Mbp)与环境条件对细菌发生的影响之间的相关性
Y轴表示环境条件的影响大小,它是细菌发生概率与环境条件之间关系的斜率(β参数),根据物种群落的层次模型 (HMSC) 计算得出。正(或负)效应大小表明当特定环境条件增加时,细菌属倾向于以更高(或更低)的概率出现。蓝色实线表示相关性显着(P < 0.05),并且考虑到细菌属之间的系统发育相关性。干旱指数反映了土壤水分的有效性(即年平均降水量/蒸散量)。MAXT:年平均最高气温。
我们还发现细菌基因组大小与土壤碳(C,图 2D,R2 = 0.07,P = 0.001)和 P(图 2E,R2 =0.02,P = 0.035)浓度微弱但显着相关,在该细菌中小基因组对增加土壤 C 表现出较少的负面反应,或对增加土壤 P 表现出更多积极反应,这表明较高的土壤肥力有利于基因组较小的细菌。土壤氮没有表现出显着的相关性(图 2F)。虽然与我们的假设(基于大基因组约束理论)相矛盾,但我们的结果得到了先前研究的支持,在这些研究中,土壤养分(例如 P)的富集降低了草原土壤中群落水平细菌的平均基因组大小。这些发现表明,较大的基因组大小的细菌物种可以在资源稀缺但多样化的环境中占主导地位,并且在土壤中的定植和竞争方面,生长缓慢的土壤细菌几乎没有受到惩罚。在未来的研究中,研究基因组大小对土壤养分可用性的反应如何与其 r/K 策略相关联也很重要。例如,之前的研究表明,通过添加养分,来自富养(r 策略)厚壁菌门的土壤细菌的相对丰度增加,而放线菌等贫养(K 策略)细菌的丰度减少。一致地,我们发现厚壁菌门的基因组相对较小,而放线菌门的基因组较大(图 S5),因此,我们的结果(较高的土壤肥力有利于基因组较小的细菌)可以用 r/K 选择理论来解释。我们还发现基因组大小不影响细菌出现概率对土壤 pH 值和年平均最低温度变化的敏感性(图 S9)。相比之下,细菌基因组大小与净初级生产(NPP,植物或其他初级生产者保留的碳量)和紫外线 (UV) 显着相关,因为具有大基因组的细菌与增加 NPP 和紫外线呈正相关,而细菌具有小基因组的属往往与 NPP 和紫外线没有任何相关性(图 S9)。增加 NPP 有利于具有大基因组的细菌与我们的结果一致,即变暖和干燥的生态系统选择较小的细菌基因组,因为 NPP 在压力环境(例如干旱)下通常较低。对紫外线强度的积极反应也表明大基因组大小的细菌对强紫外线更耐受。这些发现的机制尚不清楚,需要未来的研究进行深入调查。
我们承认群落加权特征均值 (CWM),即每个地点的物种特征值的平均值,按物种丰度加权,也常用于研究特征-环境关系。然而,我们没有观察到 CWM 基因组大小和环境梯度之间的显着模式(图 S10),这可能突出了联合物种分布方法在检测土壤细菌对环境变化的反应方面的优势。最近的研究表明,基于模型的方法在结果准确性和统计能力方面优于基于CWM的方法。
总的来说,我们的研究提供了新的证据,表明与干旱和气候变暖相关的环境压力有利于土壤中基因组较小的细菌。干旱引起的生长生理限制(例如细胞代谢缺水)可能导致了这些相关性。细菌群落的变化会导致碳封存等土壤生态过程的变化。例如,之前的研究表明,具有小基因组的细菌与高碳利用效率(CUE,即净碳增益与总碳同化的比率,控制碳封存的关键因素)相关。干旱胁迫会降低植物对碳的同化和对土壤微生物的利用。转向以小基因组大小为主的群体对于增加群体级别的CUE并帮助生态系统在压力条件下维持功能非常重要。偏爱基因组较小的细菌物种也可能对微生物功能和恢复力的冗余产生影响。土壤微生物构成了一个复杂的基因库;减少或丢弃多余代谢负担的基因可能是微生物在压力下生存的一种策略。现在,我们正面临比以往任何时候都更快的气候变化。尽管此类微生物变化对生态系统功能的影响未知,但未来的气候变化研究和政策制定应考虑土壤细菌及其功能特征,因为它们在土壤固碳和维持生态系统功能方面发挥着重要作用。我们的研究还表明,较低的土壤肥力(土壤C和P)和较高的紫外线水平有利于基因组较大的细菌,这些细菌可以在资源稀缺的土壤中占主导地位,但对缓慢生长的细菌没有重大限制。这些发现指向支持全球土壤细菌群落中核苷酸选择的基本机制。这些知识对于预测未来气候系统中生物地球化学反馈的努力也至关重要,在未来气候系统中,干旱事件预计会更频繁地发生,并在全球范围内持续更长时间。我们的研究通过从公开可用的数据库中获取基因组大小数据,在属水平上进行细菌分析。这种方法在检验我们的假设方面是可靠的,它提供了一个有用的工具来研究基于扩增子测序的土壤基因组,该测序仍然广泛用于微生物组研究。由于16S rRNA基因扩增子测序在分析细菌群落方面的固有局限性(例如16S rRNA基因是保守的,它可能掩盖细菌群落内的某些性状变异),因此它的不足之处在于只能将一部分土壤细菌群落纳入分析。未来的研究有必要通过确定跨环境梯度的宏基因组的基因组大小来调查土壤和其他生态系统(例如海洋生态系统)中的细菌群落组装来验证我们的发现。
作者贡献
刘洪伟、张海洋、Brajesh Singh和Manuel Delgado-Baquerizo构思了这个想法,张海洋、刘洪伟、Jeff Powell和王军涛进行数据分析。刘洪伟和张海洋撰写了这篇论文,所有作者都对手稿的修订做出了重大贡献。
致谢
这项研究得到了澳大利亚研究委员会 (DP190103714、DP210102081、FT190100590) 的支持。M.D-B.感谢西班牙科学与创新部对由MCIN/AEI/10.13039/5 资助的研发项目PID2020-115813RA-I00的支持M.D-B.还得到了欧洲区域发展基金 (EFR) 和安达卢西亚军政府经济转型、工业、知识和大学委员会的一个项目的支持(FEDER安达卢西亚2014-2020主题目标“01 - 加强研究,技术发展和创新”)与研究项目P20_00879 (ANDABIOMA) 。
数据可用性声明:
本研究中使用的16S rDNA数据存储在Doi:10.6084/m9.figshare.5611321。补充材料(图形、表格、脚本、图形摘要、幻灯片、视频、中文翻译版本和更新材料)可以在https://doi.org/10.1002/imt2.70 或iMeta Science http://www.imeta.science/ 中找到
引文格式:
Hongwei Liu, Haiyang Zhang, Jeff Powell, Manuel Delgado‐Baquerizo, Juntao Wang, and Brajesh Singh. 2022. “Warmer and drier ecosystems select for smaller bacterial genomes in global soils.” iMeta. e70. https://doi.org/10.1002/imt2.70
作者简介

刘洪伟(第一作者)
● 博士毕业于澳大利亚昆士兰大学
● 研究方向为生物防控,植物微生物互作用和昆虫肠道微生物。他致力于揭示农业微生物与植物,昆虫之间相互作用机制以及利用微生物促进可持续发展农业。目前以第一作者身份在国际较高影响力期刊Trends in Ecology & Evolution, Trends in Plant Science, New Phytologist,Geoderma,Soil & Tillage Research, Biology and Fertility of Soils和iMeta 上发表15篇论文。论文总引用1700余次。目前担任Journal of Sustainable Agriculture and Environment等期刊编委。担任Nature Ecology and Evolution, Nature Food, Ecology Letters, Trends in Plant Science等二十几种期刊和基金审稿人。目前承担澳洲科学委员会(ARC)两项科研项目的研究(DP210102081,DP190103714)

张海洋(第一作者兼通讯作者)
● 河北大学
● 从事生物地球化学和植物-真菌相互作用研究,重点是共生微生物介导的地下生态过程(例如,碳和养分循环)。在博士和博士后期间主要从事植物-微生物相互作用研究,尤其关注菌根真菌和豆科固氮过程及其对全球变化的响应。利用稳定同位素示踪标记,微生物高通量分子测序和二次离子质谱分析技术,重点研究了全球变化背景下的根系性状介导的植物间养分传递,及菌根真菌性状介导的植物-土壤-微生物互作下的生物地球化学循环。在以下三方面取得进展:(1)揭示了菌根真菌介导的土壤养分循环,研发新的菌丝性状量化技术体系,提出菌丝生态化学计量学的概念框架,为跨物种和时空尺度研究菌根生态学提供了新思路; (2)评价了植物碳可利用性对菌根真菌养分传递的调控,利用稳定同位素技术深化了对植物-微生物互作机理的认识;(3)阐明了地下根系和菌丝性状介导的植物氮素传递,拓展了对地下功能性状介导的养分循环过程的理解。上述结果为研究共生微生物的生态功能提供了科学依据。近五年,作为第一作者发表研究论文10篇,包括在生态学或地学主流杂志Global Ecology and Biogeography, Geochimica et Cosmochimica Acta, Molecular Ecology, Ecology和Functional Ecology等发表。参与了3项国内研究课题和5项国际研究课题并在多个国际会议作学术报告。研究成果受到国内外同行认可和关注,论文(28篇)SCI引用530余次,H指数为14
更多推荐
(▼ 点击跳转)
高引文章 ▸▸▸▸
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
▸▸▸▸
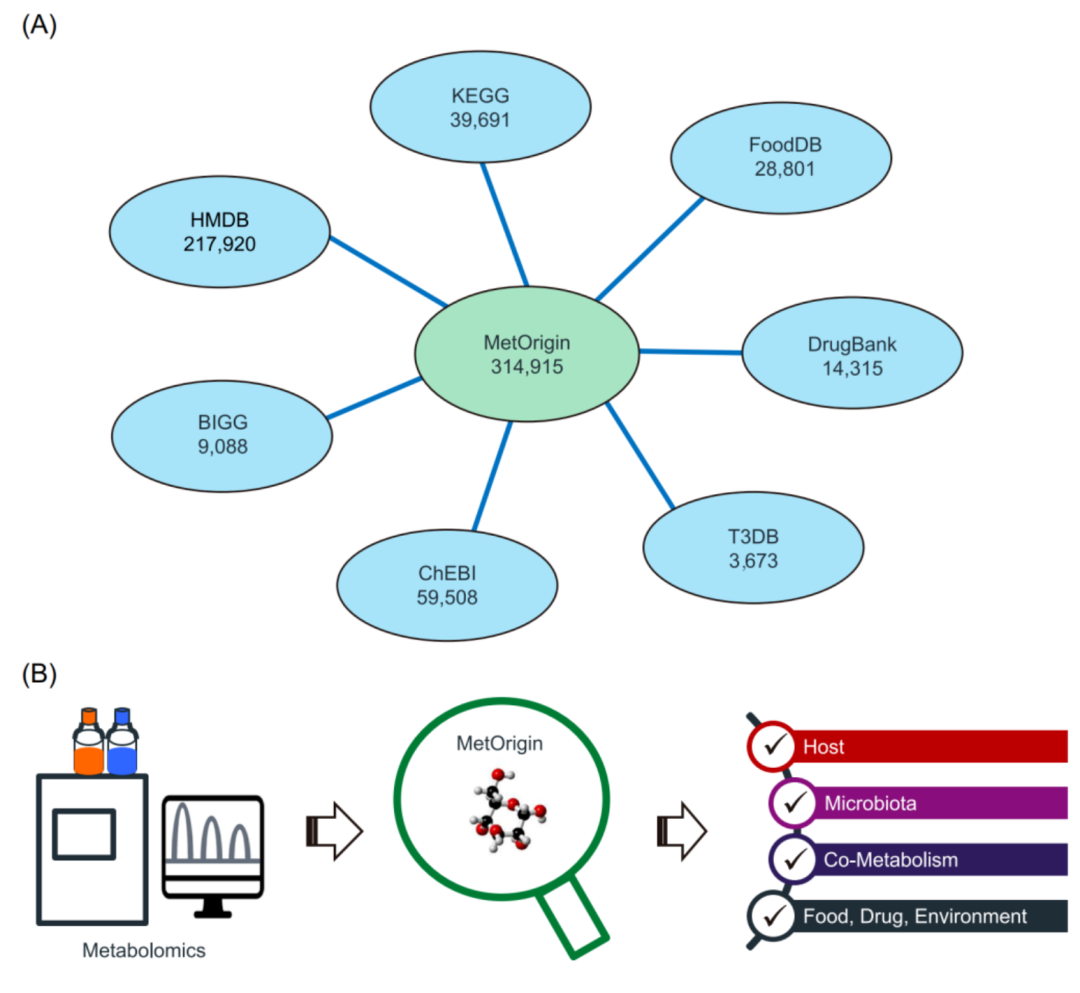
iMeta | 浙大倪艳组MetOrigin实现代谢物溯源和肠道微生物组与代谢组整合分析
▸▸▸▸
iMeta | 高颜值绘图网站imageGP+视频教程合集

第1卷第1期

第1卷第2期

第1卷第3期

第1卷第4期
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 15)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!
联系我们
iMeta主页:http://www.imeta.science
出版社:https://onlinelibrary.wiley.com/journal/2770596x
投稿:https://mc.manuscriptcentral.com/imeta
邮箱:office@imeta.science
这篇关于iMeta | 西悉尼大学刘洪伟/张海洋-温暖和干燥环境选择基因组较小的细菌的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!









