本文主要是介绍富集分析的原理与实现,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
一般做完差异分析都会做这一步,目的是找到差异基因富集到的通路,进而与生物学意义联系起来。具体的统计方法很简单,这篇笔记里面的代码可以从零搭建一个富集分析工具。
后台回复20211007获取本文的测试数据和代码,以及(单细胞)转录组分析中可能用到的GO KEGG富集分析代码(这部分本文不演示)。
关于Gene Ontology (GO), KEGG这些背景就不讲了,网上很多资料。
将富集分析中的问题抽象出来,其实就是下图的“摸球”问题。
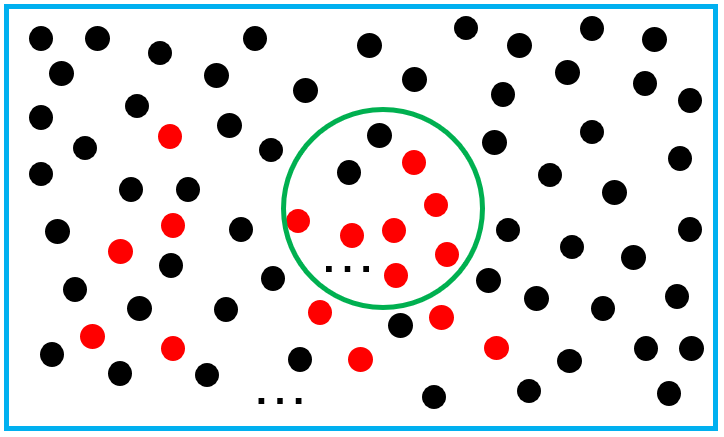
蓝色方框中的球是所有的基因【共N个】,在探究某个特定通路P时,通路里面涉及到的基因用红色表示【共M个】。绿色圆圈是一次摸球事件,用来表示做了一次差异分析得到的基因【共n个】,这些基因中,有属于通路P的(红色球)【共k个】,有不属于的(黑色球)。
用摸球问题中的语言再描述一次:袋子中共有黑球和红球N个,其中红球M个。某次抽样中,一共摸球n个,其中红球k个,问在这次摸球中,红球的占比是否显著高于袋子中红球的占比? (以前学摸球问题/超几何分布的时候,可能只求概率,没有进一步到这个统计检验)
回答这个问题,需要求出问题中这个事件的概率以及更极端事件的概率之和,也就是p值,小于0.05或者0.01就能认为是显著了。
1. 一个通路,计算p值
接下来以一个GO term (GO_extracellular_matrix_organization)为例,计算p值。 用的通路基因集是小鼠 GO BP的基因集,差异基因集是单细胞转录组分析中一个cluster的高表达基因
library(tidyverse)
library(clusterProfiler)gmt.df=read.gmt("Mm.c5.bp.v7.1.SYMBOL.gmt")
deg=read.table("test_deg.txt",header = T,sep = "\t",stringsAsFactors = F)
deg=deg[deg$gene %in% gmt.df$gene,]这种情况下,前面说的几个参数的值如下:
- 全部球的个数/全部基因数:
N=length(unique(gmt.df$gene)) - 全部红球的个数/通路基因集的基因数:
one.set=gmt.df[ gmt.df$term %in% c("GO_extracellular_matrix_organization") ,] M=length(one.set$gene) - 摸球数/差异基因数:
n=length(deg$gene) - 摸球中红球的个数/差异基因中属于这个通路的基因数:
k=sum(deg$gene %in% one.set$gene)
N M n k的值分别为: 23210, 271, 47, 6
求p值之前,先看一下满足N M n这三个参数的超几何分布,在不同的k值之下的概率:
df1=data.frame(x=1:47,y=dhyper(x=1:47, M, N-M, n))
df1%>%ggplot(aes(x,y))+geom_point()+geom_line()+geom_vline(xintercept=k,color="red",linetype=5)+ #这里k等于6,其作为阈值theme_bw()+theme(panel.grid = element_blank())dhyper()用来求概率,四个参数分别是:摸球中红球个数的向量,袋中红球数,袋中黑球数,摸球数
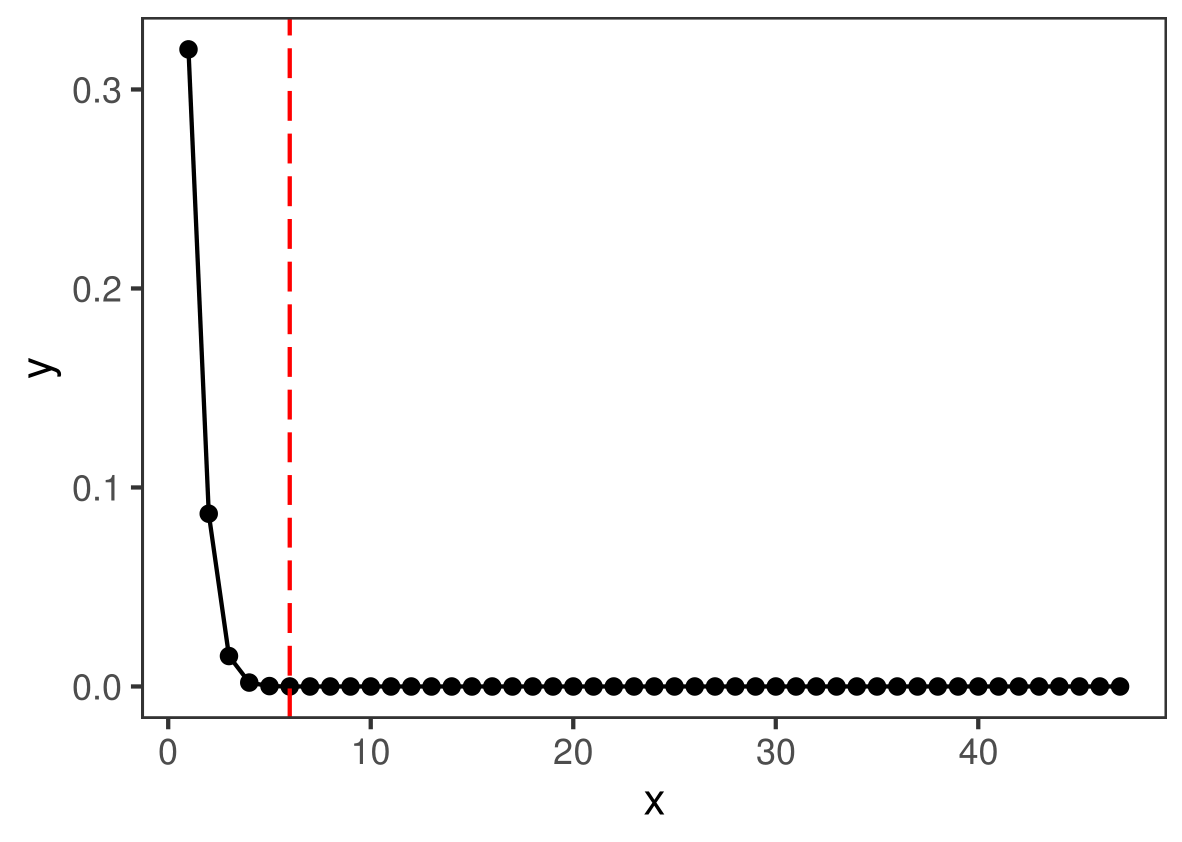
我们要计算的就是红线以及右边那些红球数对应的概率之和,如下:
> phyper(k-1,M, N-M, n, lower.tail=FALSE)
[1] 1.722659e-05当lower.tail=FALSE,计算的是P[X > x],即大于第一个参数的概率之和。上面的代码第一个参数写的是k-1,因为我们需要求k以及k右边的概率之和。
以上是对一个通路求p值
2. 多个通路,依次计算p值
如果是多个通路,需要循环操作,依次对每个通路进行富集分析。 下面的演示用到的差异基因集和GO BP基因集同上
分析哪些pathway?要满足两个条件:
- 通路里面基因的数量满足一定要求
- 至少和deg有基因交集
下面的代码就是对通路做过滤的
bp.stat=as.data.frame(table(gmt.df$term))
colnames(bp.stat)[1]="pathway"
bp.stat=bp.stat%>%filter(Freq >= 2 & Freq <= 2000)tmp.df=gmt.df
tmp.df$TF=tmp.df$gene %in% deg$gene
tmp.stat=as.data.frame(tmp.df %>% dplyr::group_by(term) %>% dplyr::summarize(counts=sum(TF)))
tmp.stat=tmp.stat%>%filter(counts > 0)
keep.pw=sort(intersect(bp.stat$pathway,tmp.stat$term))下面就是循环求p值了
N=length(unique(gmt.df$gene))
n=length(deg$gene)
term=c()
pvalue=c()for (i in keep.pw) {one.set=gmt.df[ gmt.df$term %in% i ,]M=length(one.set$gene)k=sum(deg$gene %in% one.set$gene)one.pvalue=phyper(k-1,M, N-M, n, lower.tail=FALSE)term=append(term,i)pvalue=append(pvalue,one.pvalue)
}my_go_res=data.frame(term=term,pvalue=pvalue)
my_go_res=my_go_res%>%arrange(pvalue)
到这会儿还没有结束,还差一个FDR
3. 计算矫正p值
FDR, False Discovery Rates。为什么要控制FDR,降低假阳性。 这里用到的是The Benjamini-Hochberg method
The Benjamini-Hochberg method
假设我们对10个通路做了富集分析,我们会先得到10个p值:
- 将这10个p值从小到大排序
- 从1到10给这些p值排序
- 最大的FDR adjusted p value(第10位)等于原来最大的那个p值
- 第9位的FDR adjusted p value等于这两个值中的较小值: ①前一位矫正的p值; ②当前未矫正的p值 * (p值总个数/当前位数)
- 重复第4步,直到第1位
代码如下:
fdr=c()
for (i in dim(my_go_res)[1]:1) {if (i==dim(my_go_res)[1]) {tmpfdr=my_go_res$pvalue[i]}else{tmpfdr=min(tmpfdr,my_go_res$pvalue[i] * (dim(my_go_res)[1] / i))}fdr=append(fdr,tmpfdr)
}
my_go_res$p.adj=rev(fdr)
到这儿富集分析的完整流程才算结束
4. 轮子有现成的
当然,这个算法已经非常常见了,clusterProfiler的enricher()就能够自定义基因集做富集分析。使用如下:
deg_gmt=clusterProfiler::enricher(deg$gene,TERM2GENE = gmt.df,minGSSize = 2,maxGSSize = 2000)
go_res=deg_gmt@result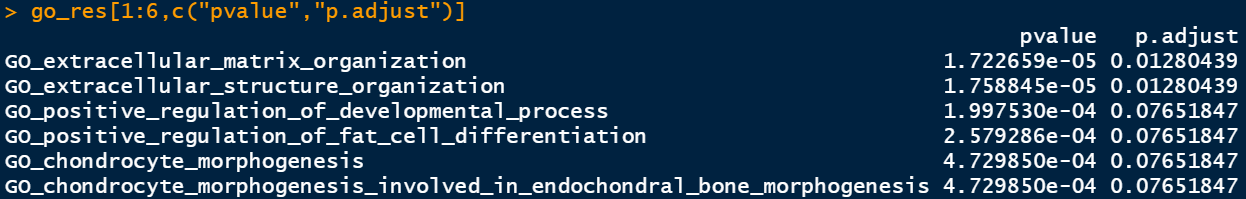
和上面的结果是一模一样的。
今天的内容就到这里,后台回复20211007获取本文的测试数据和代码,以及(单细胞)转录组分析中可能用到的GO KEGG富集分析代码(这部分本文不演示)。
ref
- 超几何分布检验(hypergeometric test):https://blog.csdn.net/linkequa/article/details/86491665
- 富集分析的p值是怎么算出来的?:公众号【YuLabSMU】
- R tips 富集分析及其p值在R中的计算:公众号【生信菜鸟团】
- False Discovery Rates, FDR, clearly explained:https://www.youtube.com/watch?v=K8LQSvtjcEo&t=909s&ab_channel=StatQuestwithJoshStarmer
因水平有限,有错误的地方,欢迎批评指正!
这篇关于富集分析的原理与实现的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!






