本文主要是介绍sc.pl.umap 画feature plot,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
今天有时间尝试测试了这个scanpy的feature plot,其实很简单,就是使用
sc.pl.umap(adata,color="gene name"), 但是这个地方就有一个问题,这个画出来的值是原始的基因值还是scale之后的,这个我得搞清楚
首先看使用例子,参考博客
https://www.jianshu.com/p/9c14c23755af
import scanpy as sc
import pandas as pd
from matplotlib.pyplot import rc_context
import matplotlib.pyplot as pl
sc.set_figure_params(dpi=1000, color_map = 'viridis_r')
sc.settings.verbosity = 1
sc.logging.print_header()## 加载数据
pbmc = sc.datasets.pbmc68k_reduced()
# inspect pbmc contents
pbmc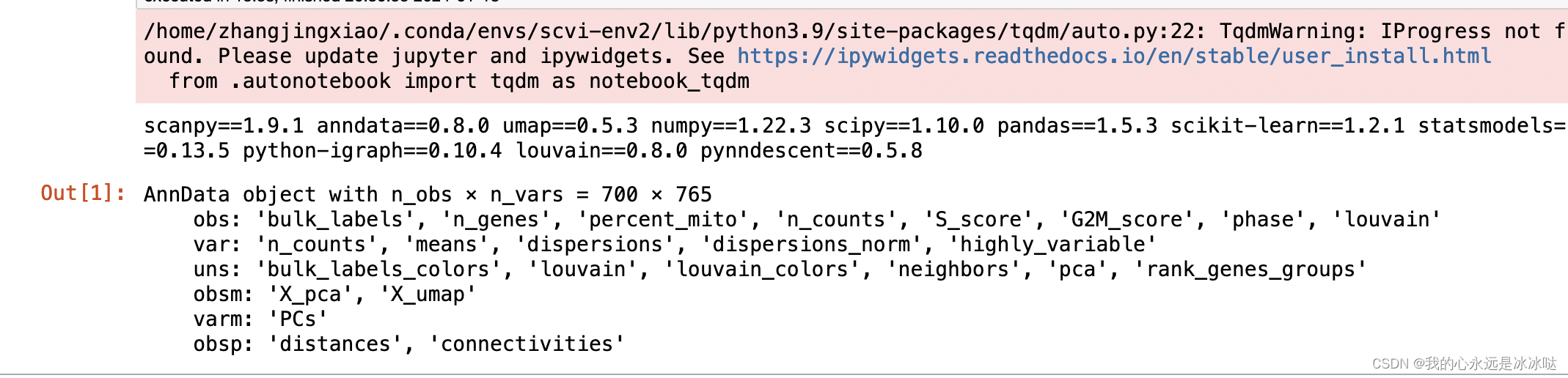
# rc_context is used for the figure size, in this case 4x4
with rc_context({'figure.figsize': (4, 4)}):sc.pl.umap(pbmc, color='CD79A')
结果如下
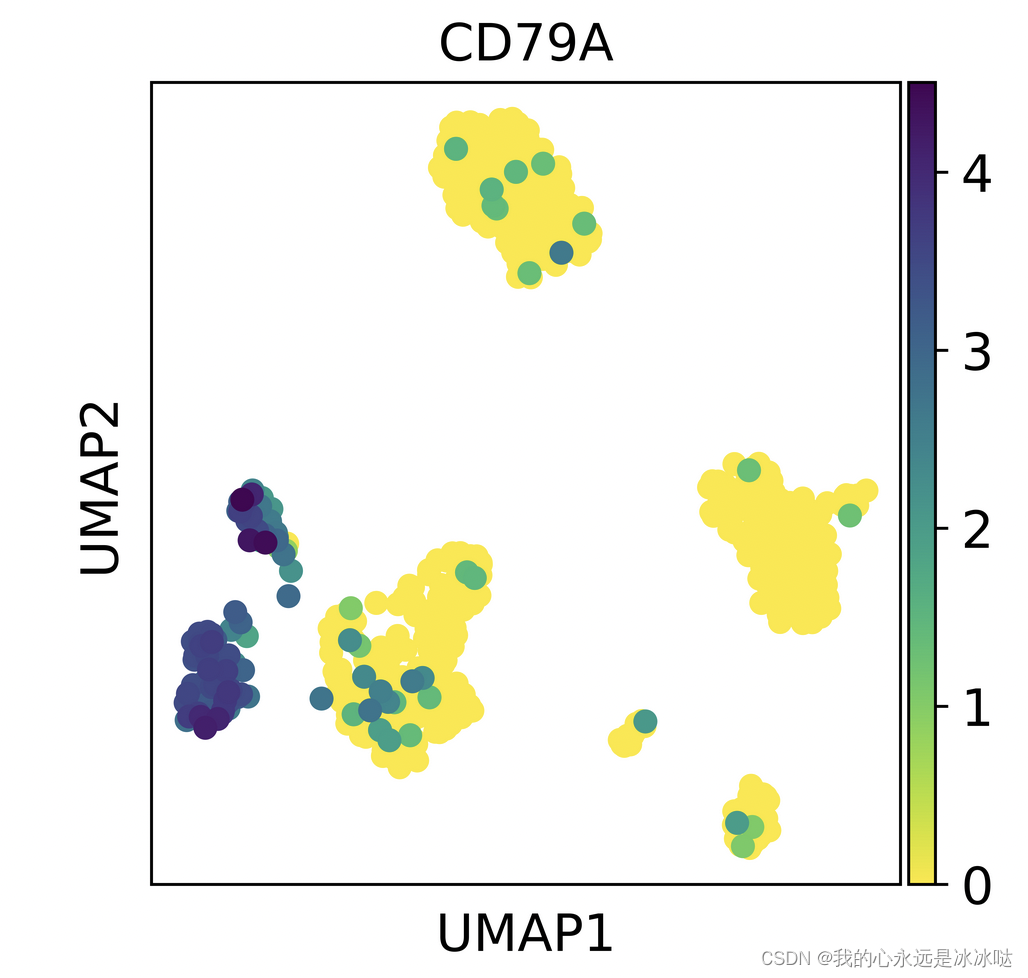
其实从这个图上大致能猜出来,估计是原始的count值,
从下面的图就可以看出
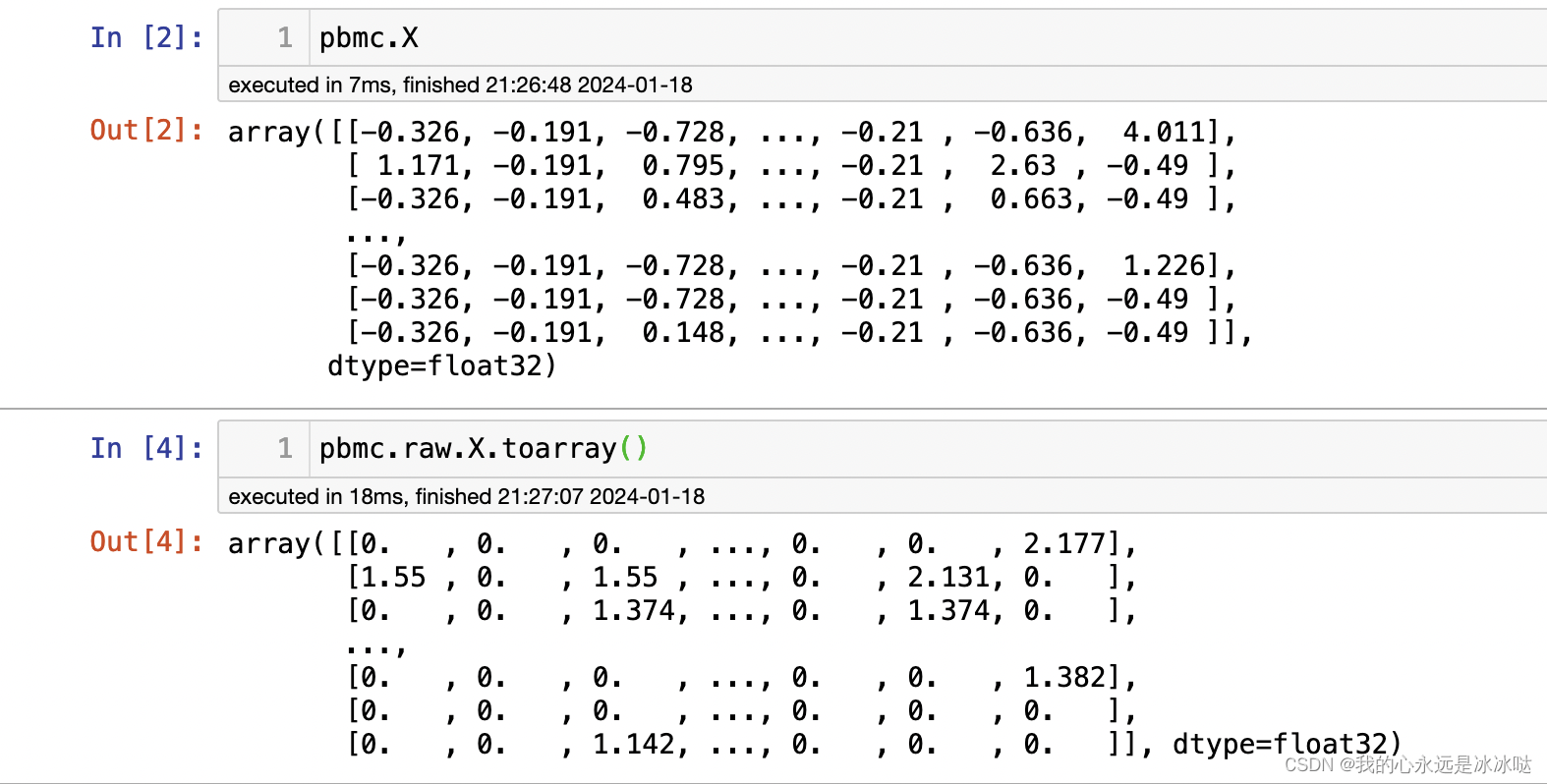
所以猜想并没有问题,下面我还查了scanpy的api,
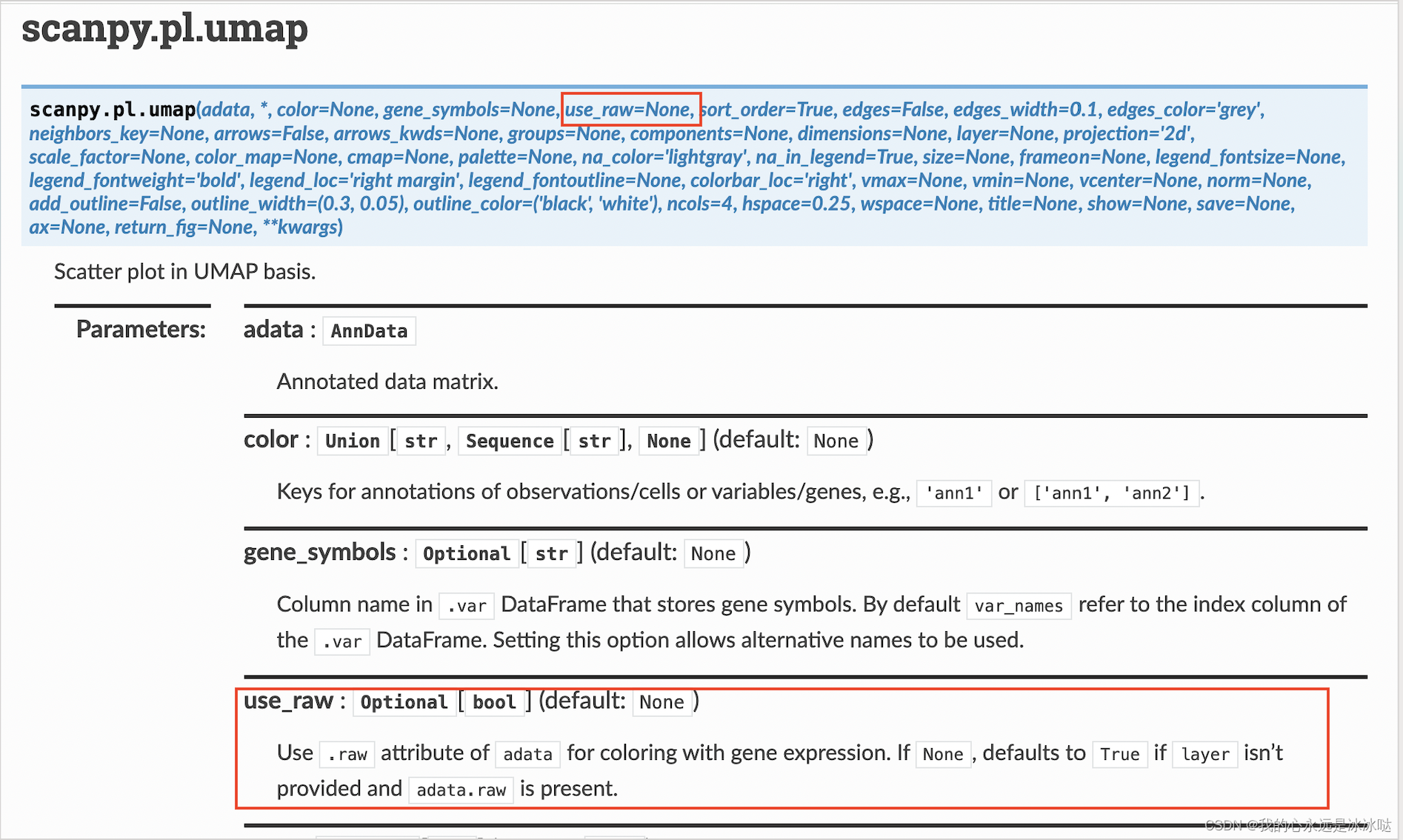
注意这个参数use_raw=None, 也就是说默认就是adata.raw
顺便验证一下
import numpy as np
import seaborn as sns
import matplotlib.pyplot as pltind = (list(pbmc.var_names =="CD79A")).index(True)
CD79A_raw_exp = pbmc.raw.X.toarray()[:,ind]
import pandas as pd
df_umap = pd.DataFrame(pbmc.obsm["X_umap"].copy(),columns =["umap_1","umap_2"])df_umap["exp"] = CD79A_raw_exp cmap = sns.cubehelix_palette(as_cmap=True)f, ax = plt.subplots()
points = ax.scatter(df_umap["umap_1"].values, df_umap["umap_2"].values, c=df_umap["exp"].values, s=20, cmap=cmap)
f.colorbar(points)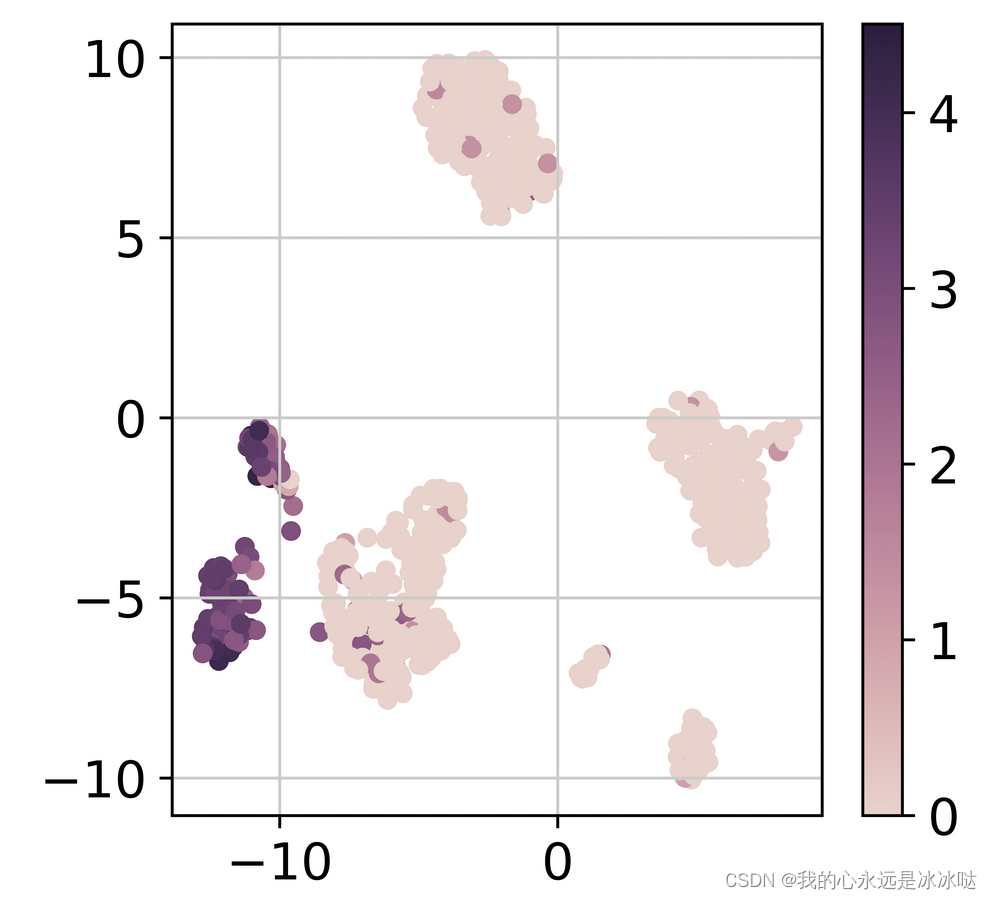
这个图和直接使用sc.tl.umap图其实是一样的,嘿嘿
那么再继续画,如果我不想画原始的raw.X的值,我想画adata.X的值应该如何呢,
其实也很简单
sc.pl.umap(pbmc,use_raw=False, color='CD79A')
结果如下
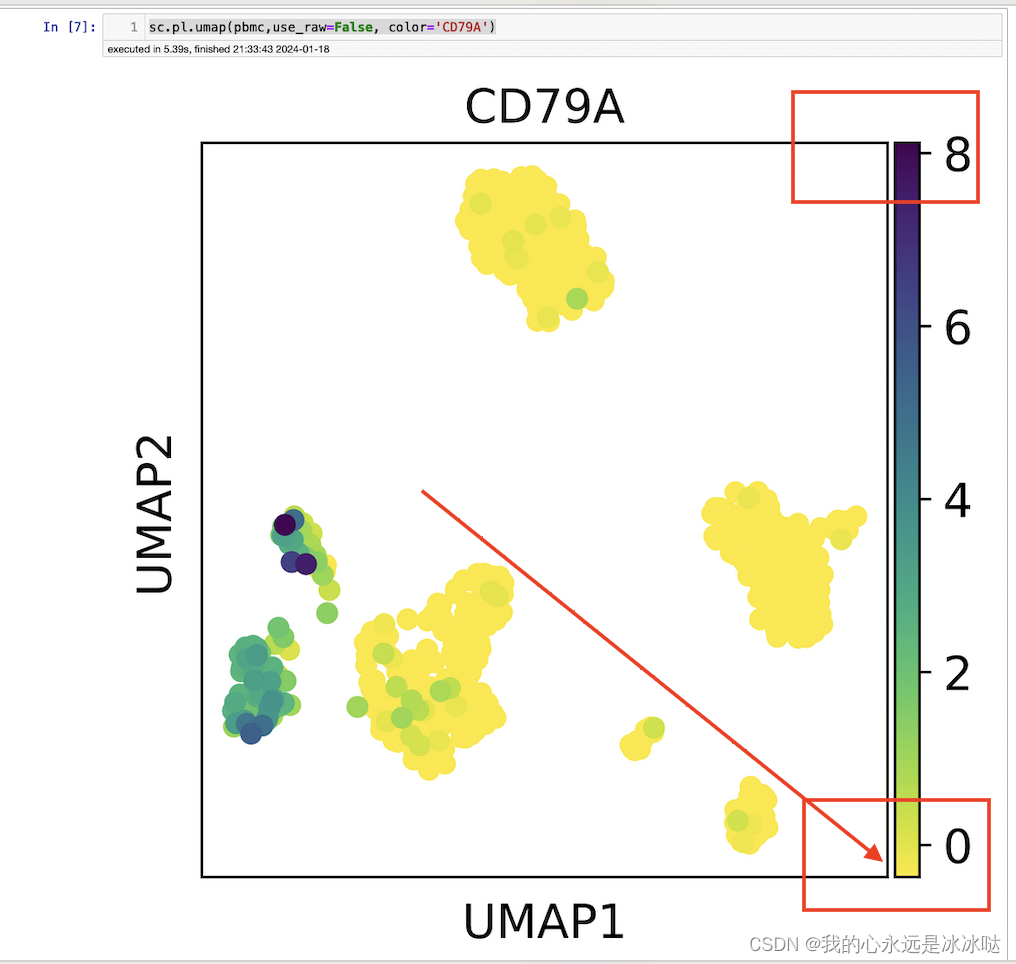
注意这个最大值和最小值的变化
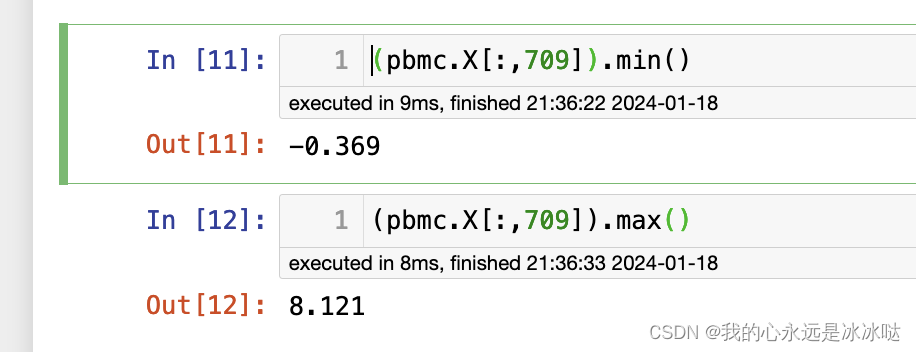
刚好和这个值是对应上的,嘿嘿嘿,这样就清楚了,以后feature plot应该怎么画了
这篇关于sc.pl.umap 画feature plot的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







