本文主要是介绍【论文阅读】General Framework to Evaluate Unlinkability in Biometric Template Protection Systems,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
事实上,仍然没有一个通用的框架来客观地评估生物特征模板的不可链接性,因为现有的文章有一些共同的缺点,包括:对生物特征数据的一致性的不切实际的假设和针对特定系统的非通用方法的开发[5]-[7],将可链接性视为二元决策[5]、[6]、[8]-[11],缺乏量化措施[12]-[14]或使用用于验证准确性评估的度量,不适合于链接性评估[6]、[7]、。[9]-[11]、[15]。
本文提出的不可关联性评估的一般框架解决了以前方法的现有缺点,并提供了以下优点:
·不对数据作出任何假设,既不对独立性也不对一致性进行假设。
·只假定存在一个名为“链接函数”的分类函数,以评估不可链接性属性的非二进制性质[7]。
·提出的指标基于从链接函数获得的分数分布来评估链接性,而与链接函数是什么无关。这允许一个一般的度量,因为它可以对任何勒贝格可积链接函数计算。
·还提出了每个链接分数的局部非链接性措施,以便能够进行更彻底的评估。
·能够独立于链接功能使用相同的度量的优势在于,当使用不同的功能比较模板时,允许监控系统的链接性的变化。
1. 提出的方法
从分析的角度来看,不可链接性可以定义为模板的渐进性:
可链接性的定义:如果存在某种方法来确定两个模板是从相同的生物测定实例中提取的,则它们是完全可链接的。如果存在某种方法来确定两个模板更有可能是从同一实例中提取的,而不是从不同实例中提取的,则两个模板在一定程度上是可链接的。
因此,该属性与用于确定两个模板是否源自同一实例的方法(即,链接函数)完全相关。
A. Local Measure  : System Score-Wise Linkability
: System Score-Wise Linkability
∈[0,1]针对每个特定链接分数s=LS(t1,t2)评估系统的链接性。因此,该度量适合于在一个系统内分析它不能提供不可链接性的链接分数域的哪些部分。如果对于特定分数s1,系统产生D↔(s1)=1,这意味着,在链接函数产生s1的情况下,我们将能够几乎完全确定地将模板T1和T2链接到相同的实例。另一方面,D↔(s0)=0应被解释为该特定分数s0的完全不可链接性。换句话说,如果s0是由链接功能产生的,则两个模板更有可能来自不同的实例,因此无法将它们链接到单个数据主题。D↔(s)在0和1之间的所有中间值都报告了链接性程度的增加。
成功链接到模板的关键在于确定在给定分数s的情况下,是否有两个模板更有可能来自配对样本(Hm)而不是来自非配对样本(Hnm):P(Hm|s)>p(Hnm|s)。
这种关联性可以用以下条件概率的差异来解释:
![]()
(差异越大说明mated的可能性越大,既然都能判定是mated的说明是可以链接的)
然而,这两个条件概率是未知的。因此,我们从已知概率之间的似然比LR(S)=p(s|Hm)/p(s|Hnm)开始计算。
将ω=p(Hm)/p(Hnm)表示为配对样本的未知先验概率与非配对样本分布的未知先验概率之比,我们可以将D↔(S)定义为s的两段函数如下:
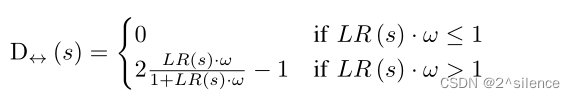
,当式子≤1时表示确定在给定分数s的情况下,mated的概率小于non-mated的概率,因此不可链接,当式子≥1时表示mated的概率大于non-mated的概率,此时可能具有部分链接性。
其中,对于s,D↔(s)=0,使得LR(S)·ω≤1(即,不可链接的分数值,其中p(Hm|s)≤p(Hnm|s))。如果先验概率p(Hm)和p(Hnm)可用,则使用它们来计算ω。否则,我们可以假设p(Hm)=p(Hnm),从而设置ω=1。
B. Global Measure  : System Overall Linkability
: System Overall Linkability
对整个系统的不可链接性进行估计也是有用的,这可能会使两个或更多系统的不可链接性水平有一个更公平的基准。为此,我们引入了全局度量∈[0,1],它独立于分数给出了系统的全局链接性的估计。这样,如果系统具有
=1(即,配对样本和非配对样本分布两者没有重叠的情况),则意味着它对于配对样本分布域的所有分数是完全可链接的。类似地,
=0意味着系统对于整个分数域是完全不可链接的(即,分布完全重叠)。也就是说,与链接函数产生的分数无关,这两个模板来自相同实例(Hm)的可能性与来自不同实例(Hnm)的概率相同。
在0和1之间的所有中间值都报告了链接性程度的增加。
因此,我们感兴趣的是衡量从交配样本分布获得分数的可能性有多大。这可以通过计算差值p(Hm∩s)−p(Hnm∩s)并对其进行积分来实现。关于模板链接的成功与否,我们只对来自配对样本分布的概率感兴趣,并且只有当p(Hm|s)>p(Hnm|s)时才能链接两个模板。因此,定义如下:
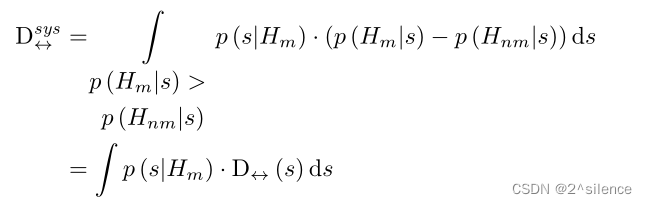
这样,的最终值取决于:i)系统可链接的分数域;ii)系统在该分数域中的可链接程度;以及iii)产生此类分数的可能性。因此,这一新的全球措施为中间情景分配了不同级别的可关联性,而不是完全不可链接或完全可链接。
2. 提出的链接性评估协议
应当指出,在实践中,链接性被定义为跨不同应用程序(即,存储在不同应用程序使用的数据库中)链接模板的能力。考虑到这一点,拟议的议定书运行如下:
1)生成K个受保护模板数据库,每个数据库使用不同的密钥。建议K>5。
2)跨在步骤1中生成的K个数据库计算所选链接函数的配对样本和非配对样本得分分布。
3)如果p(Hm)和p(Hnm)可用,则使用它们来计算ω。否则,将ω设置为1。
4)计算和
。
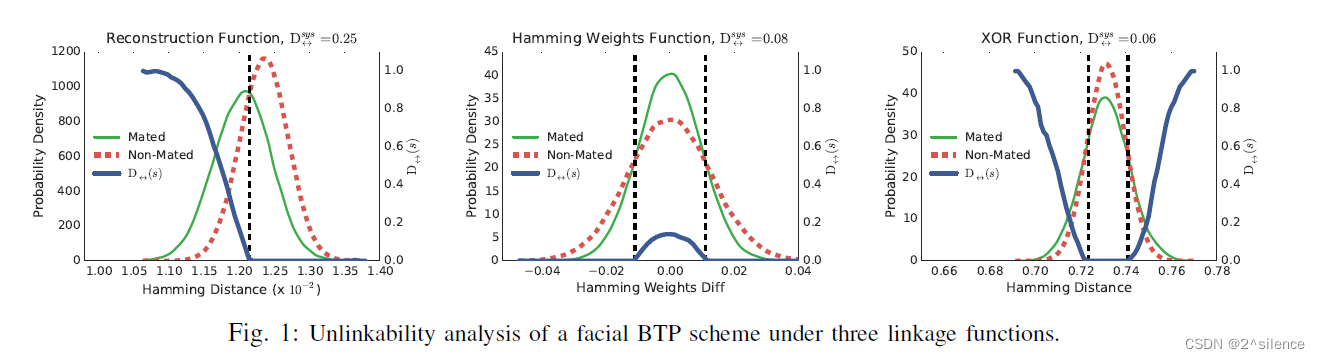
5)报告图,以及配对样本和非配对样本分布,以及相应的全局链接性值
(见图1中的示例)。
6)分析曲线图和值
3. 总结
我们提出了两个新的量化方法(D↔(S)和)来分析生物特征模板的去连接性,它们可以应用于任何BTP方案。它们提供了对模板的关联性进行详细的计分分析以及对不同系统的关联性进行基准的能力。此外,为了开发生物模板保护方案的完整安全基准,已经提出了实现完全去链接性评估的必要步骤。因此,我们相信,拟议的框架将有助于推动未来生物识别技术的发展。
这篇关于【论文阅读】General Framework to Evaluate Unlinkability in Biometric Template Protection Systems的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!