本文主要是介绍squidpy 教程 3:使用 Napari 进行交互式可视化,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
「写在前面」
学习一个软件最好的方法就是啃它的官方文档。本着自己学习、分享他人的态度,分享官方文档的中文教程。软件可能随时更新,建议配合官方文档一起阅读。推荐先按顺序阅读往期内容:
文献篇:
1.文献阅读:Squidpy: 一个可扩展的空间组学分析框架
教程篇:
1.squidpy 教程 1:在 AnnData 和 Squidpy 中导入空间数据
2.squidpy 教程 2:ImageContainer 对象
目录
-
1 可视化 AnnData 的观测和特征 -
2 使用形状层注释组织区域
官网教程:
https://squidpy.readthedocs.io/en/stable/notebooks/tutorials/tutorial_napari.html
本教程展示如何在 Napari 中可视化 squidpy.im.ImageContainer 和 AnnData。
以交互方式探索 Scanpy/Squidpy 分析结果非常有用。Napari 是 Python 的多维图像查看器,这使得它非常方便地实现此目的。在本教程中,我们将展示 Squidpy 如何实现 Napari 与 AnnData 对象的无缝集成,从而非常轻松地交互式探索分析结果并在 ImageContainer 中包含的高分辨率组织图像上将其可视化。
对于本教程,您可能需要安装其他库,请确保使用 pip install 'squidpy[interactive]' 安装了 Squidpy。
像往常一样,让我们导入相关库并加载数据。
import squidpy as sq
print(f"squidpy=={sq.__version__}")
## squidpy==1.2.2
adata = sq.datasets.visium_hne_adata()
img = sq.datasets.visium_hne_image()
该步骤可能因为网络问题无法自动下载数据,需要自己手动下载数据: https://ndownloader.figshare.com/files/26098397 https://ndownloader.figshare.com/files/26098124
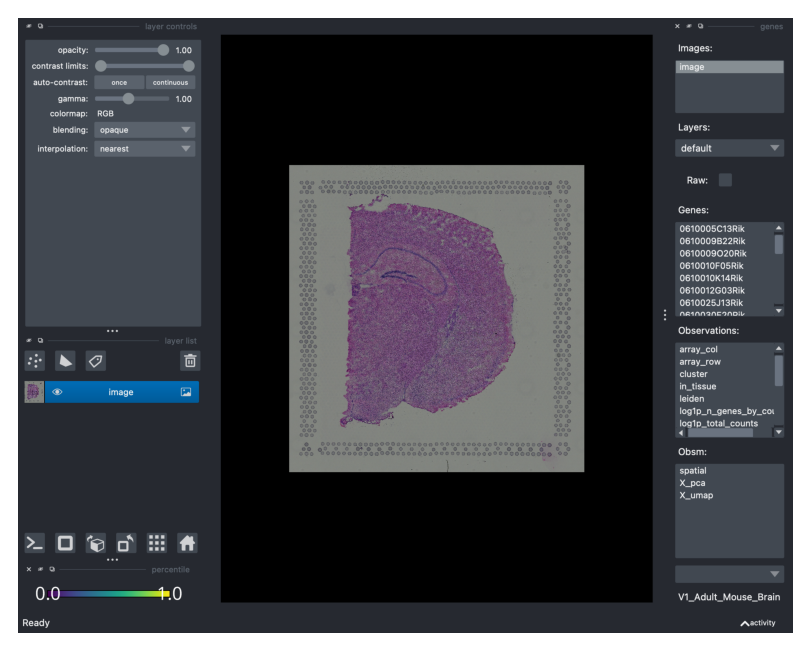
在下面的行中,我们实际上启动了 Napari。您会注意到,将弹出一个带有 Napari viewer 的附加窗口。我们将 Napari viewer 分配给一个新变量,viewer,以便执行一些有用的操作,例如屏幕截图。对于熟悉 napari 的用户来说,viewer 是 napari.Viewer 的包装器,而不是同一个对象。
viewer = img.interactive(adata)
通过方便地捕获当前图像的屏幕截图,我们始终可以在此笔记本中可视化 Napari 会话。这可以通过 viewer.screenshot() 来完成。所有描述的功能均基于 Napari layers 和 widget。您可能想熟悉一些 Napari tutorials。
在这里,我们截取画布以及带有所有可用小部件的 GUI 的屏幕截图。
viewer.screenshot(canvas_only=False)
1 可视化 AnnData 的观测和特征
在 Napari GUI 的右侧,您可以选择并可视化 AnnData 对象中的不同插槽,例如:
-
来自默认 layer adata.X或其他 layers 例如adata.raw的 Genes。 -
Observations 保存在 adata.obs中(例如聚类结果、QC 指标、其他类型的 observation-level 注释)。 -
来自 adata.obsm的特征(例如,PCA 结果、计算图像特征和其他 observation-level 表示)。
这些 widgets 是可搜索的,这意味着您可以输入首字母缩写,例如您最喜欢的基因,菜单将自动滚动到该位置。为了将感兴趣的特征可视化为附加 layer,您可以按 Enter 键或双击它。
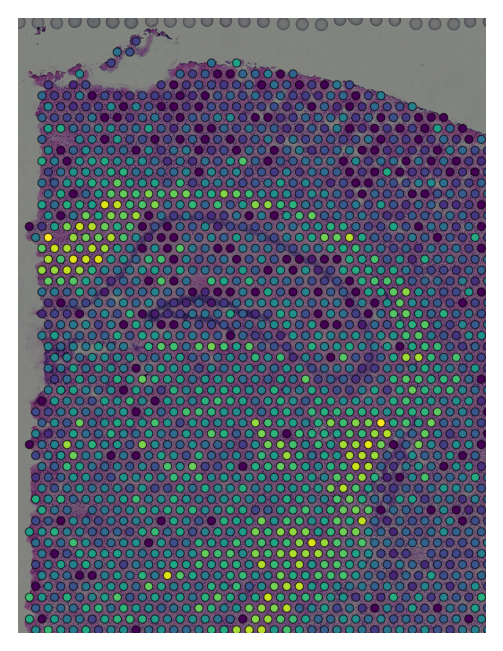
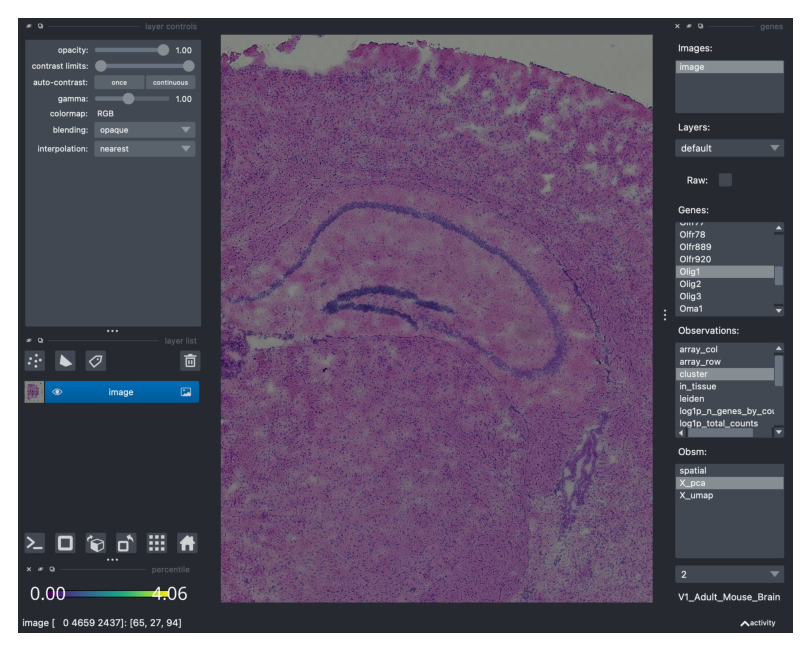
例如,让我们可视化 Olig1 的表达水平……
viewer.screenshot(canvas_only=False)

如果您希望仅可视化画布,则默认情况下 canvas_only = True。这是放大的可视化:
viewer.screenshot()
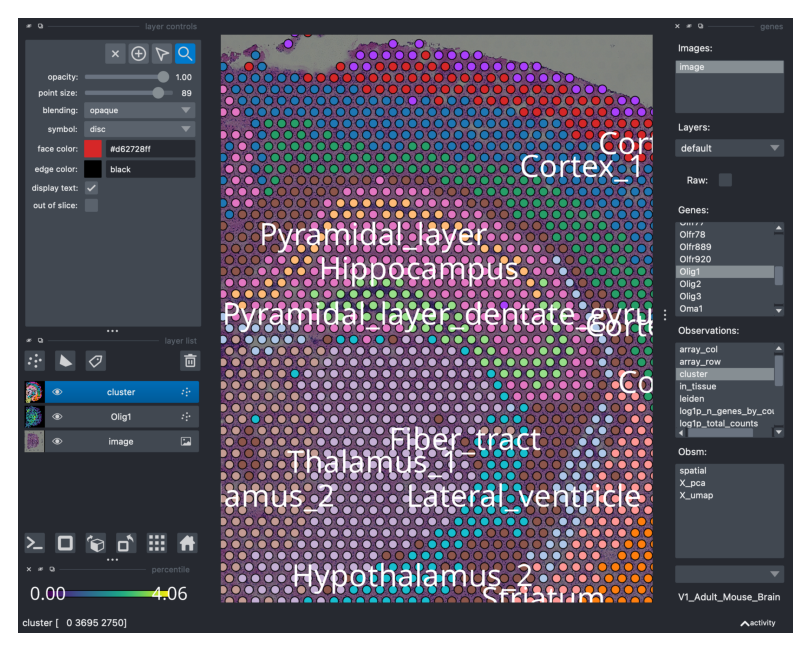
如前所述,我们可以可视化保存在 adata.obs['cluster'] 下的聚类注释。请注意,通过选择并单击注释,将在 Napari 中创建一个新图层,位于前一个图层的顶部(请参见 Napari GUI 的左侧)。您可以通过简单地关闭图层(单击“眼睛”图标)或修改不透明度(左上角)来修改图层的可视化。
viewer.screenshot(canvas_only=False)
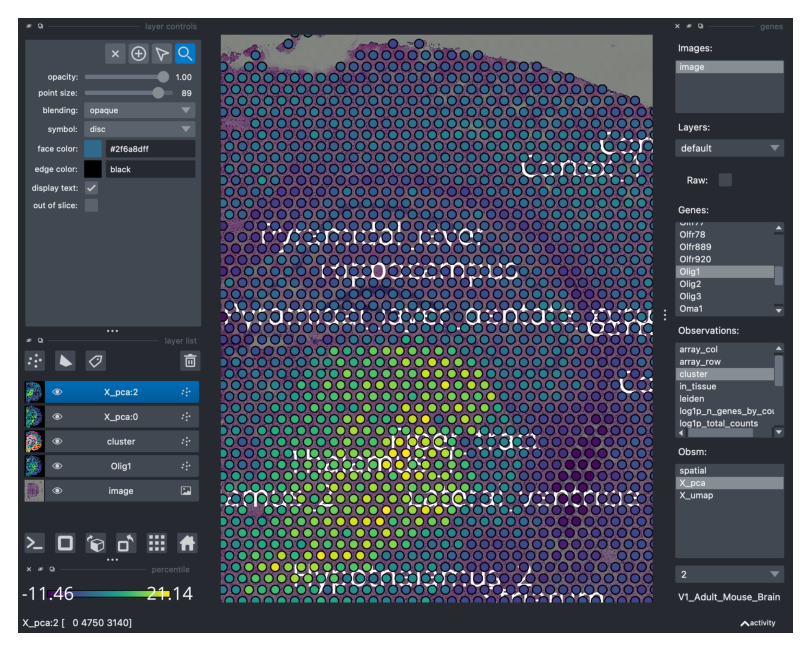
adata.obsm 中的插槽也可以搜索并且可以可视化。例如,Squidpy 将图像特征提取步骤的结果存储在 adata.obsm 中。出于本教程的目的,让我们可视化第三主成分(选择 X_pca,然后在右下菜单中搜索它)。
viewer.screenshot(canvas_only=False)
您只需单击 GUI 中左侧的 “bin” 即可删除所有图层。
viewer.screenshot(canvas_only=False)
2 使用形状层注释组织区域
Napari 不仅可以用作交互式图像查看器,还可以用于手动注释组织中感兴趣的区域。为此,您需要选择 Napari 中的 Shapes layer(左侧的小部件,形状像梯形的图标),然后选择 add polygons 图标并在感兴趣的区域中绘制多边形。您可以查看 Napari shapes layer tutorial 以获取更多说明。
在这里,我们将裁剪 2 个感兴趣的区域(分配给相同的 Shapes 图层)。
viewer.screenshot(canvas_only=False)

现在,在组织上绘制多边形可能很有趣,但不一定有用。如果我们能够将组织区域注释的信息转移到我们的观察结果中(存储在 adata.obs 中),那将会非常有用。幸运的是,使用我们的包装器很简单:只需键入 SHIFT+E,注释就会“转换”为 adata.obs 中的布尔向量。如果该 spot 被多边形“包含”,则它将被注释为 True,否则为 False。
viewer.screenshot(canvas_only=False)
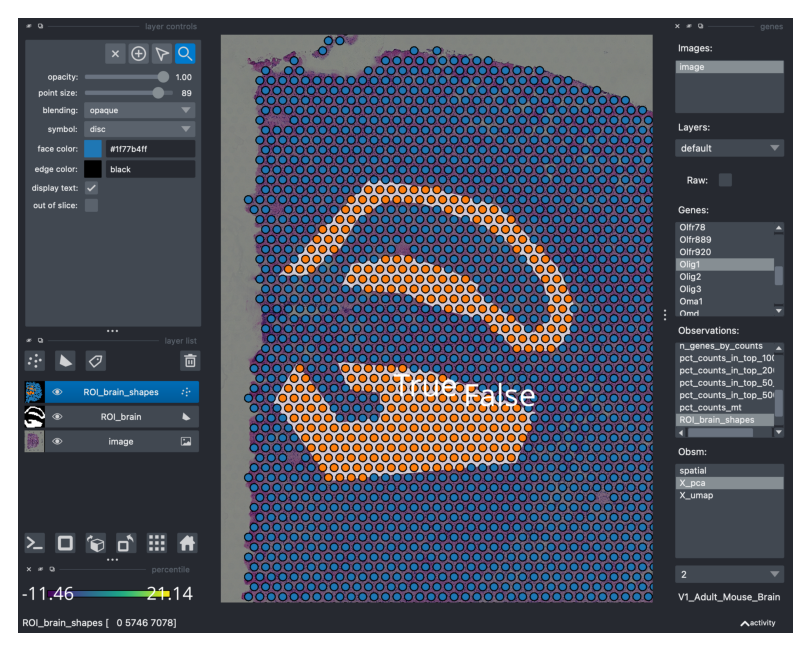
当键入 SHIFT+E 时,注释将保存在 adata.obs 下,并且可以方便地进行可视化。
这种交互式注释在癌症病理学背景下特别有用,病理学家经常根据形态信息对组织进行注释,并且分析人员可能希望在下游分析中将此注释与其他类型的特征(离散或连续)相关联。
最后,回想一下,Napari 查看器上出现(并且可以可视化)的所有内容都存储在 AnnData 对象中。因此,我们可以随时使用 Squidpy 访问它并可视化它。
sq.pl.spatial_scatter(viewer.adata, color=["ROI_brain_shapes", "cluster"])
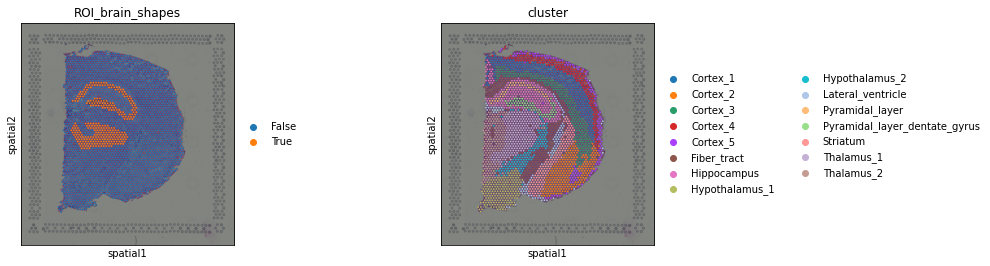
本教程到此结束,欢迎查看其他 Squidpy 教程以及 Napari resources。
viewer.close()

本文由 mdnice 多平台发布
这篇关于squidpy 教程 3:使用 Napari 进行交互式可视化的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







