本文主要是介绍R语言绘制桑基图教程,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
原文链接:R语言绘制桑基图教程
写在前面
在昨天3月10日,我们在知乎、B站等分享了功能富集桑基气泡图的绘制教程。相关链接:NC|高颜值功能富集桑基气泡图,桑基气泡组合图。
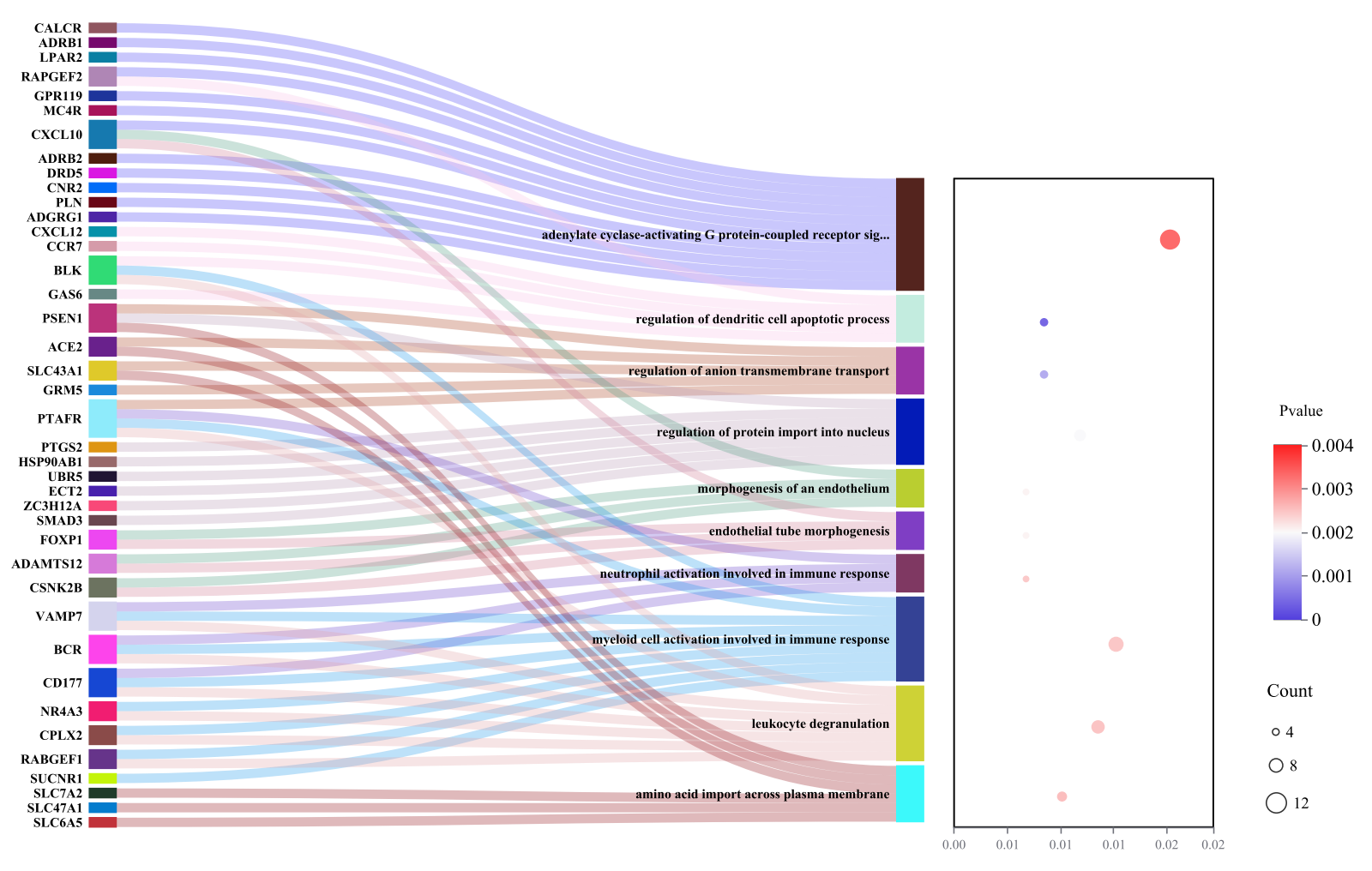
确实,目前这个图在文章中出现的频率相对比较少,但是可能随着的代码的开放,此图会别大家玩出新花样,功能富集图可能会有不同的可视化方式,若你有好的想法,也可以在自己想到后进行实现,你可能成为此图的开山鼻祖。
做生物信息的,对图形的要求是比较高的,因此,需要不断的创新,不断的调整,不断的失败,更重要的是需要不断的思考。
因此,我们每天学习一点点,也就是一种进步;每天整理一下笔记,也算是一种总结。
本期教程图形

绘制
- 导入所需的R包
library(tidyverse)
# devtools::install_github("davidsjoberg/ggsankey")
library(ggsankey)
library(ggplot2)
#install.packages("cols4all")
library(cols4all)
#BiocManager::install("dittoSeq")
library(dittoSeq)
- 导入数据
setwd("E:\\小杜的生信筆記\\2024\\20240311_桑基图绘制教程")df <- read.csv("01_inputdata.csv",header = T)
head(df)
> head(df)Pathway geneID FreqFlavonoid biosynthesis pmb0751 1Flavonoid biosynthesis pmp000571 1Flavonoid biosynthesis mws0914 1Flavonoid biosynthesis pme2960 1Flavonoid biosynthesis pme1201 1Flavonoid biosynthesis mws1068 1
- 转化格式
df2 <- df %>% make_long(Pathway, geneID)##'@指定绘图显示顺序
df2$node <- factor(df2$node, levels = c(rev(unique(df$geneID)),rev(unique(df$Pathway))))head(df2)
# A tibble: 6 × 4x node next_x next_node<fct> <fct> <fct> <chr>
1 Pathway Flavonoid biosynthesis geneID pmb0751
2 geneID pmb0751 NA NA
3 Pathway Flavonoid biosynthesis geneID pmp000571
4 geneID pmp000571 NA NA
5 Pathway Flavonoid biosynthesis geneID mws0914
6 geneID mws0914 NA NA
绘制基础图形
代码一
mycol3 <- sample(mycol3, length(mycol3)) ##'@代码一
ggplot(df2, aes(x = x, next_x = next_x, node = node, next_node = next_node,fill = node, label = node)) +scale_fill_manual(values = mycol3)+geom_sankey(flow.alpha = 0.5, #条带不透明度smooth = 7, #条带弯曲度width = 0.18) + #节点宽度geom_sankey_text(size = 3.2, color = 'black') +theme_void() +theme(legend.position = 'none') #隐藏图例ggsave("桑基图.jpg",width = 6, height = 4)

代码二
ggplot(df2, aes(x = x, next_x = next_x, node = node, next_node = next_node,fill = node, label = node)) +#设置geom_sankey(flow.fill="#DFDFDF",#连线颜色flow.alpha = 0.5, ## 条带透明度flow.color="grey60",#连线边框颜色#node.fill=dittoColors()[1:36],#节点颜色,[1:36]数值需要根据自己的数据进行修改width=0.2) + #node的宽度#设置桑葚图文字geom_sankey_text(size = 3,#文字大小color= "black",#文字颜色hjust=1) + #文字位置,右对齐theme_void()+#隐藏图例theme(legend.position = 'none') ggsave("桑基图02.jpg",width = 6, height = 4)

注意:#node.fill=dittoColors()[1:36],#节点颜色,[1:36]数值需要根据自己的数据进行修改,如下图所示。
参考:
https://mp.weixin.qq.com/s/Wv90W3In7vzhnRjCYZXs7A
若我们的分享对你有用,希望您可以点赞+收藏+转发,这是对小杜最大的支持。
往期文章:
1. 复现SCI文章系列专栏
2. 《生信知识库订阅须知》,同步更新,易于搜索与管理。
3. 最全WGCNA教程(替换数据即可出全部结果与图形)
-
WGCNA分析 | 全流程分析代码 | 代码一
-
WGCNA分析 | 全流程分析代码 | 代码二
-
WGCNA分析 | 全流程代码分享 | 代码三
-
WGCNA分析 | 全流程分析代码 | 代码四
-
WGCNA分析 | 全流程分析代码 | 代码五(最新版本)
4. 精美图形绘制教程
- 精美图形绘制教程
5. 转录组分析教程
转录组上游分析教程[零基础]
一个转录组上游分析流程 | Hisat2-Stringtie
小杜的生信筆記,主要发表或收录生物信息学的教程,以及基于R的分析和可视化(包括数据分析,图形绘制等);分享感兴趣的文献和学习资料!!
这篇关于R语言绘制桑基图教程的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!





