本文主要是介绍生物信息学-山大精品课,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
文章目录
- 常用专业名词定义
- 生物信息学(山大国家精品课)
- 第一章:绪论
- 1.1 课程介绍
- 1.2 探索生物信息学神秘岛
- 1.3生物信息学究竟是神马?
- 1.4 这门课到底学什么?
- 第二章:生物数据库(第一部分)
- 2.1为什么需要生物数据库?
- 2.2生物数据库的分类
- 2.3文献数据库
- 2.4一级核算数据库GenBank
- 2.5一级核酸数据库:人的基因组数据库 Ensemble
- 2.5.2 一级核酸数据库:微生物宏基因组数据库 JCVI
- 2.6二级核酸数据库
- 第二章:生物数据库(第二部分)
- 第三章:序列比较 第一部分
- 3.1 认识序列
- 3.2序列相似性
- 3.3 替换计分矩阵
- 3.3.1 DNA替换积分矩阵
- 3.3.2 蛋白质的替换计分矩阵
- 3.4 序列两两比较:打点法
- 3.4.1 打点法介绍以及用途
- 3.4.2 在线打点工具界面介绍
- 3.4.3 在线打点工具应用实例
- 3.5序列两两比较:序列比对法
- 3.5.1序列比对法
- 3.5.2双序列·全局比对及算法
- 3.5.3 双序列·局部比对
- 3.5.4 一致度和相似度
- 第三章:序列比较 第二部分
- 3.7 在线双序列比对工具
- 3.7.1 GAP的类型及分值设置
- 3.8 BLAST搜索
- 第三章:序列比较 第三部分
- 3.9 多序列比对介绍
- 3.10 在线多序列比对工具
- 3.11 多序列比对的编辑和发布
- 3.12 寻找保守区域
- 第四章:分子进化与系统发生
- 4.1 进化的故事
- 4.2基本概念
- 4.3系统发生树
- 4.4 系统发生树的构建
- 4.5 MEGA7构建NJ树
- 4.6课后甜品
- 第五章:蛋白质结构预测与分析(第一部分)
- 5.1蛋白质的结构
- 5.2 蛋白质的二级结构
- 5.3 蛋白质的三级结构
- 5.4 三级结构可视化软件VMD
- 第五章:蛋白质结构预测与分析(第二部分)
- 5.5计算方法预测三级结构
- 5.6 三级结构的比对
- 5.7 蛋白质分子表面性质
- 第五章:蛋白质结构预测与分析(第三部分)
- 5.8 获取蛋白质四级结构
- 5.9 蛋白质-蛋白质分子对接
- 5.10 蛋白质-小分子分子对接
- 5.11 虚拟筛选与反向对接
- 5.12 分子动力学模型
- 第六章:高通量测序技术及应用
- 6.1基因组学与预测技术
- 6.2 高通量测序技术在精准医学中的应用
- 6.3 生物信息学面临的挑战
- 6.4 从头测序
- 6.5 重测序
- 6.6 转录组测序
- 6.7 表观基因组学
- 6.8 猛犸象基因组测序计划
- 6.9 古基因组学面临的挑战
- 6.10 古基因组学研究中的生物信息技术
- 第七章:统计基础与序列算法
- 7.1贝叶斯公式机器生物学应用
- 7.2 二元预测的灵敏度和特异度
- 7.3 基本序列算法
- 第八章:数据挖掘
- 8.1 什么是数据挖掘
- 8.2 数据库系统
- 8.3机器学习
- 8.4weka
- 第九章:编程基础与网页制作(第一部分)
- 9.1 Linux操作系统
- 9.2 Linux基本命令
- 9.3Perl语言基础入门
- 9.4Perl语言基础高级
- 第九章:编程基础与网页制作(第二部分)
- 9.5 前端开发和HTML介绍
- 9.6 HTML常用标签
- 9.7 设计简单的网页
- 9.8 HTML与CGI简单交互
常用专业名词定义
| 名称 | 解释 |
|---|---|
| DAN序列 | 基因 |
生物信息学(山大国家精品课)
第一章:绪论
1.1 课程介绍
推荐资料:[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-qdsQBnME-1632484541410)(picofwfy.wangfuyu.cn/img/wfy20200529212423.png)]
普通高等教育“十二五”规划教材:生物信息学(第二版)陈铭,2015,科学出版社
全国高等学校教材:生物信息学(第二版),李霞,2015,人民卫生出版社
生物信息学(中译本),T.Charlie Hodgamn,2013,科学出版社
生物信息学,许忠能,2010,清华大学生出版社
生物信息学,赵国屏,2017,科学出版社
Bioinformatics For Dummies,2nd Edition,Jean-Michel Claverie,2007,Wiley
Bioinformatics-An introduction,2nd Edition,Jeremy Ramsden,2009,Springer
期刊:
Bioinformatics,Impactfactor:5.5,Oxford University Press
期刊:BMC Bioinformatics,Impact factor:2.8,BioMed Central
1.2 探索生物信息学神秘岛
生物信息学的起源
[
克里克提出的中心法则主要是说:DNA复制形成DNA,DNA转录形成RNA,RNA再翻译成为蛋白质。
今天我们知道:病毒中的RAN可以自我复制,RNA还可以逆转录为DNA,甚至理论上可以实现遗传物质从DNA到蛋白质的直接转移。
随着测序技术的出现以及计算机技术的快速发展
1.3生物信息学究竟是神马?
- 生物信息学的定义
HGP人类基因组计划第一个五年总结报告给出了生物信息学的完整定义:。。。
总结起来:生物信息学就是用计算机来解决生物问题。
- 生物信息学的研究对象
1.4 这门课到底学什么?
序列算法这一章将会告诉你如何将生物问题转化为数学问题并高效解决,
第二章:生物数据库(第一部分)
2.1为什么需要生物数据库?
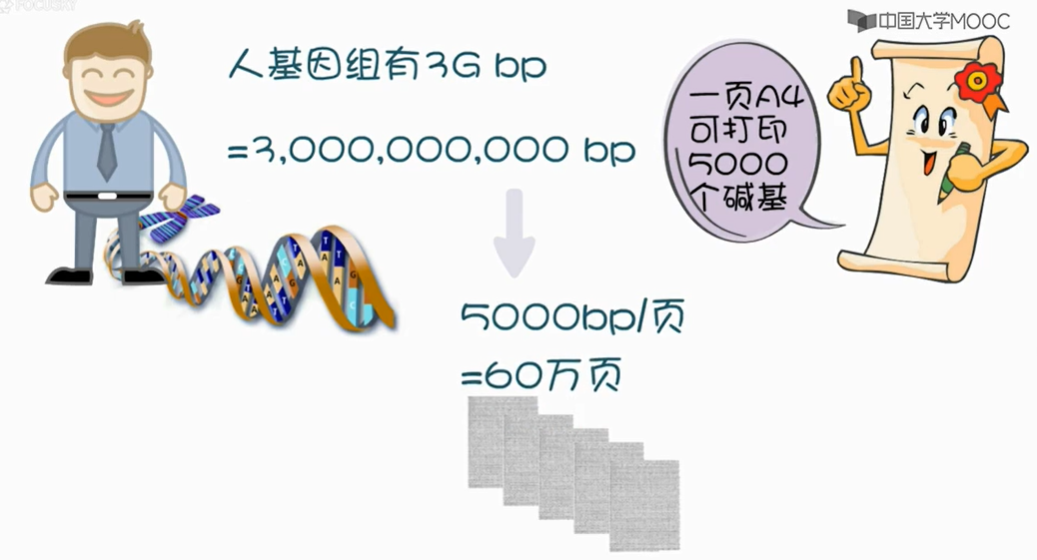

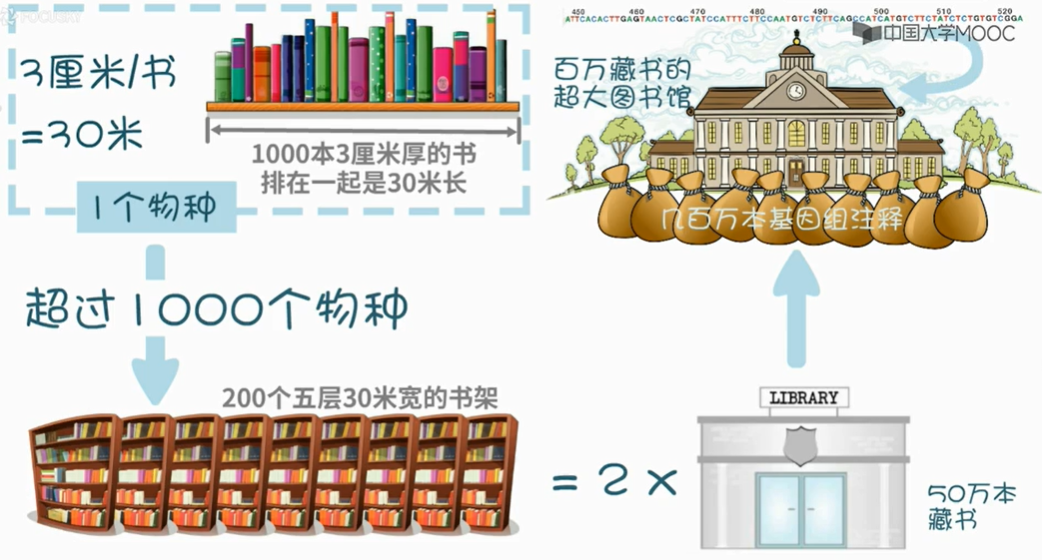
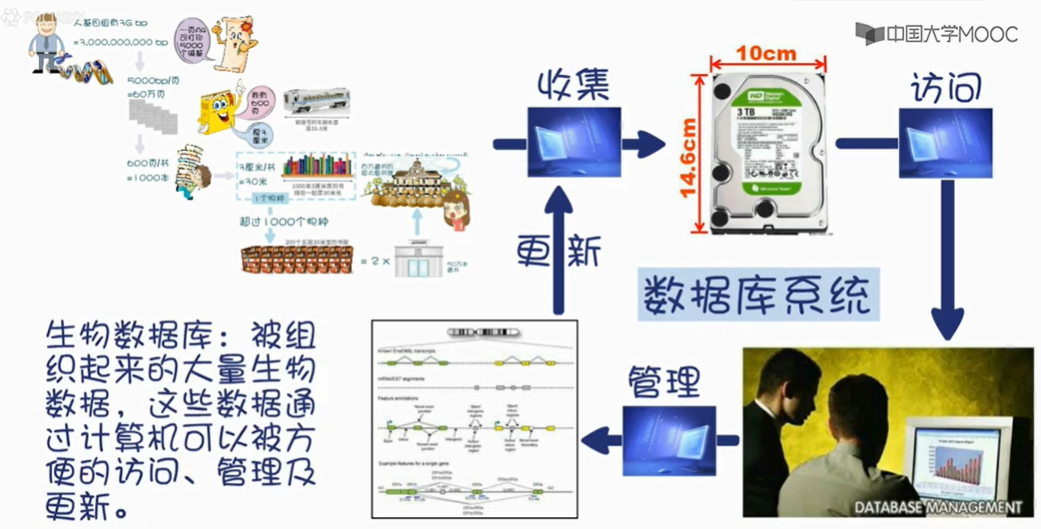

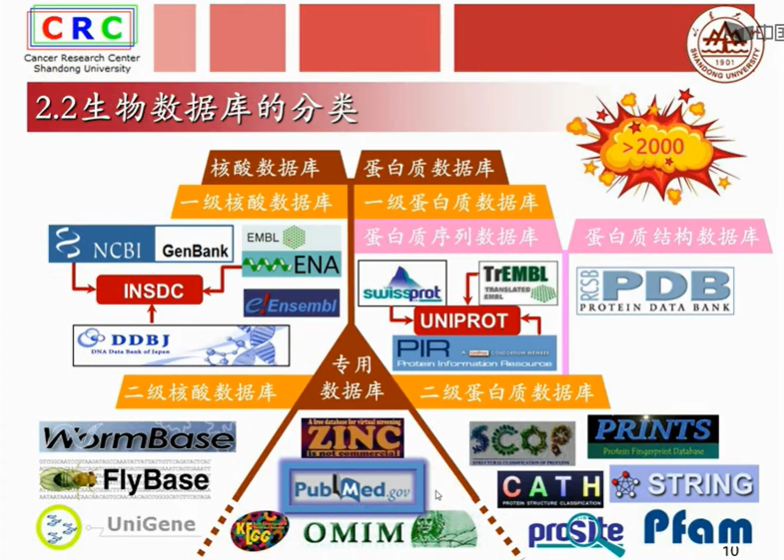
2.2生物数据库的分类

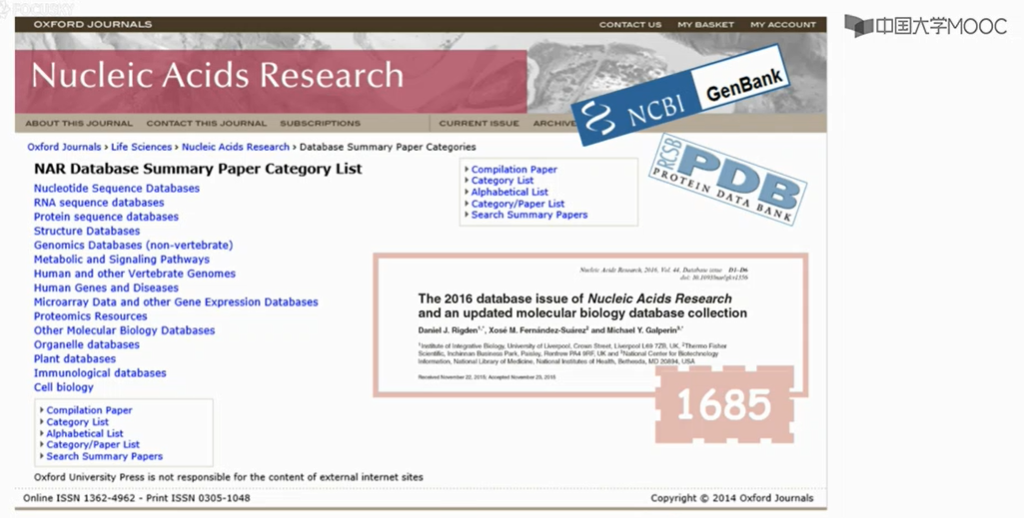
著名的学术期刊,NAR有一个生物数据库专刊,有点规模的数据库都争相在这里发表,包括基因Bank和PDB等等。截止2015年底,这个专刊收录的生物数据库大概1685个。当然还有在其它刊物发表的小型专项数据库,这些数据库加起来,目前世界上大概得有超过2000个生物数据库。当然了,不是所有的数据库都是活的。


这么多数据库讲不完,所有我们只挑最重要的,最常用的,以及和你的专业最密切相关的进行讲解。
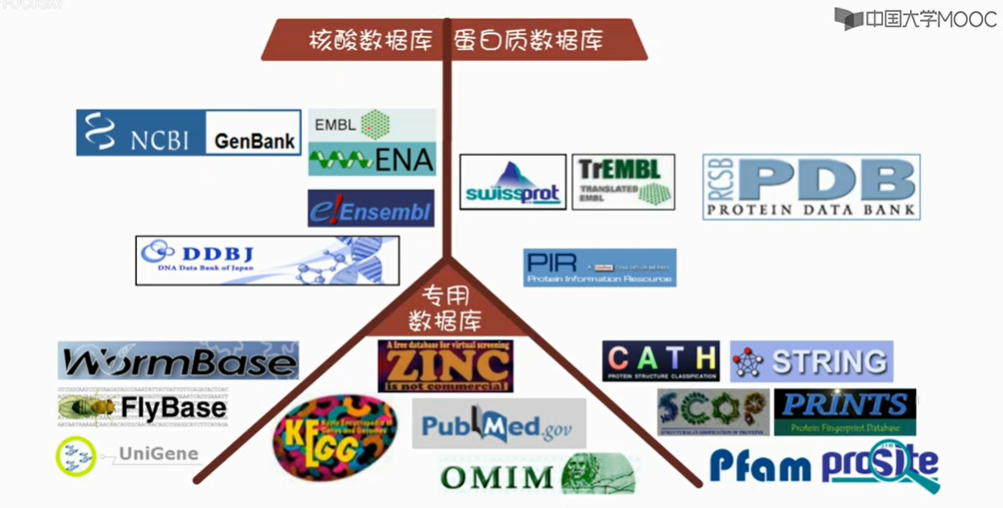
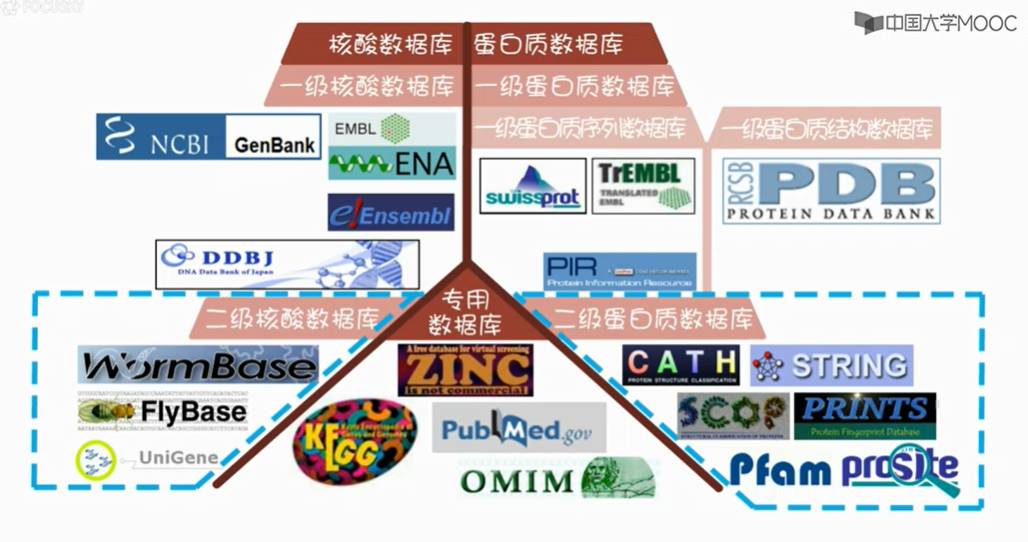
一级数据库存储的是通过各种科学手段存储的直接数据,比如测序获得的核酸序列,(注:脱氧核糖bai核酸测序即DNA测序(DNA sequencing)是指分析特定duDNA片段的碱基序列,也就是腺嘌呤(zhiA)、胸腺嘧啶(T)、胞嘧啶(C)与鸟嘌呤的(G)排列方式。快速的DNA测序方法的出现极大地推动了生物学和医学的研究和发现。)或者X射线衍射法获得的蛋白质三维结构;
蛋白质一级数据库还可以具体的分为蛋白质序列数据库和蛋白质结构数据库。(注:简单的说,核酸bai测序是以待du测核酸作为模板,通过引物边合成边测zhi序,通过碱基配对dao的原理,以新加入的碱基序列读取待测DNA的序列。
蛋白测序采用的多为末端降解的方面,即采用的是逐个从末端释放氨基酸单体,通过释放氨基酸的鉴定,得出目标蛋白的序列,所谓蛋白质测序,主要指的是蛋白质的一级结构的测定。蛋白质的一级结构(Primary structure)包括组成蛋白质的多肽链数目。很多场合多肽和蛋白质可以等同使用。多肽链的氨基酸顺序,它是蛋白质生物功能的基础。)
二级数据库是通过对一级数据库的资源进行分析整理,归纳、注释而构建的具有特殊生物学意义和专门用途的数据库,比如从三大核算数据库和基因组数据库中提取并加工出的果蝇和蠕虫数据库。 再比如根据蛋白质三级结构数据库中的结构信息,分析统计出的蛋白质结构分类数据库,cath和scope等。
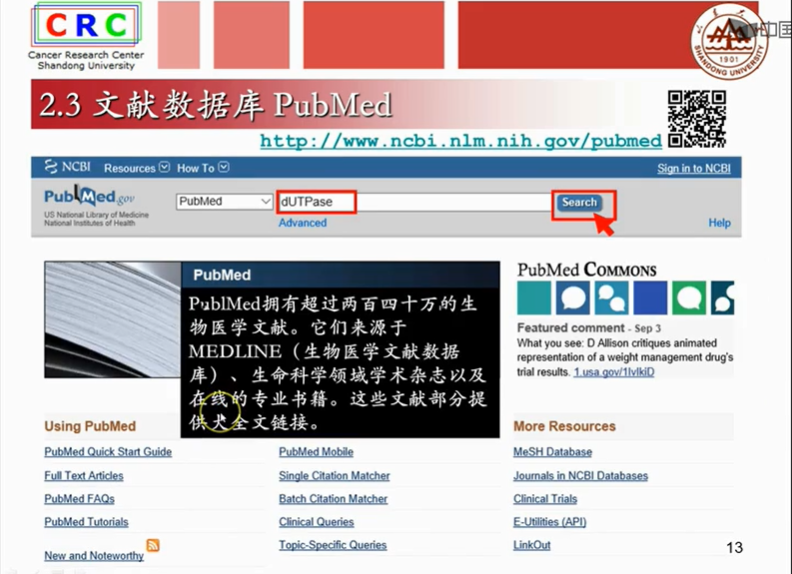
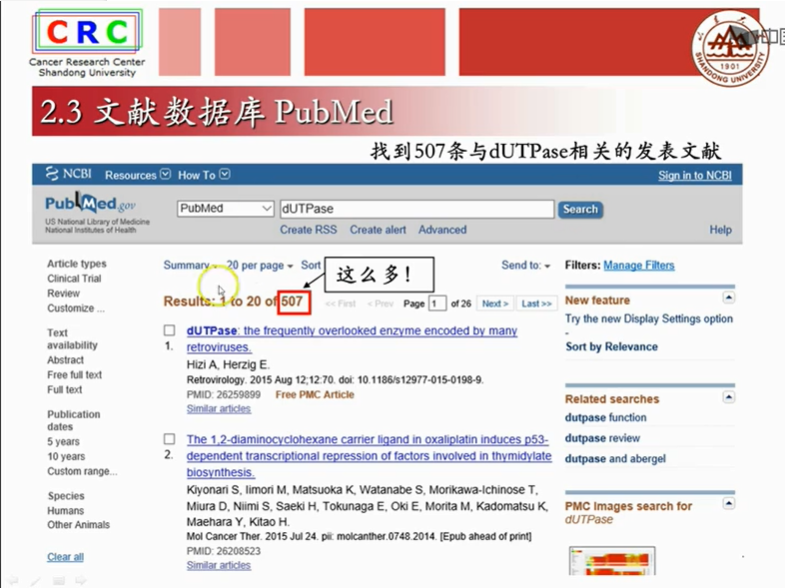
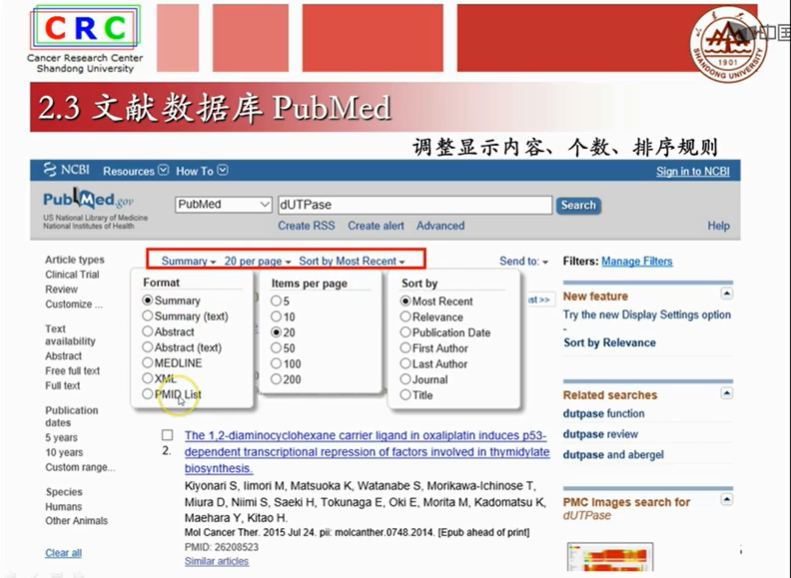
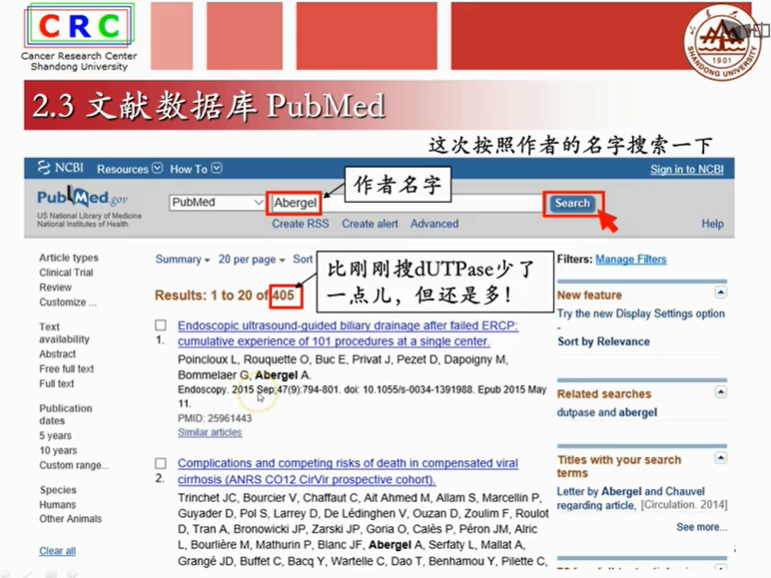
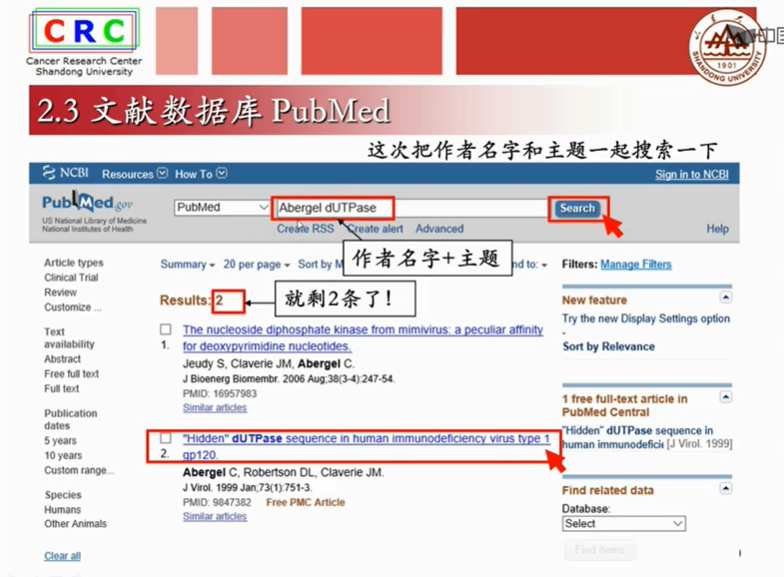
2.3文献数据库


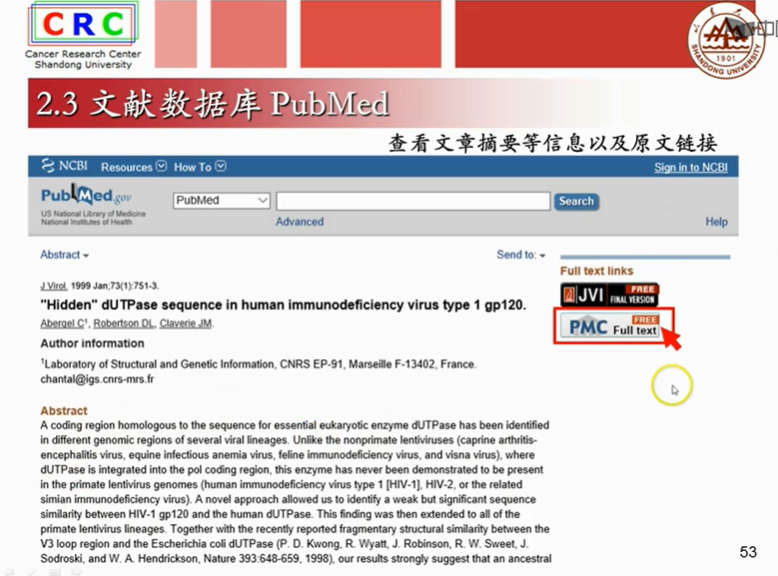
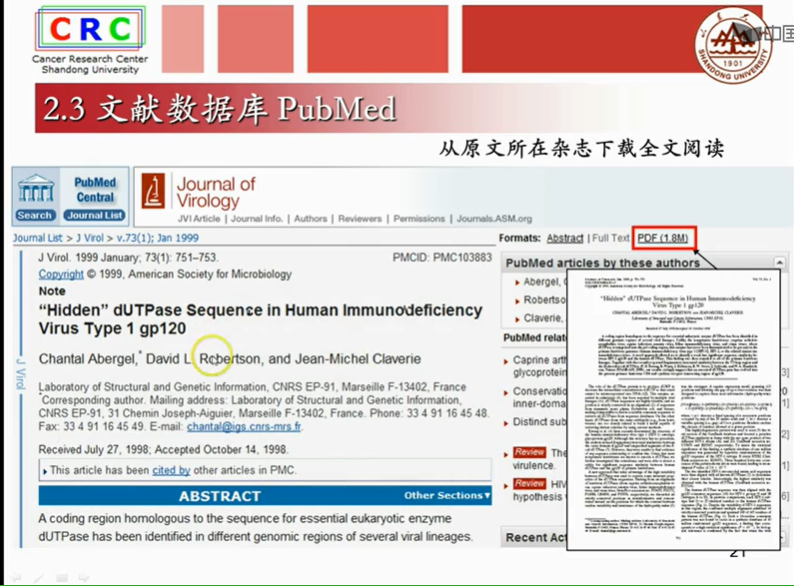
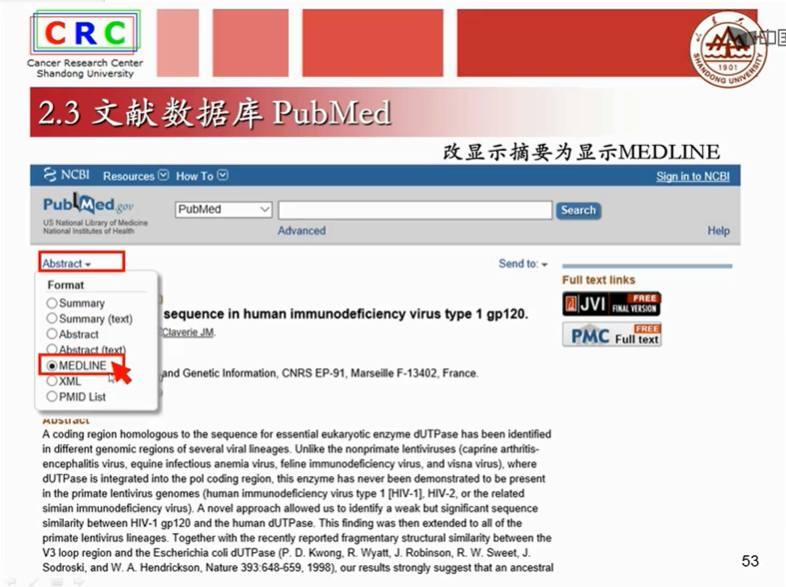

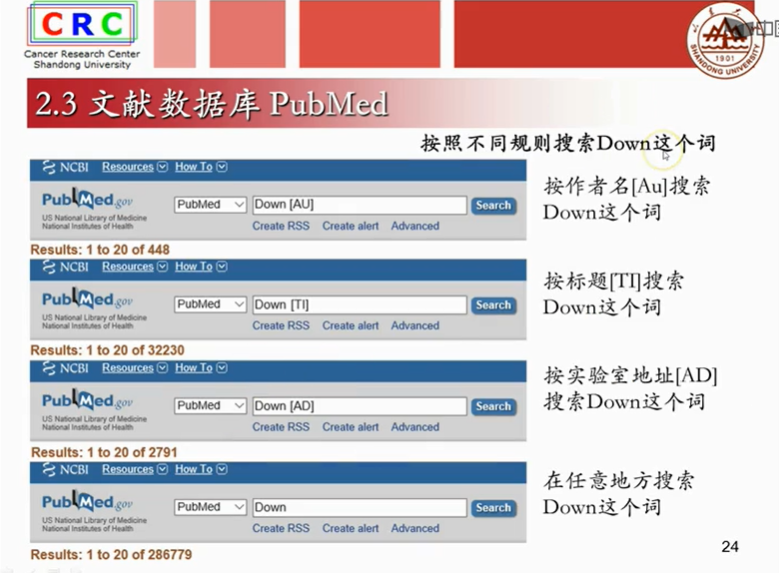

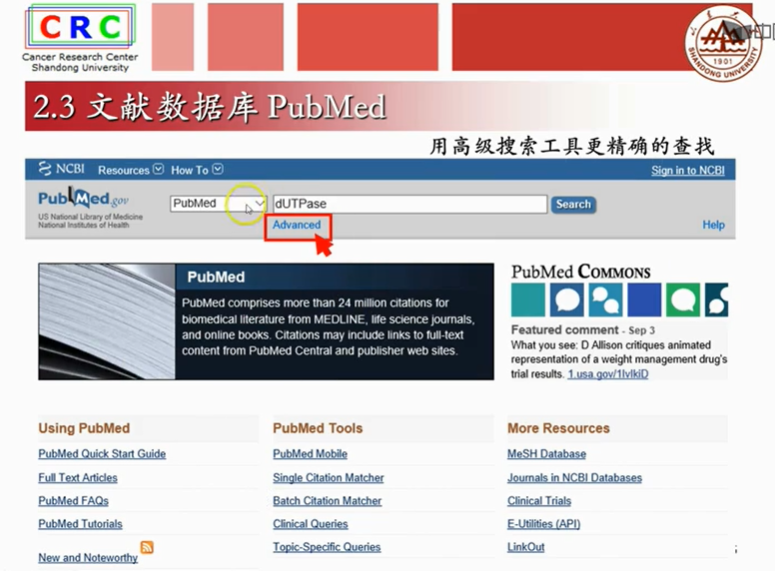
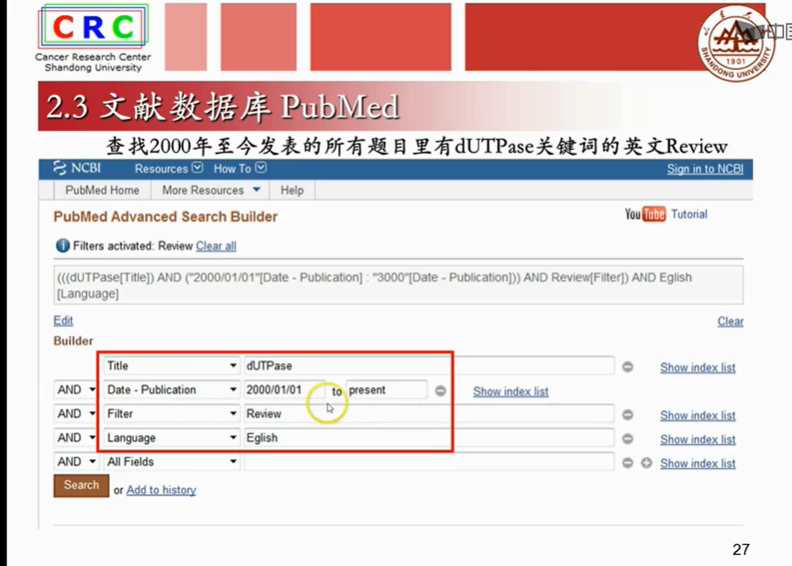


2.4一级核算数据库GenBank
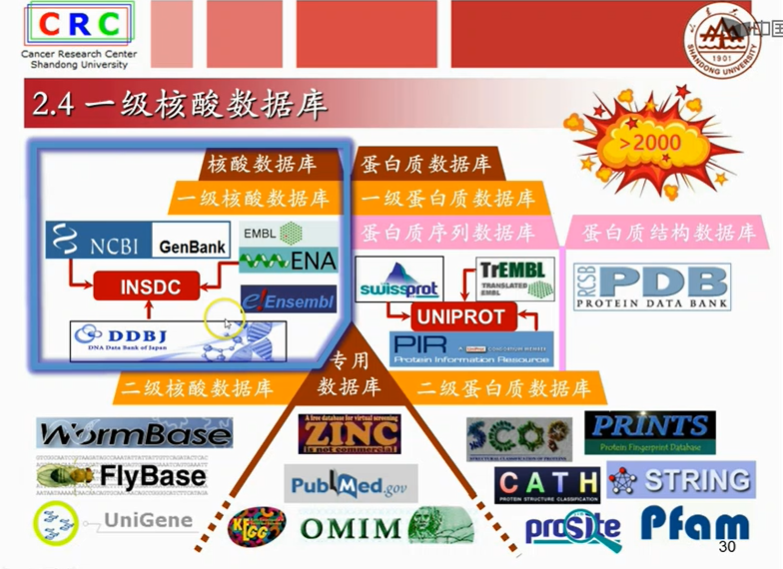
它主要包括,三大核算数据库和基因组数据库
 三大核酸数据库,共同构成国际核酸数据库。
三大核酸数据库,共同构成国际核酸数据库。
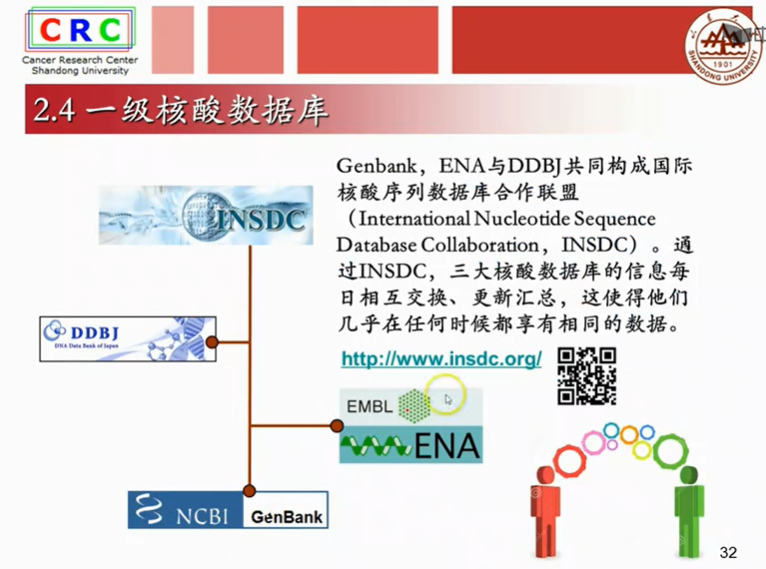
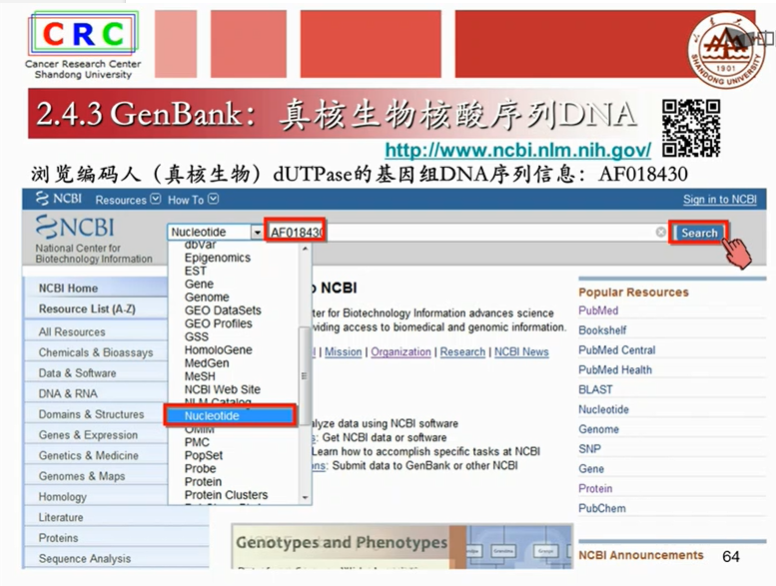
以基因Bank数据库为例,我们来看一个核酸序列的例子:

bp是碱基的意思。A T G C,腺嘌呤、胸腺嘧啶、鸟嘌呤、胞嘧啶。
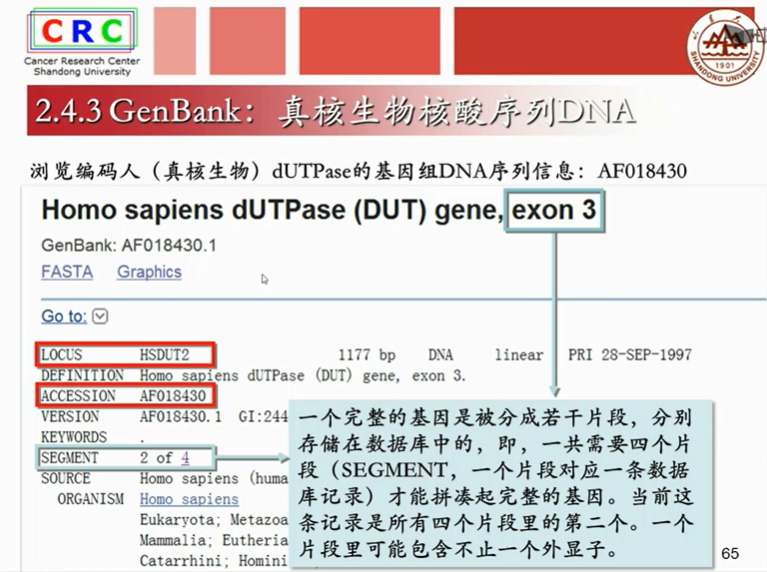
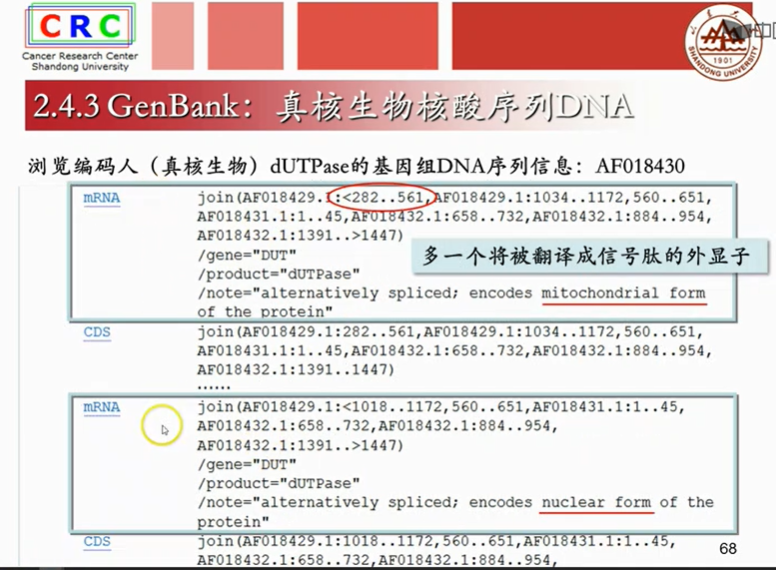
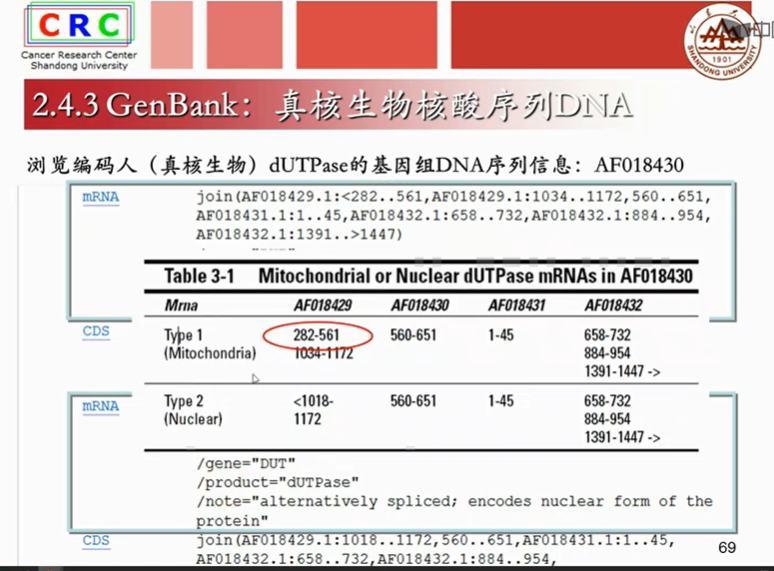
真核生物的基因不是线性分布的,是因为翻译蛋白质的外显子被内含子分割开来,也就是真核生物的mRAN(又称为信使RNA,是bai由DNA的一条链du作为模板转录而来的、携带遗传信息的能zhi指导蛋白dao质合成的一类单链核糖核酸。)要经历剪切的过程,剪切后的成熟mRNA才能进行翻译。
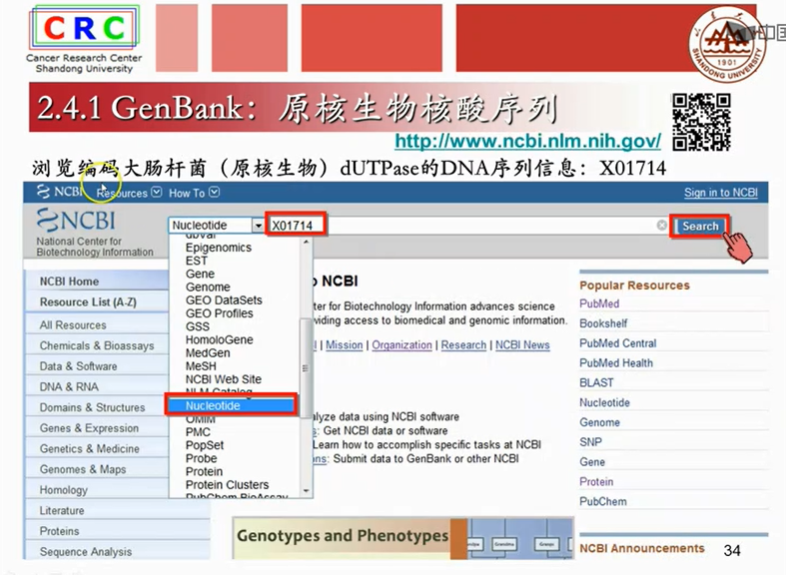
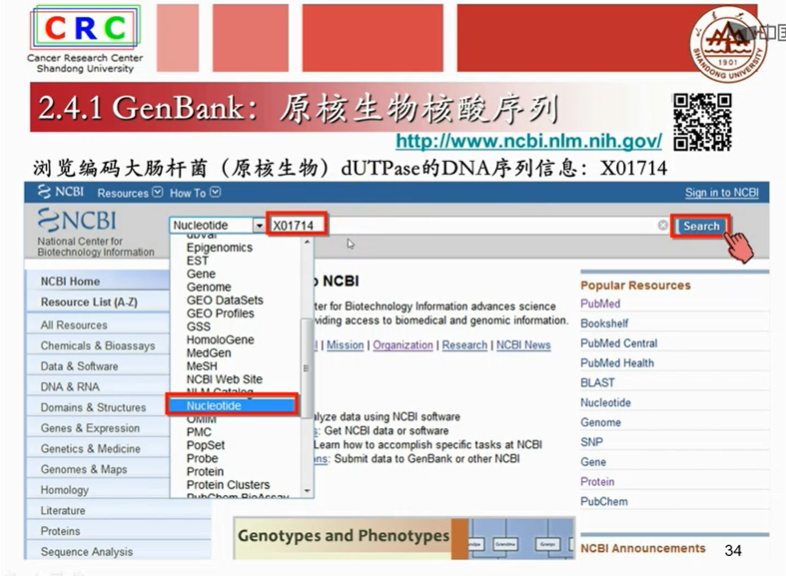
基因编号:X01714
Nucleolide就是GenBank数据库
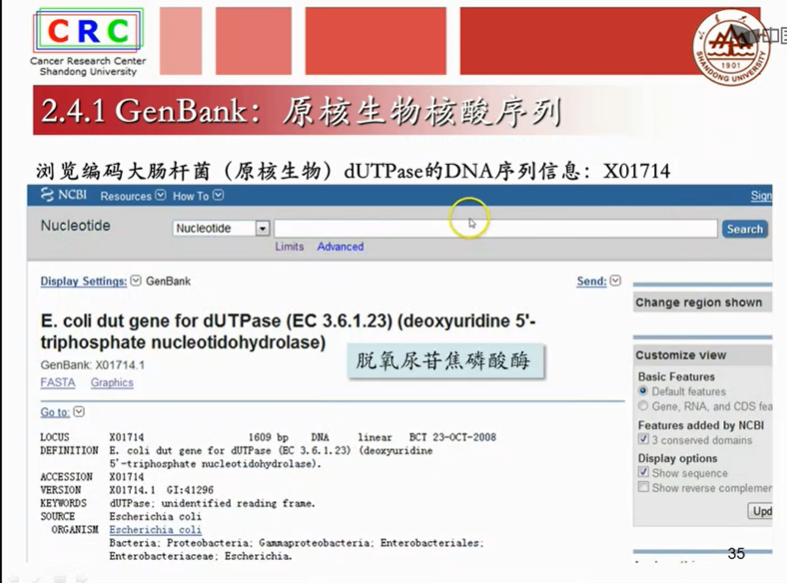

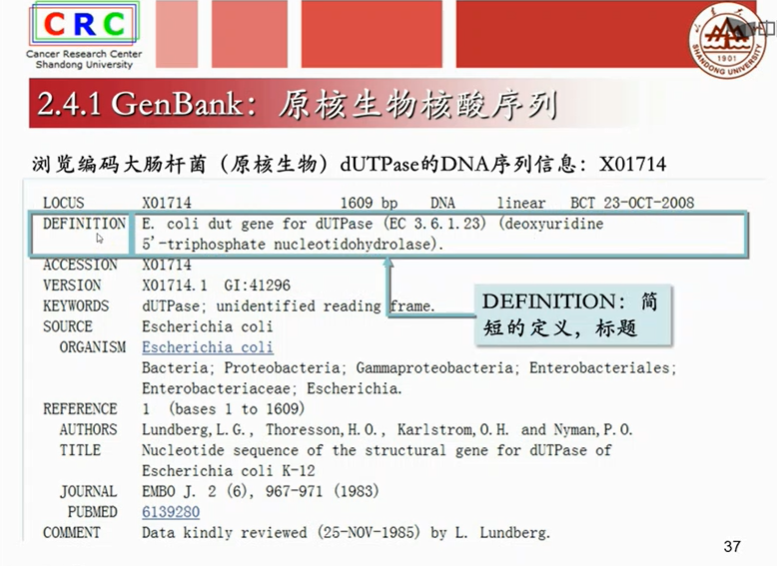

Locus是一个同学的真实名字 而ACCESSION是这个同学的学好,同一个学生在不同的学校会有不同的学好。而名字只有一个。基因也一样。同一个基因在不同的数据库当中会有不同的检索号,而基因的名字只有一个。
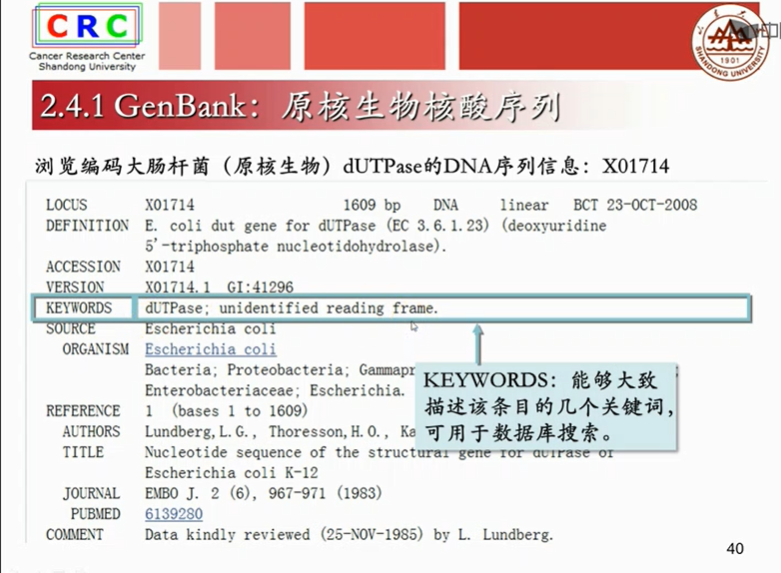
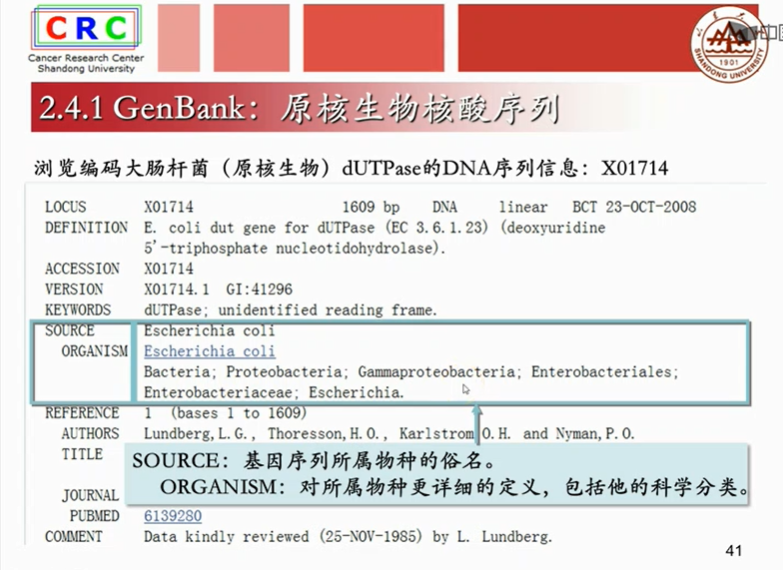

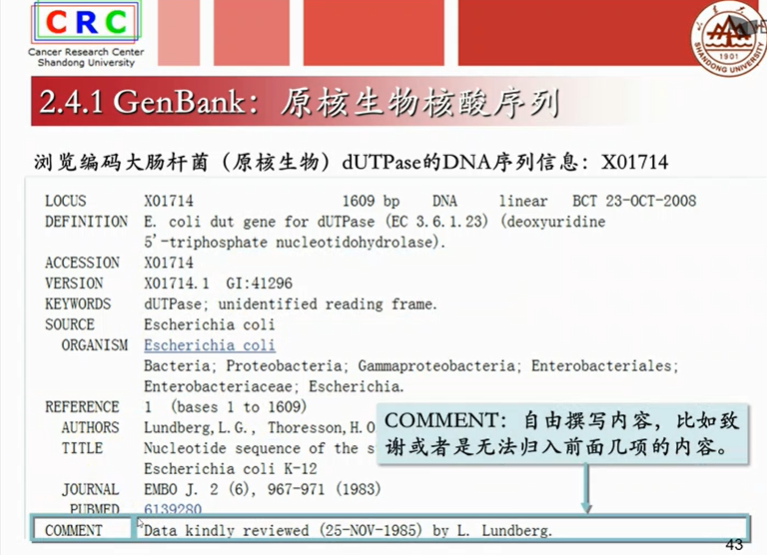
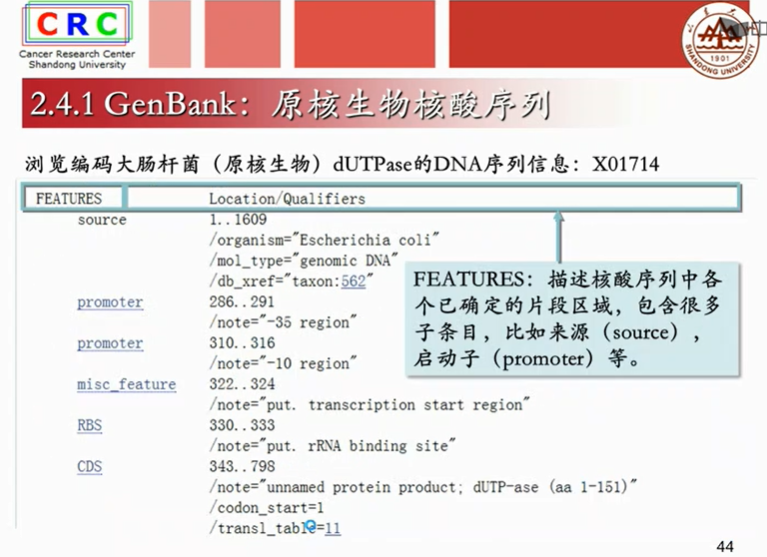
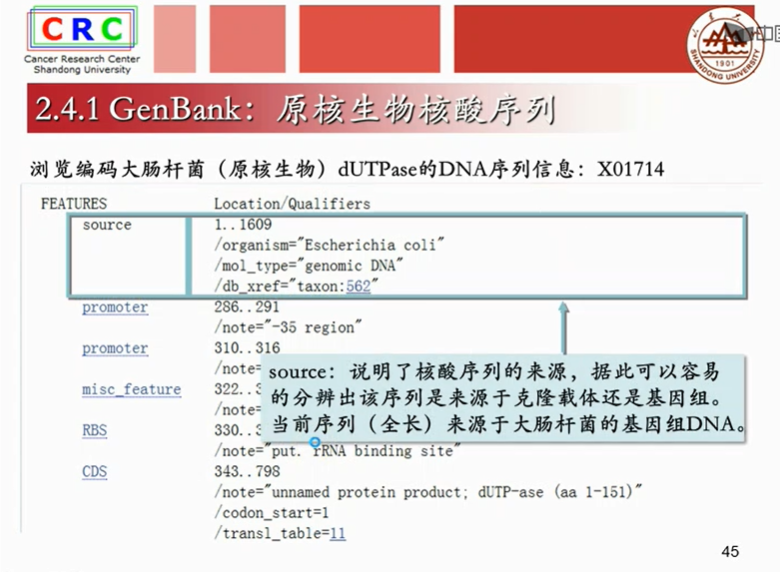
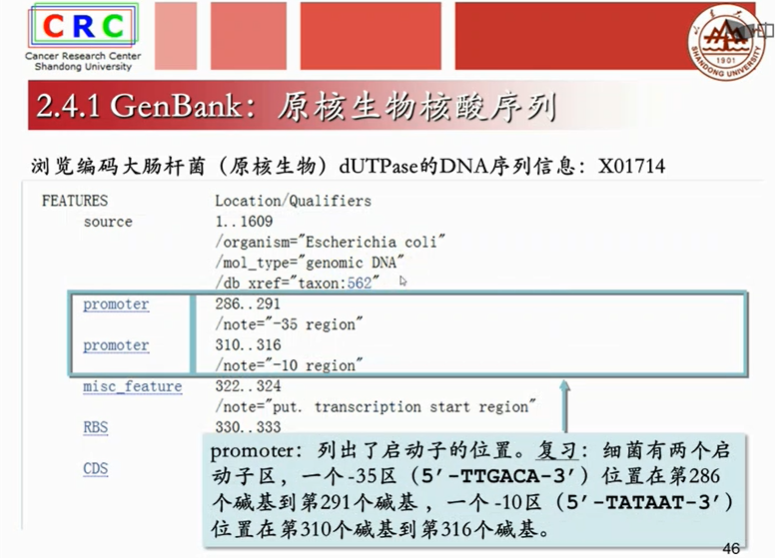
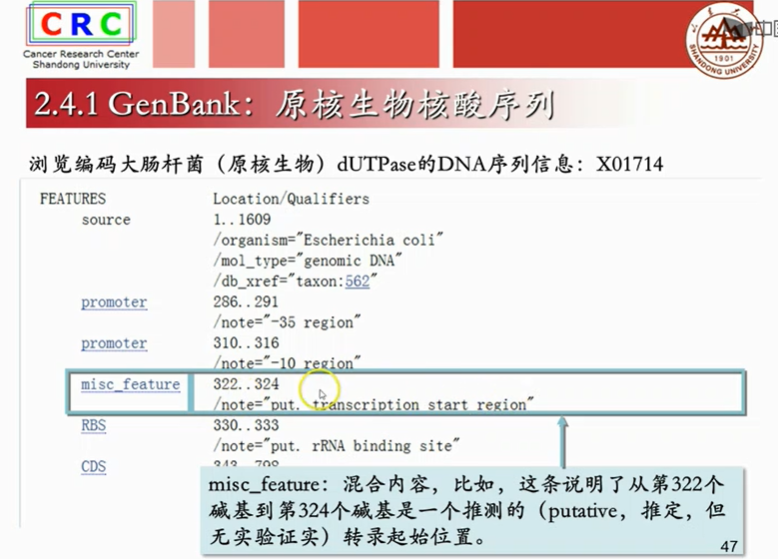
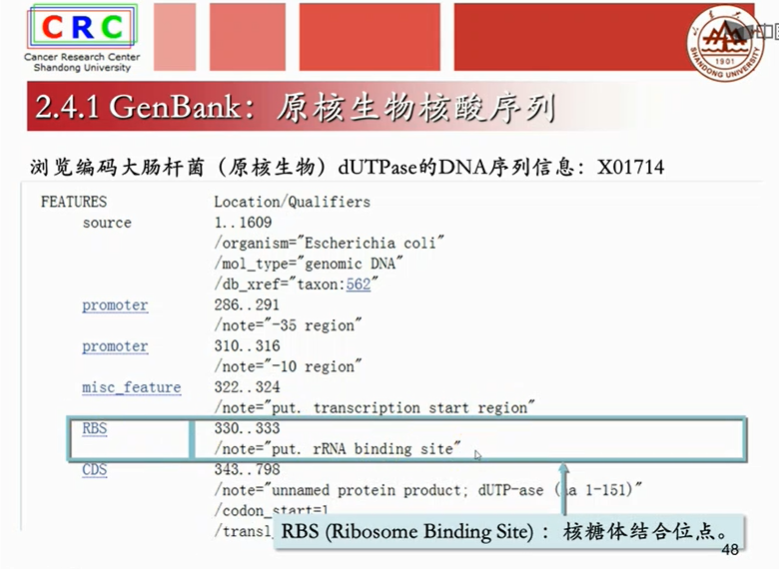
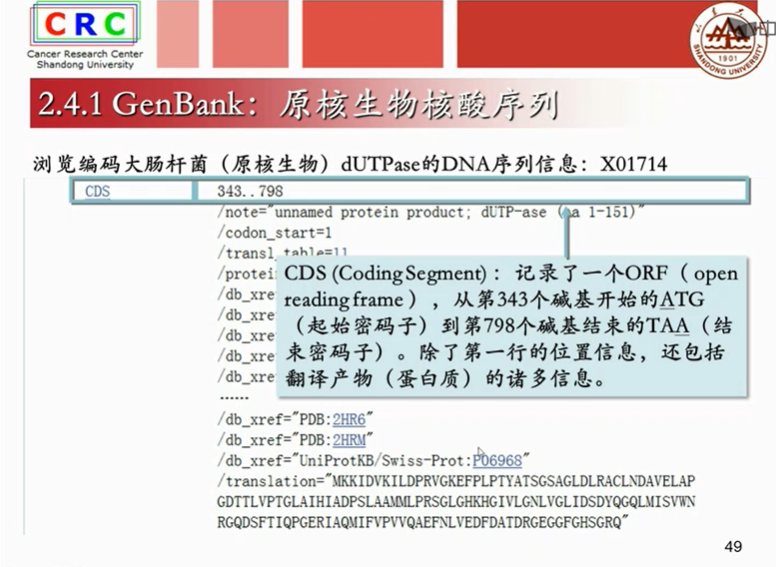
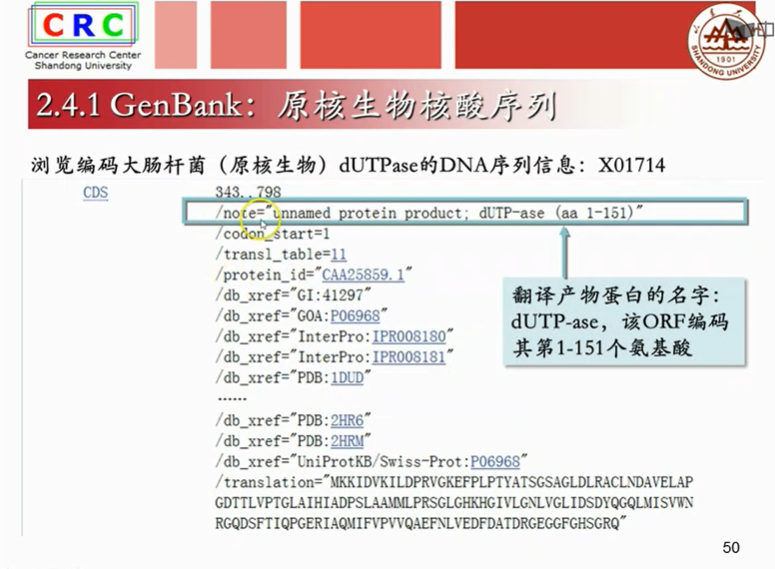

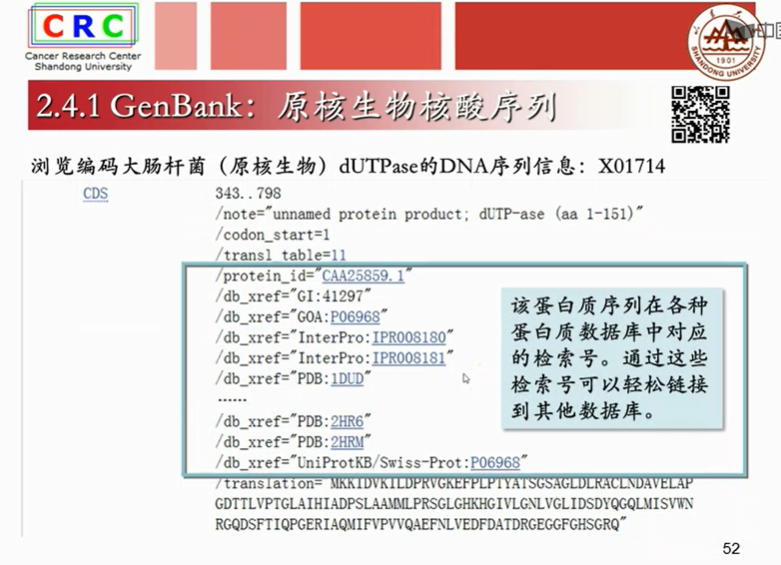
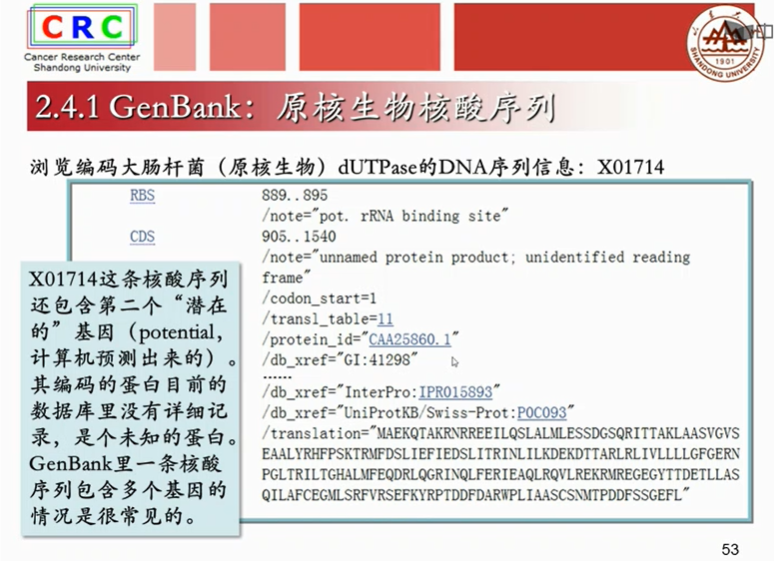

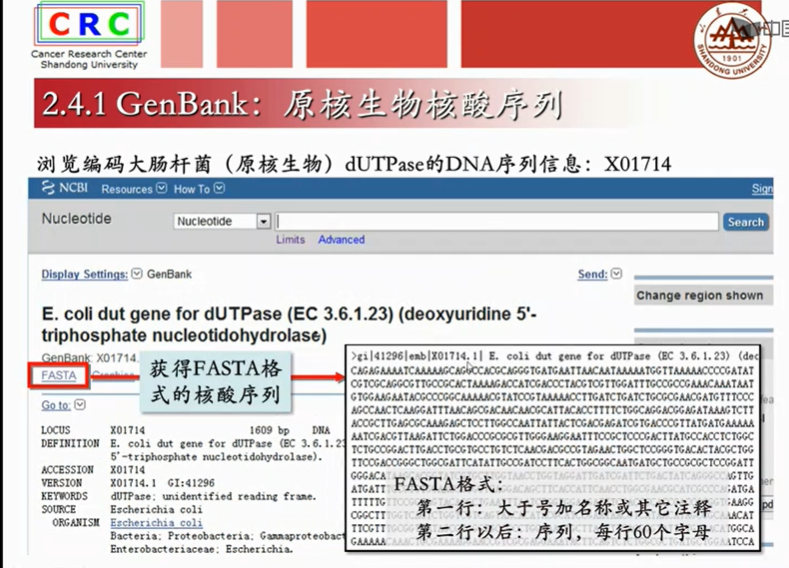
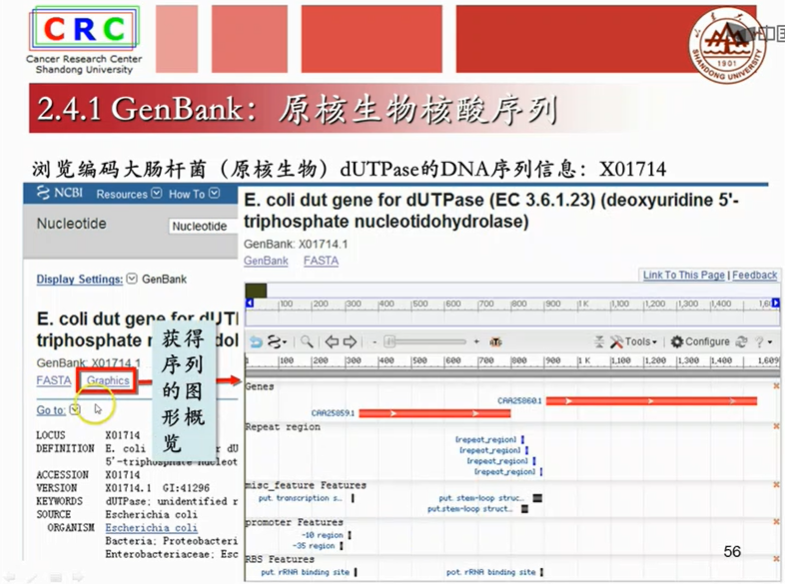

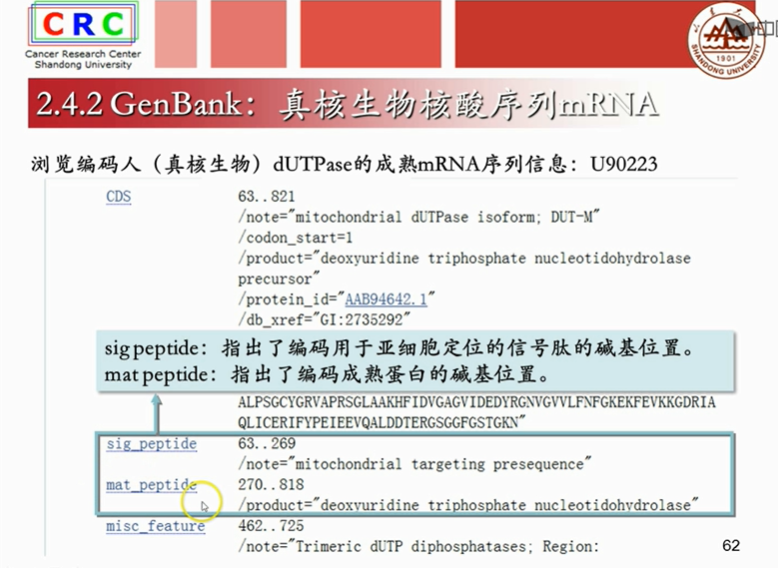
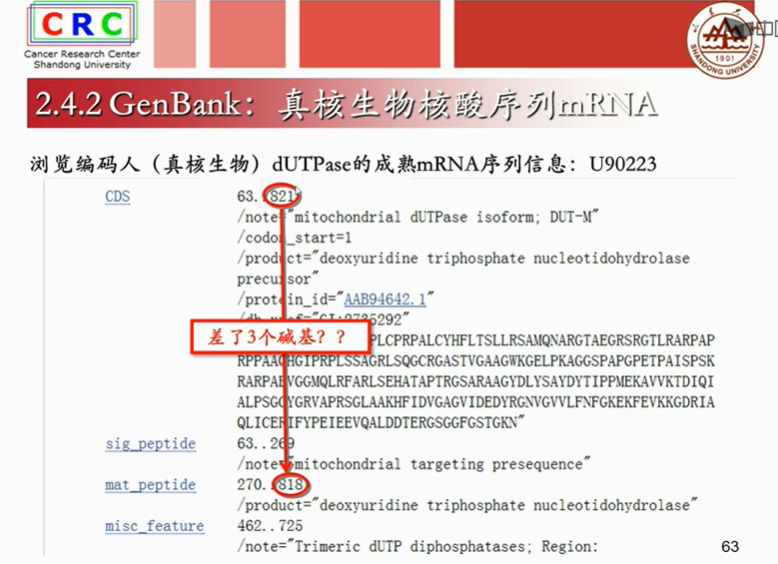
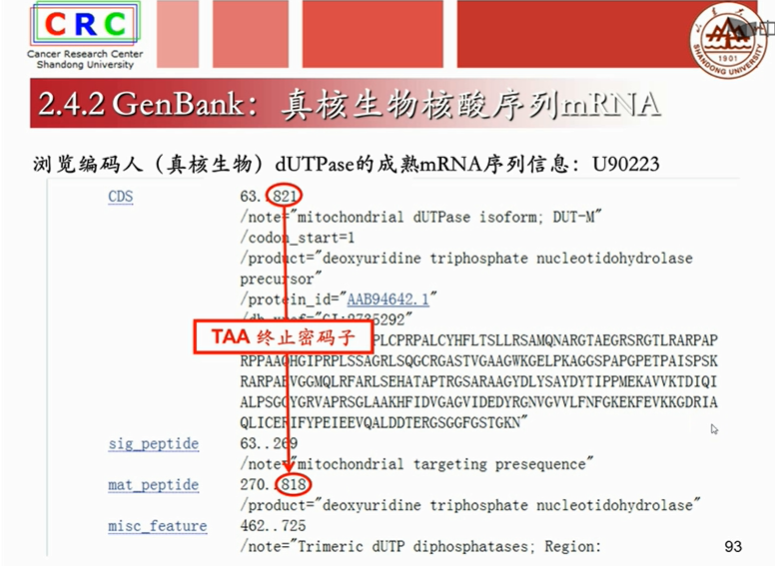
成熟mRAN是已经剪切掉内含子,只剩下外显子的序列。


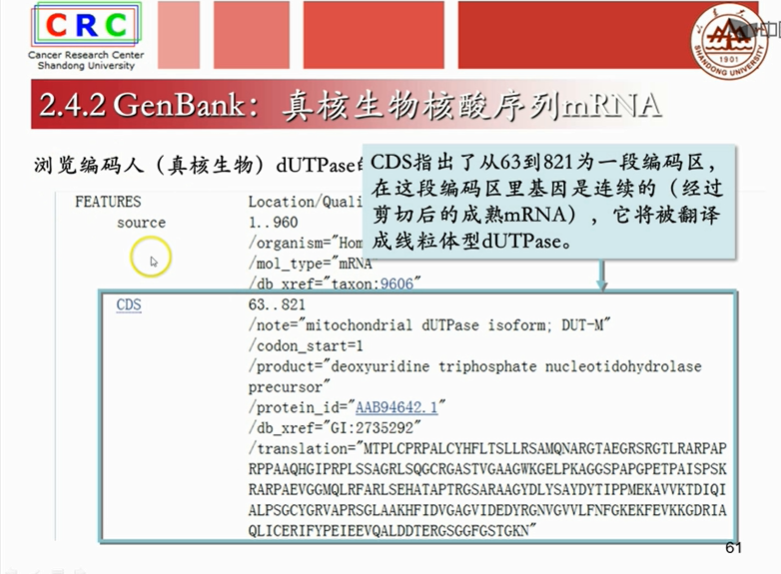
translation:是计算机翻译出得蛋白质序列




2.5一级核酸数据库:人的基因组数据库 Ensemble


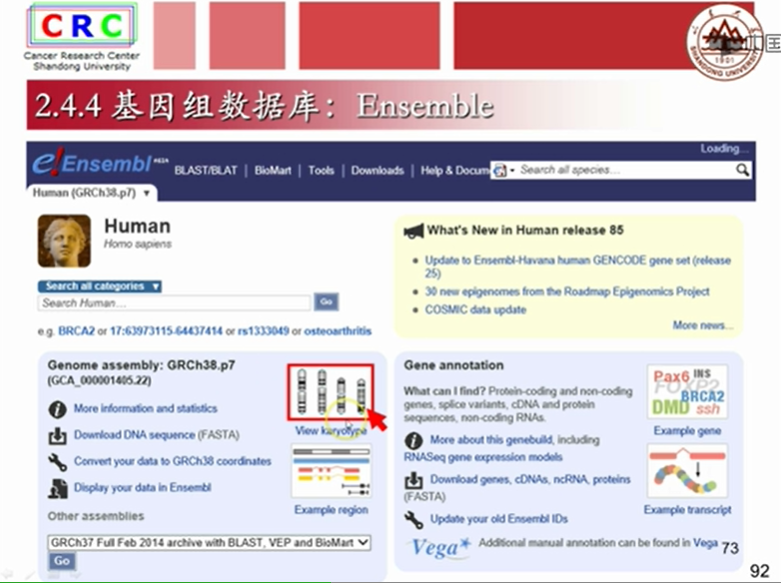
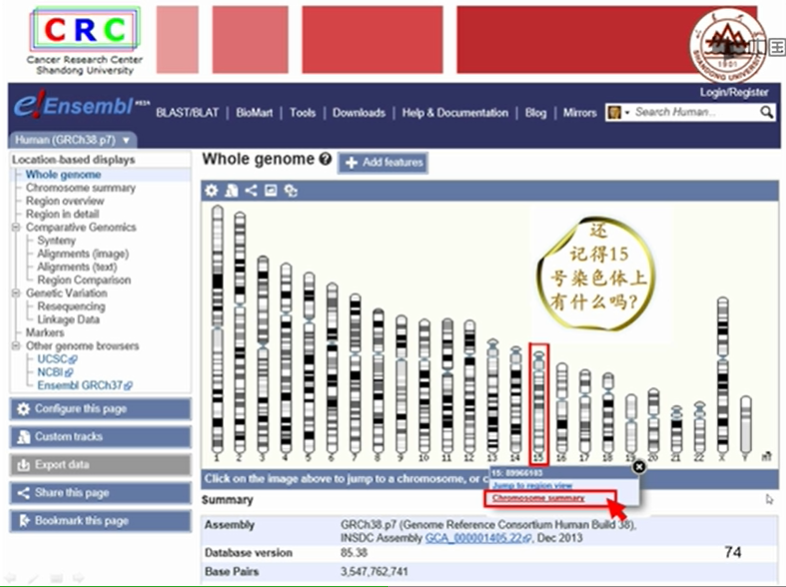
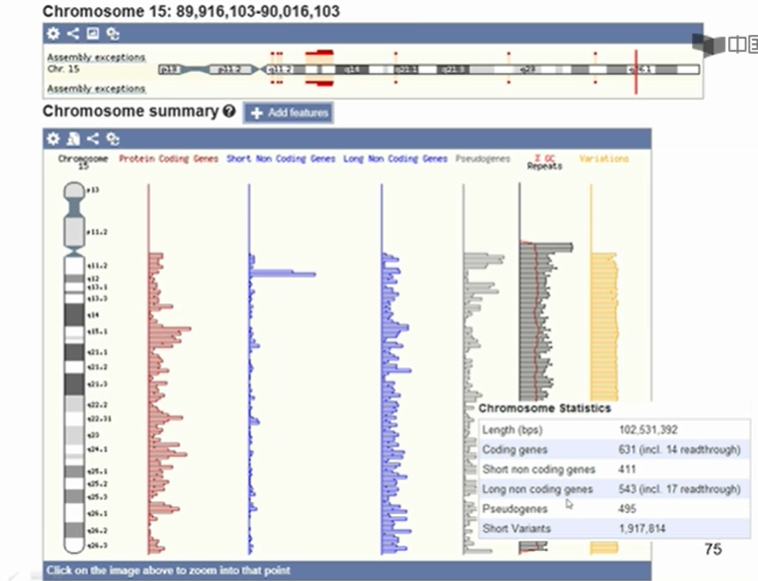
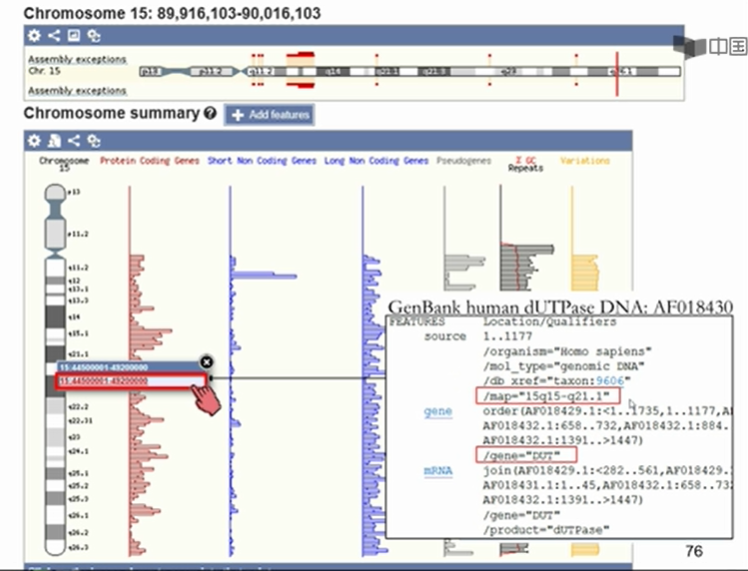
自左向右:编码蛋白的基因、非编码基因、假基因分别在染色体不同区段内的含量、GC百分比红线、未经百分比黑线
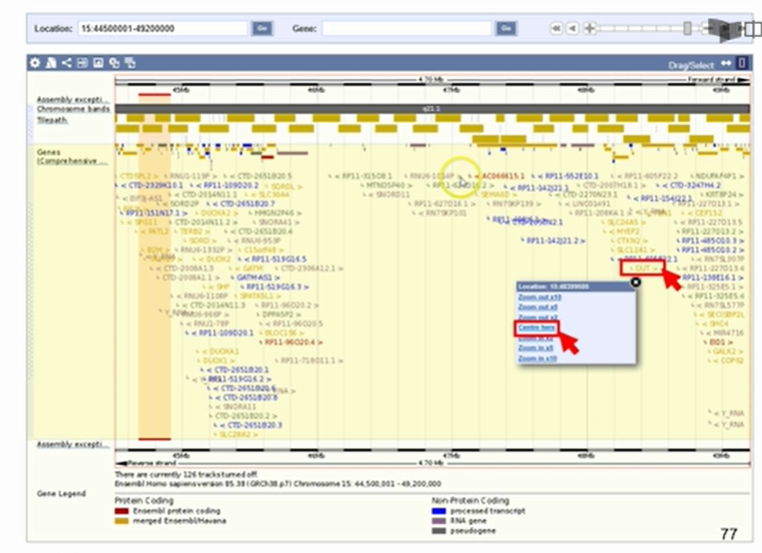
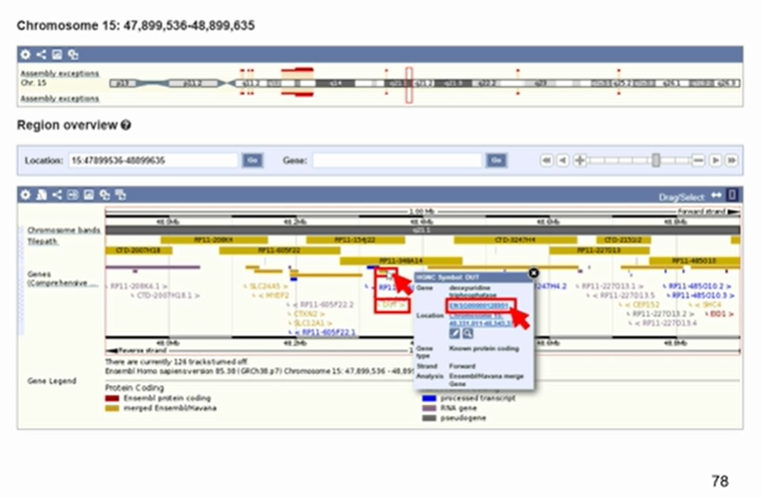
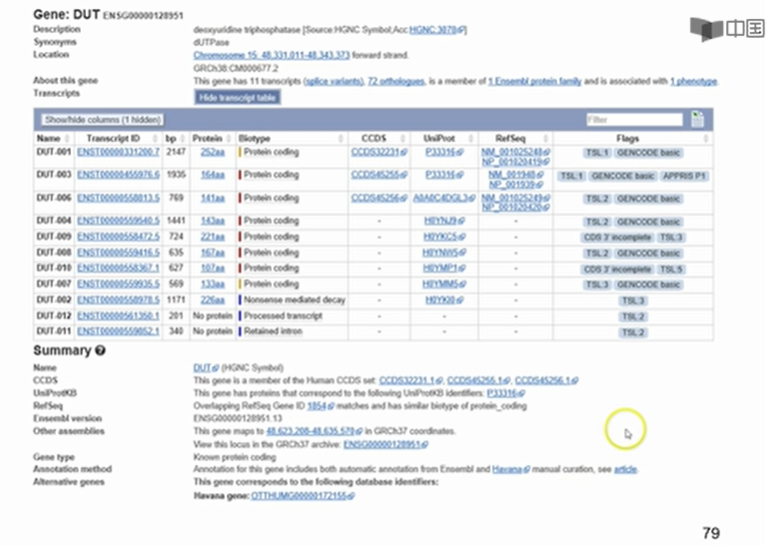
2.5.2 一级核酸数据库:微生物宏基因组数据库 JCVI

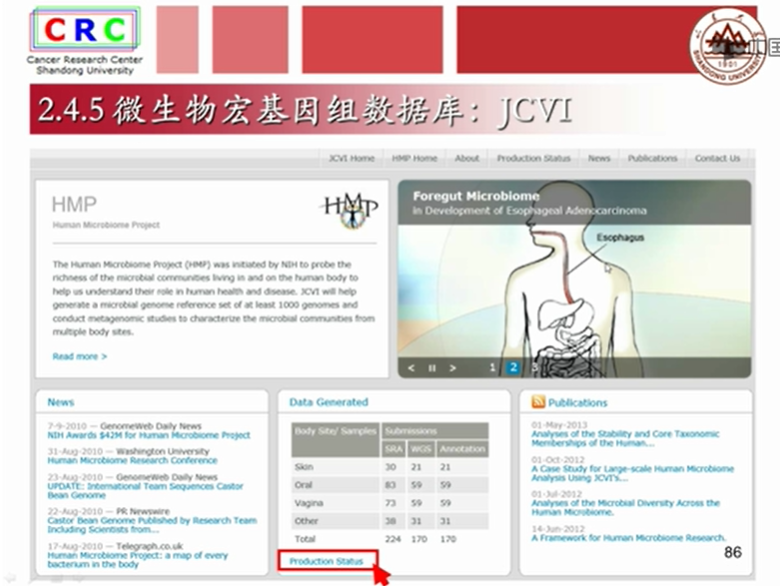

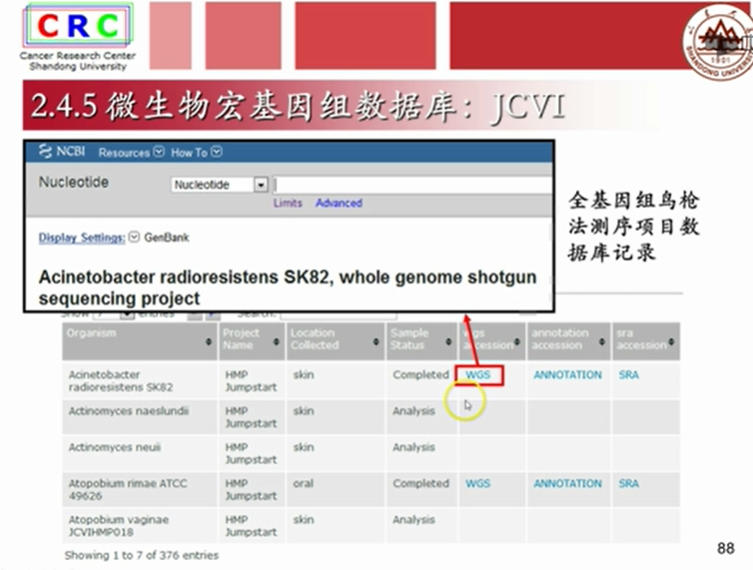
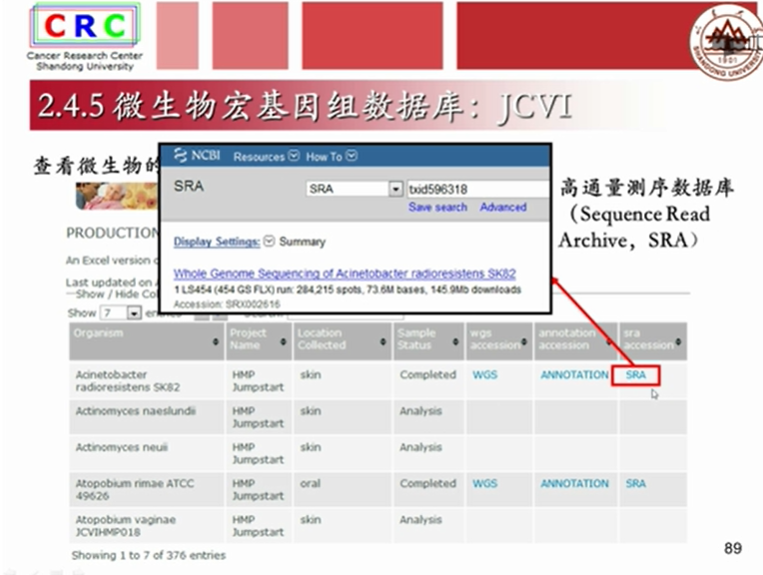
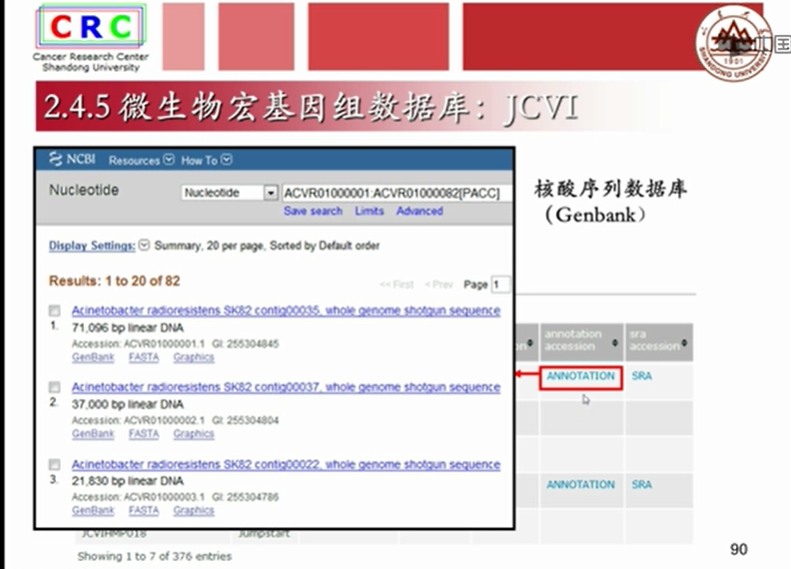
2.6二级核酸数据库

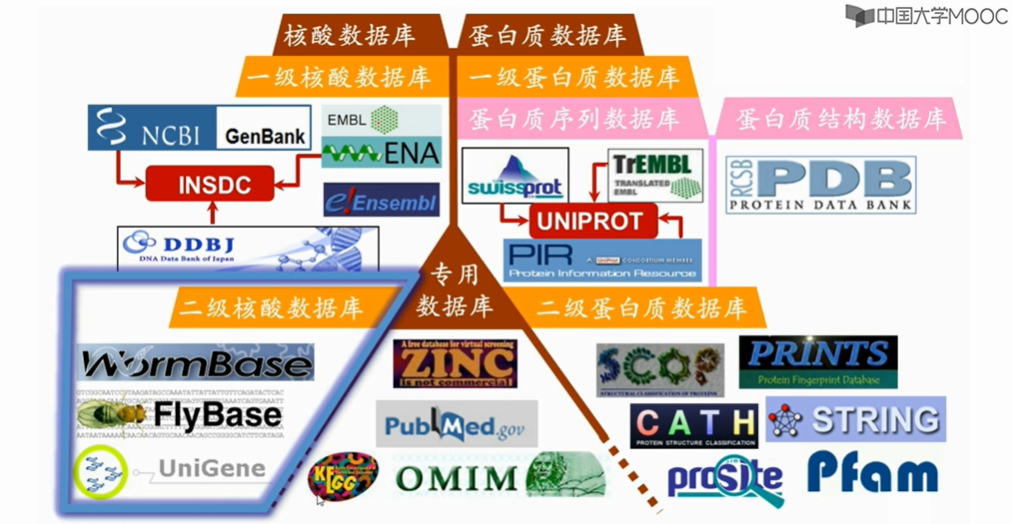


第二章:生物数据库(第二部分)
第三章:序列比较 第一部分
3.1 认识序列



3.2序列相似性

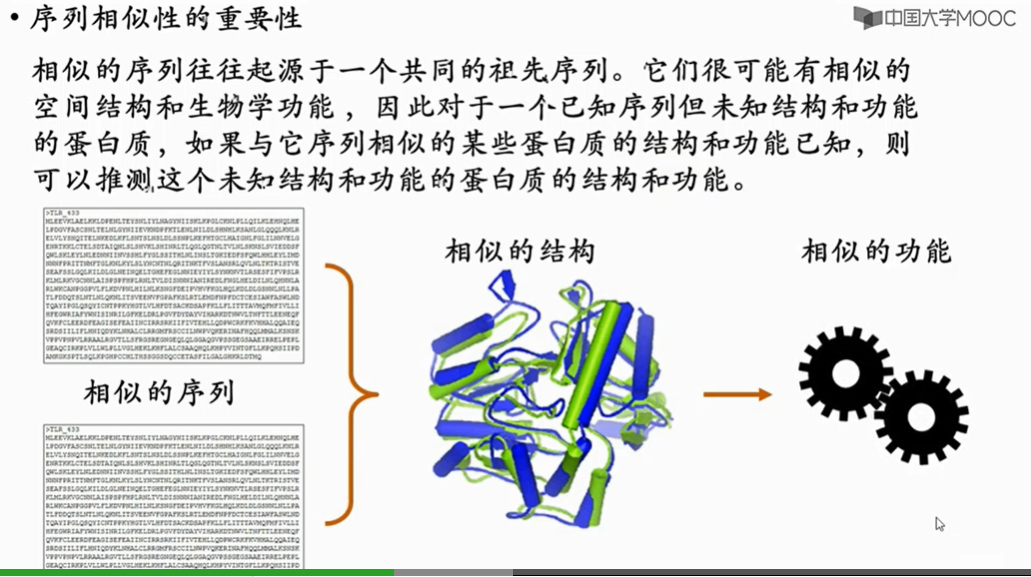

上图的相似没有办法量化,但是序列的相似度我们是有办法进行量化的。

3.3 替换计分矩阵
3.3.1 DNA替换积分矩阵



3.3.2 蛋白质的替换计分矩阵


我们通过所比较序列之间的亲缘关系的远近,来选择比较适合的矩阵。如果序列之前亲缘关系远,也就是说序列会有很多突变,那就选PAM后面跟一个大数字的矩阵,如果亲缘关系近,也就是突变比较少,序列间大多数地方都是一样的,那就选PAM后面跟一个小数字的矩阵,

由介绍可知,BLOSUM后面跟一个小数字的矩阵,适合用于比较相似度低的序列,也就是亲缘关系远的序列,而BLOSUM后面跟一个大数字的矩阵,适合比较相似度高的序列,也就是亲缘关系近的序列。BLOSUM后面的数字可以理解为相似度。 PAM后面的数字,体现的是序列的差异度,但不直接等于差异度,BLOSUM后面的数字体现序列的相似度,并且直接等于相似度。





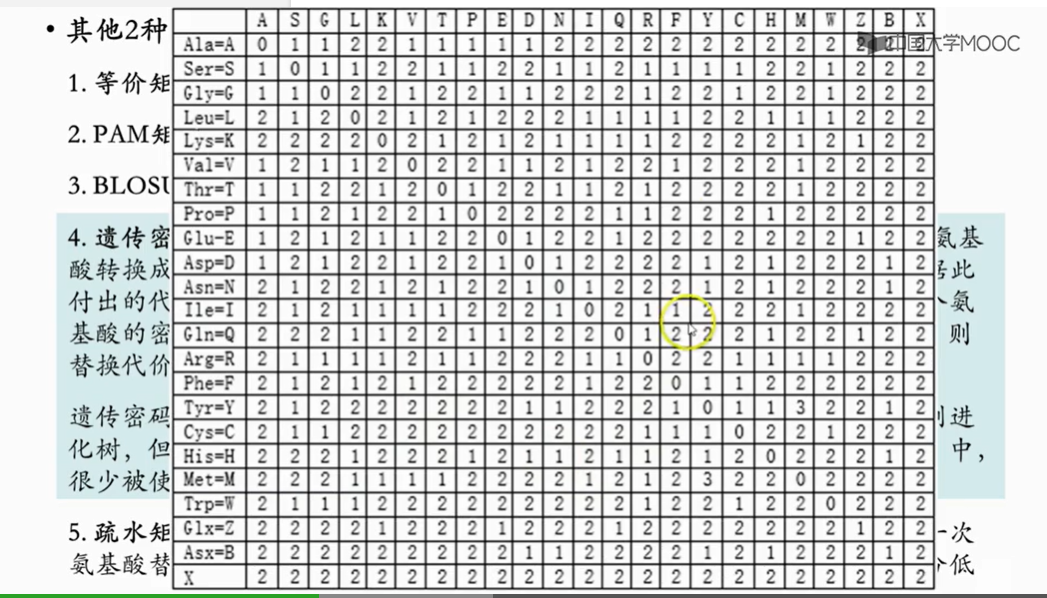

在疏水矩阵里,氨基酸按照亲疏水性排列,前面是亲水的,后面是疏水的,疏水矩阵目的和意义明确,有一定的理化性质依据。




在解决上述问题之前,我们先需要系统的学习一下比较两个序列的方法,之后这个问题便会迎刃而解。
3.4 序列两两比较:打点法
3.4.1 打点法介绍以及用途


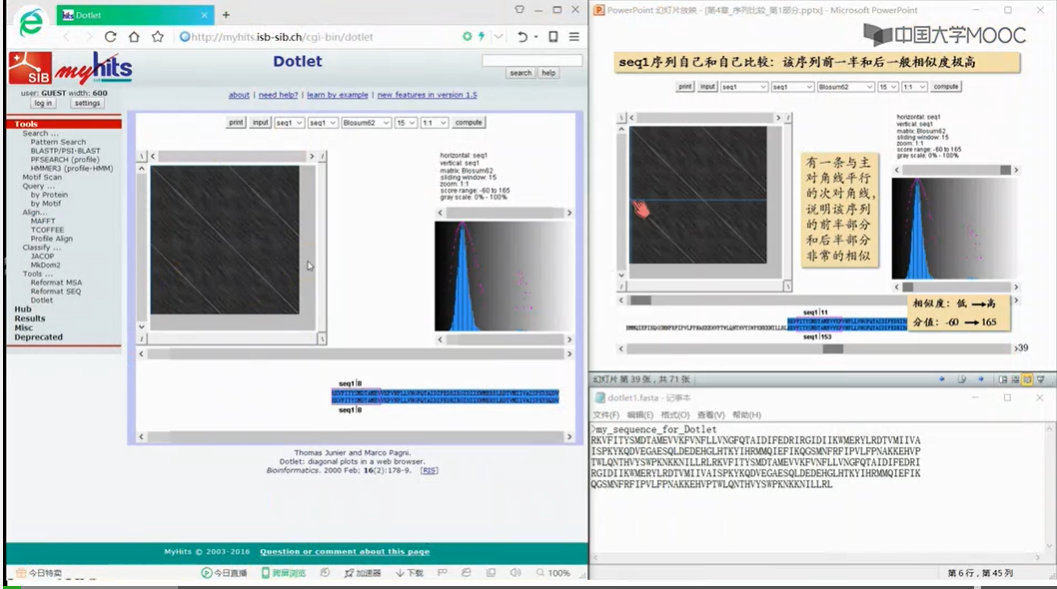
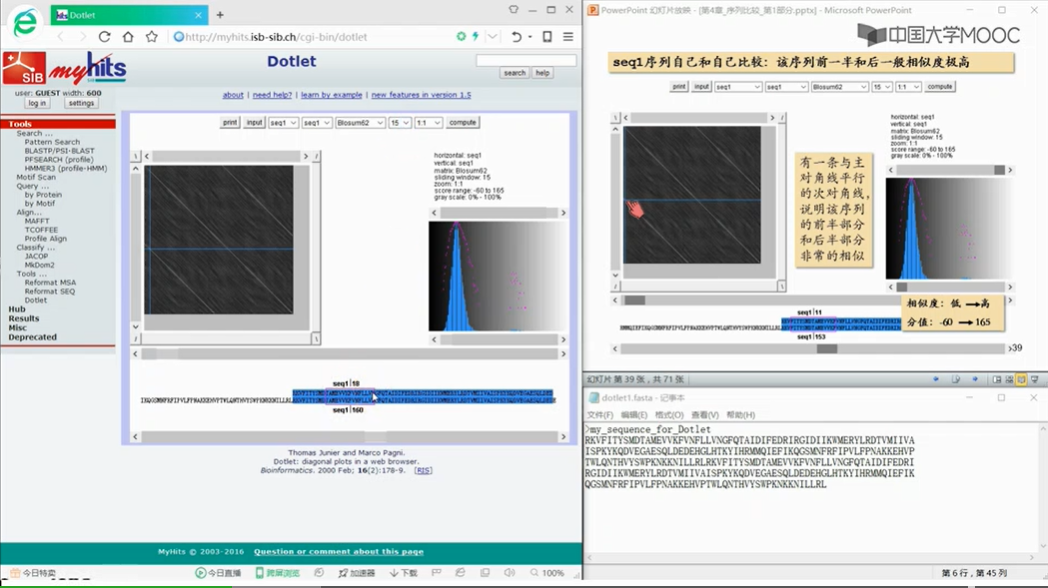
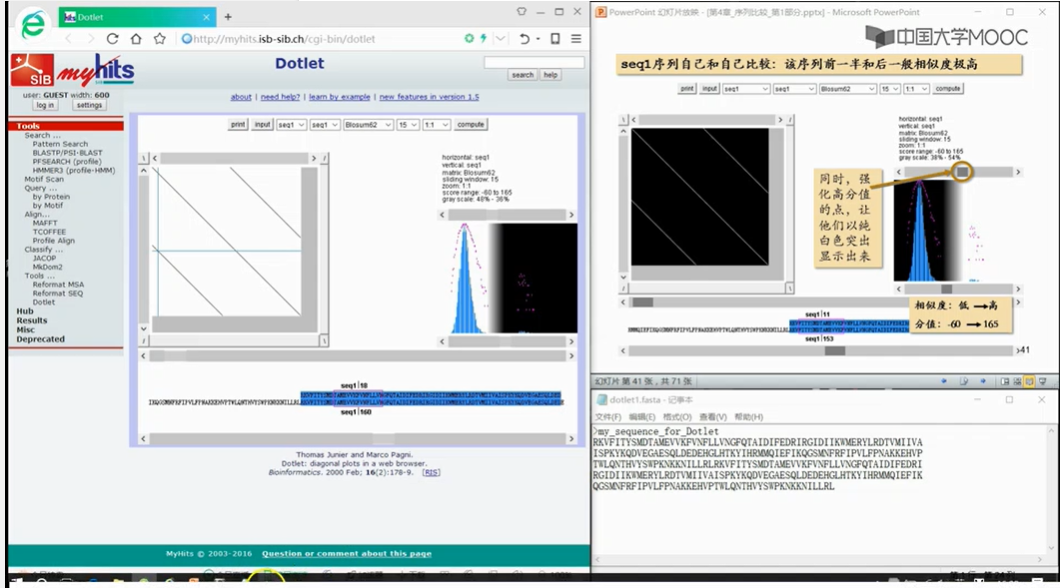
发现:这个矩阵中绝大部分地方是没有点的。
发现:这个矩阵打点打出了一个对角线

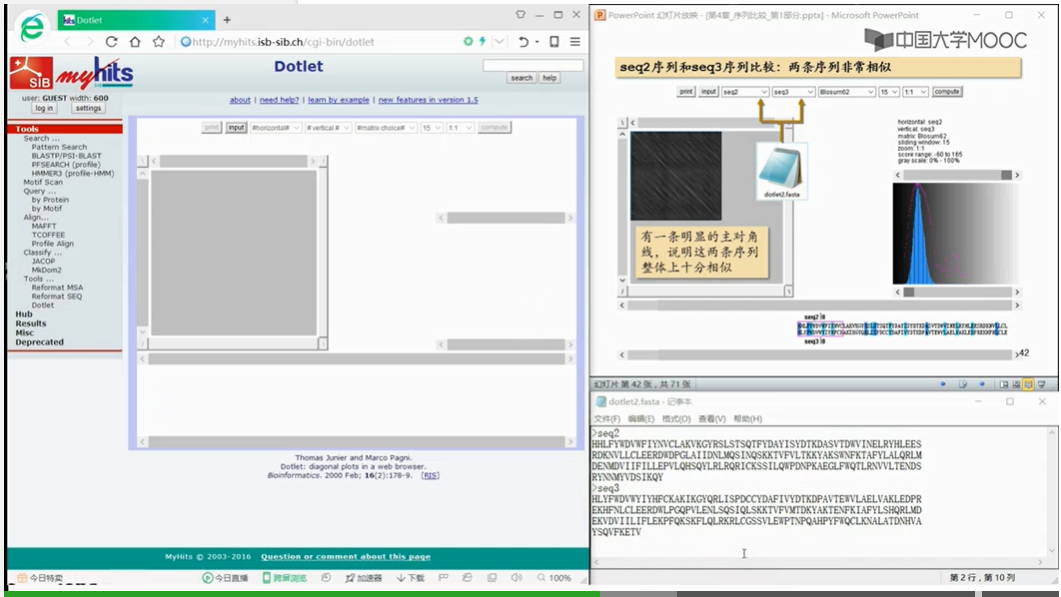
放眼望去,红色的线和蓝色的线,加起来,基本上构成了一条主对角线,由此我们可以得出结论序列一和序列二是比较相似的两条序列。

上图序列1和序列3就不相似。

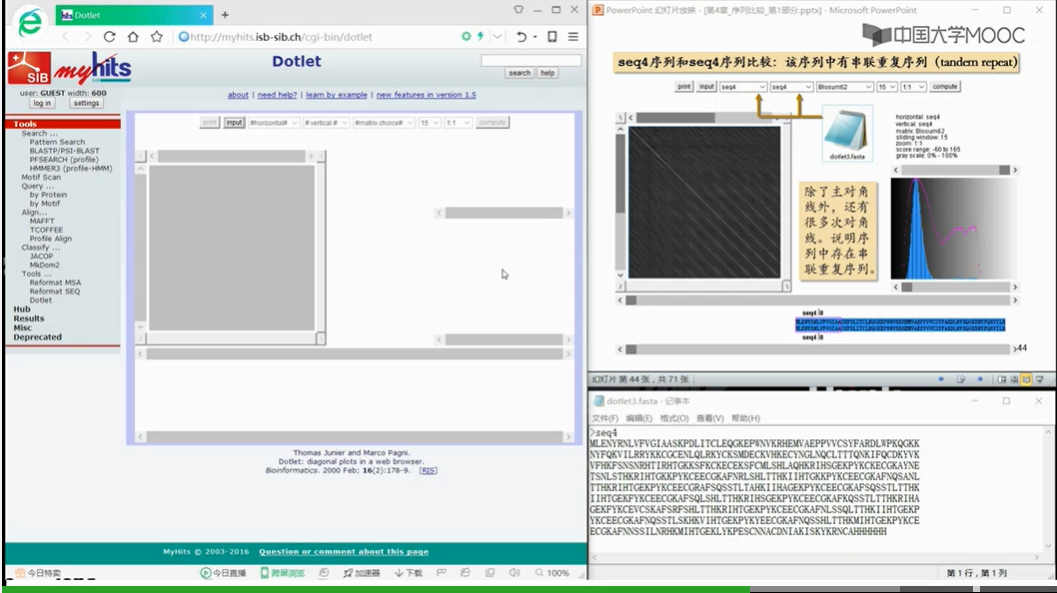
上图中必有一条主对角线,此外,在横向或纵向上,与主对角线平行的短平行线所对应的的序列片段,就是重复的部分。包括主对角线在内的平行线出现的次数就是重复的次数,如图,THE出现了三次。
用打点法对序列本身进行打点,可以发现序列中的串联重复序列,及其重复个数。我们只需要数一数在半个矩阵当中,包括主对角线在内所有等距的平行线的个数。就可以知道重复的次数了。而且最短的平行线对应的序列,就是重复单元。短的串联重复序列,具有高度多态性,也就是说不通的个体间,重复次数存在差异,而且这种差异在基因的遗传过程中一般遵循孟德尔共显性遗传规律。所以快速的查询某些特定的短的串联重复序列的重复次数,可以用于医学的个体识别和亲自鉴定等领域
3.4.2 在线打点工具界面介绍


https://myhits.sib.swiss/cgi-bin/dotlet

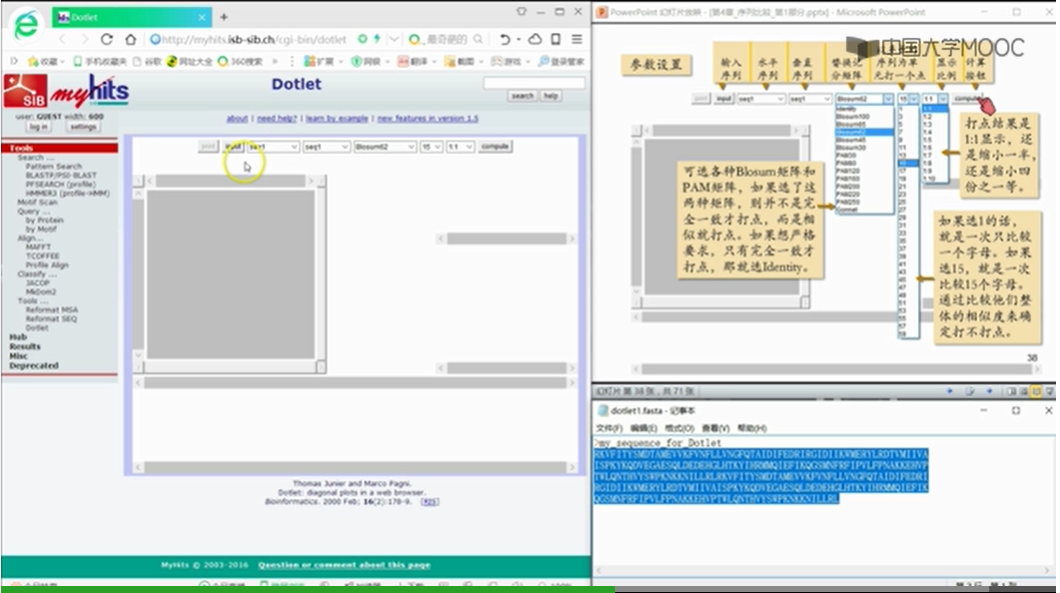
3.4.3 在线打点工具应用实例

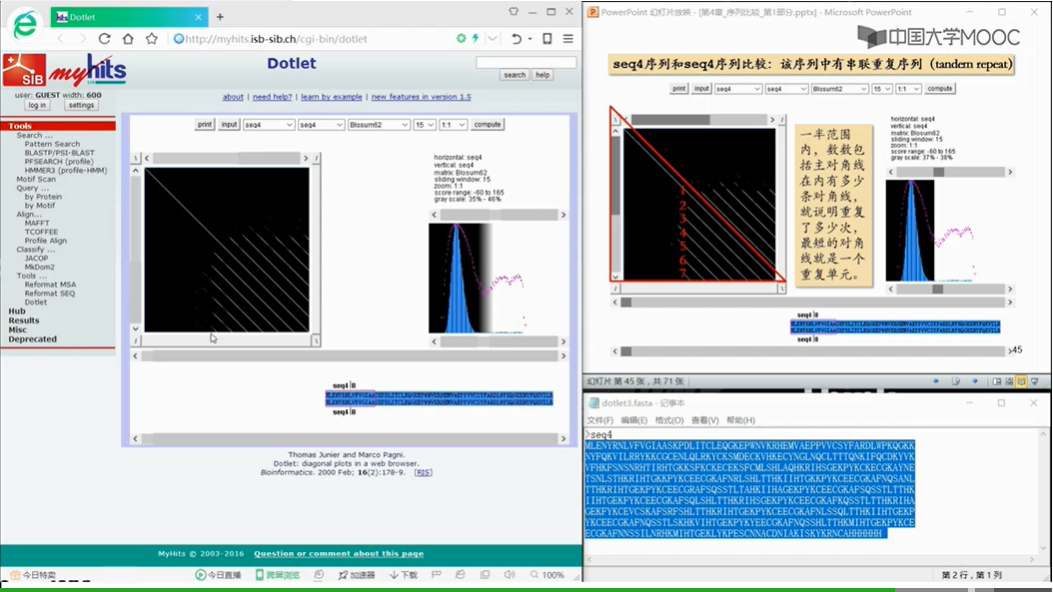
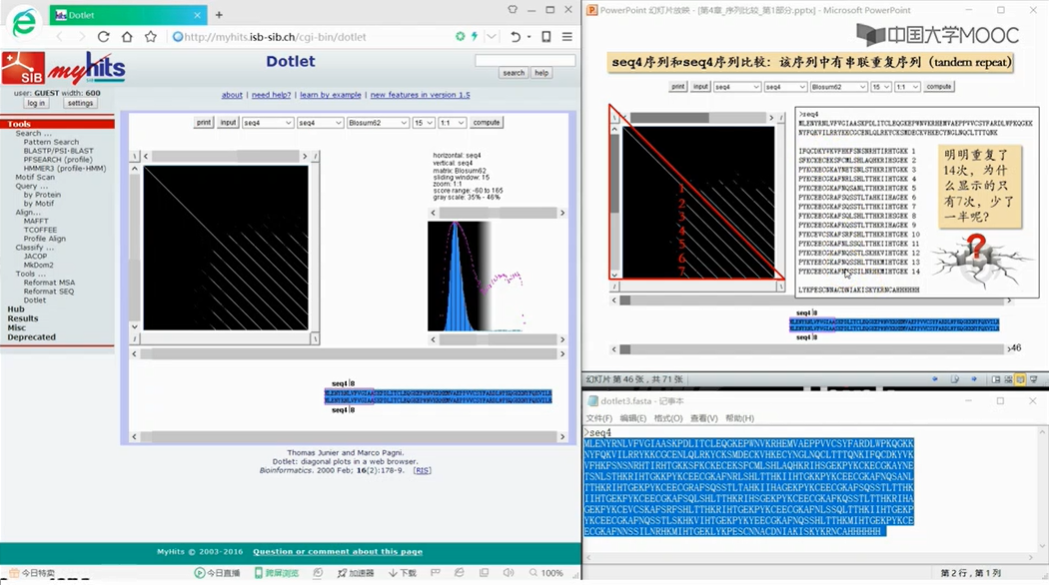
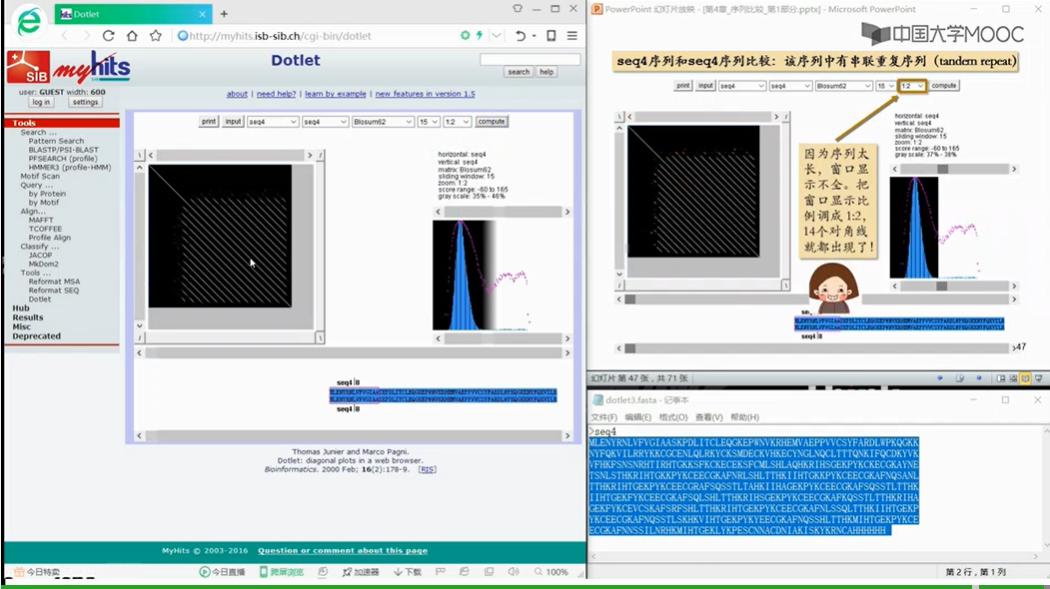
3.5序列两两比较:序列比对法
3.5.1序列比对法


根据序列比对的个数,可以把序列比对分为双序列比对和多序列比对。
另外,根据序列比对的算法不同,双序列比对又分为,全局比对和局部比对。
3.5.2双序列·全局比对及算法
全局比对,顾名思义,就是全长比较,一个都不能落下。而局部比对,是比较对的最好的局部,而对的不好的部分会被忽略不计。

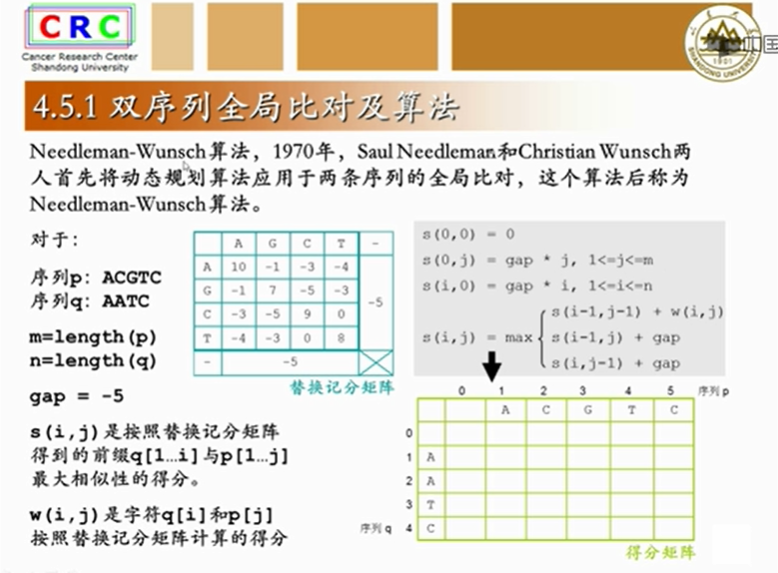 今天,所有比对软件使用的算法,都是从这个经典算法中衍生出来的,下面我们用这个算法来创建序列P和序列Q的全局比对。输入值除了有两条序列以外,还要有替换积分矩阵,来计算不同子母间的相似度得分。另外还要有空位罚分。也就是当字母对空位的时候,应该得几分,我们还是希望一致或相似的字母尽可能地对在一起。字母对空位的情况和相似的字母对在一起的情况一样,都不是我们希望的。还是少出现为好。通常字母对空位负分,称之为空位罚分。在比对中没有空位对空位的情况,空位对空位,纯属瞎占地方。我们根据公式填满得分矩阵。
今天,所有比对软件使用的算法,都是从这个经典算法中衍生出来的,下面我们用这个算法来创建序列P和序列Q的全局比对。输入值除了有两条序列以外,还要有替换积分矩阵,来计算不同子母间的相似度得分。另外还要有空位罚分。也就是当字母对空位的时候,应该得几分,我们还是希望一致或相似的字母尽可能地对在一起。字母对空位的情况和相似的字母对在一起的情况一样,都不是我们希望的。还是少出现为好。通常字母对空位负分,称之为空位罚分。在比对中没有空位对空位的情况,空位对空位,纯属瞎占地方。我们根据公式填满得分矩阵。
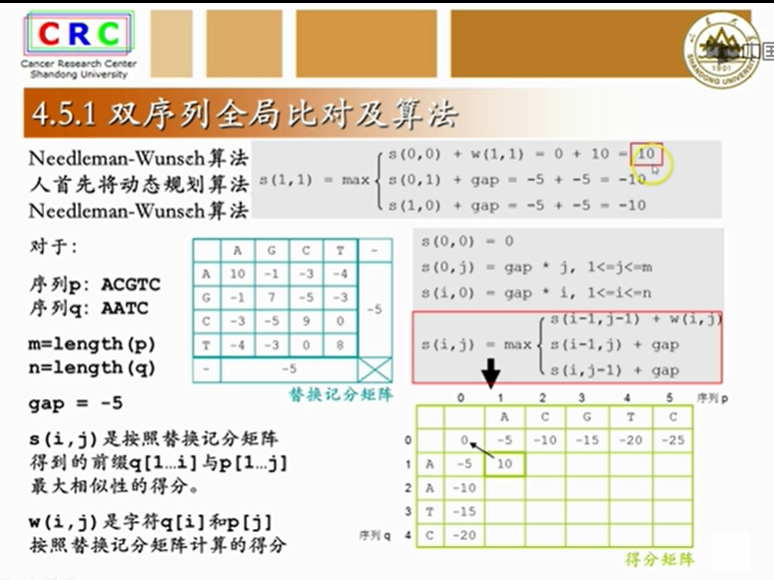
要用箭头指向得分的来源。
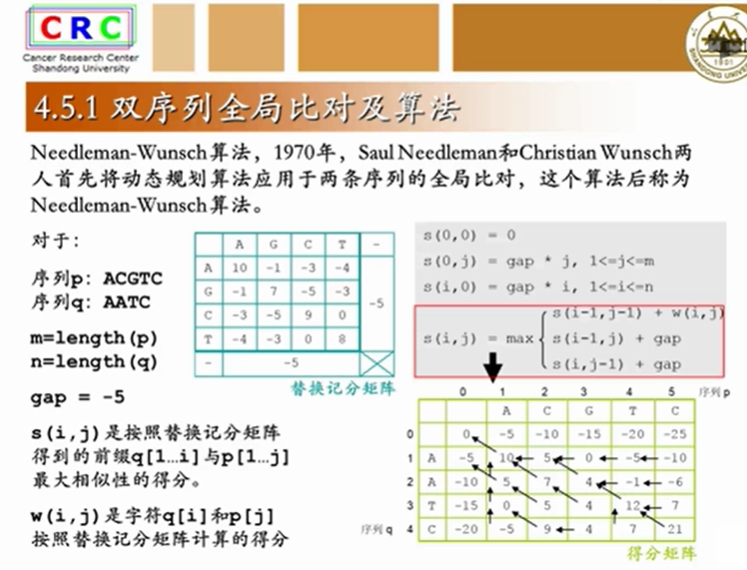
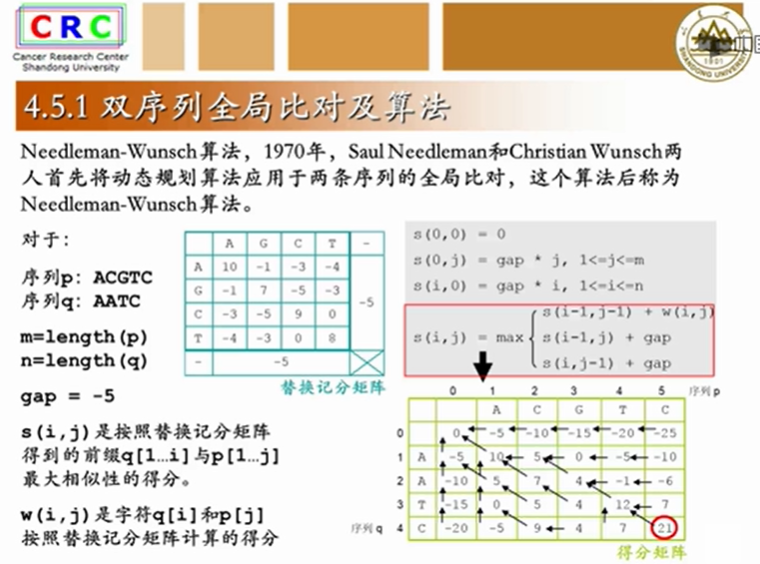
右下角的分数就是整个全局比对最终的得分
从右下角追溯到左上角起始的0,并标注红色箭头,这就是全局比对的唯一依据。
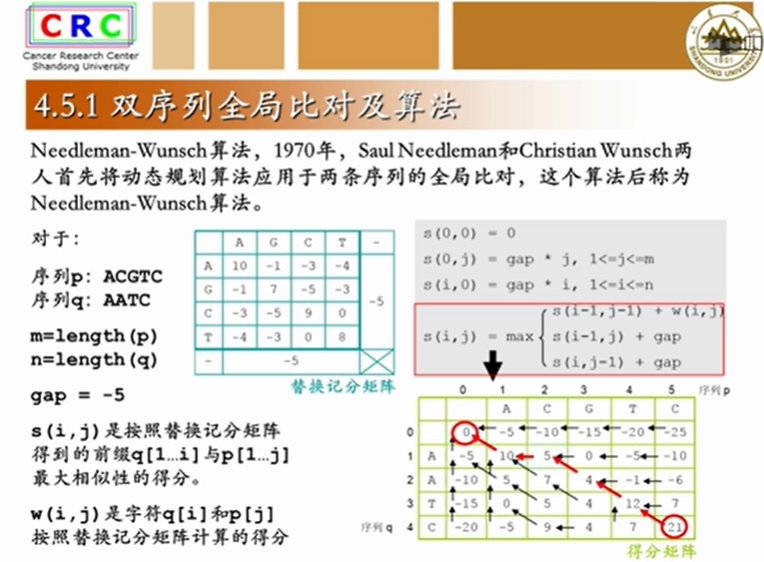
追溯箭头是从右下角开始,写全局比对从左上角开始
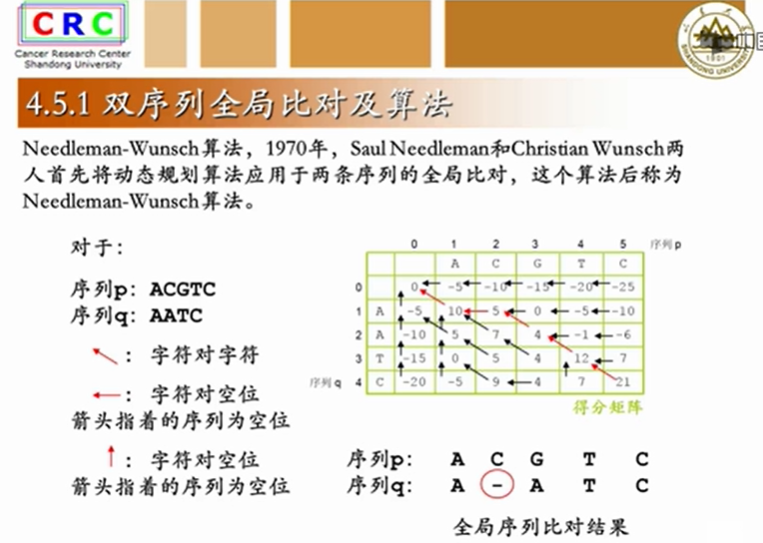
一直写到右下角,全局比对结果就出现了,唯一一个空位,插在序列A与A之间,这样,最终的比对得分最高。不信的话,可以试试,其他任何一种插入空位的比对结果,得分都不会超过21分。因为我们在得分矩阵的创建过程中,每一步都是在上一步最有的情况下,得出当前最优的结果。
3.5.3 双序列·局部比对

像是一长一短的两条序列,比对局部比比较全长更有意义。

和全局比对相似,只是通过在选最大值时,多了一个0,来达到比较局部的效果。
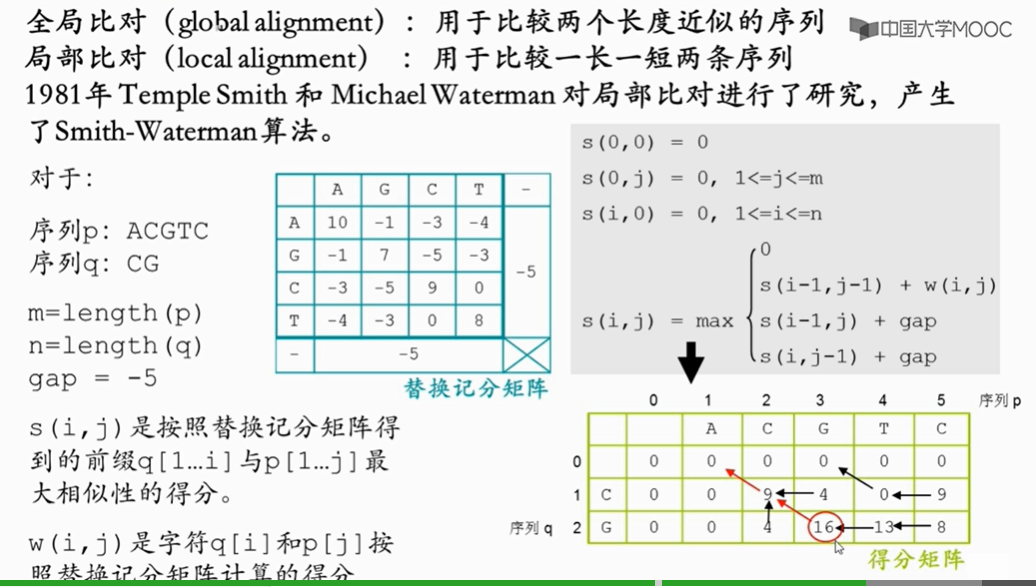
与全局比对不同,局部比对的得分,不是在右下角,而是在整个矩阵中找最大值,这个最大值才是局部比对的最终得分,它可能出现在任何一个位置。这次箭头追溯,也不是从右下角到左上角,而是从刚刚找到的最大值开始,追溯到没有箭头为止。追溯箭头终止的位置,也可以是箭头中的任何一个位置。最后根据标记好的箭头写出比对结果。
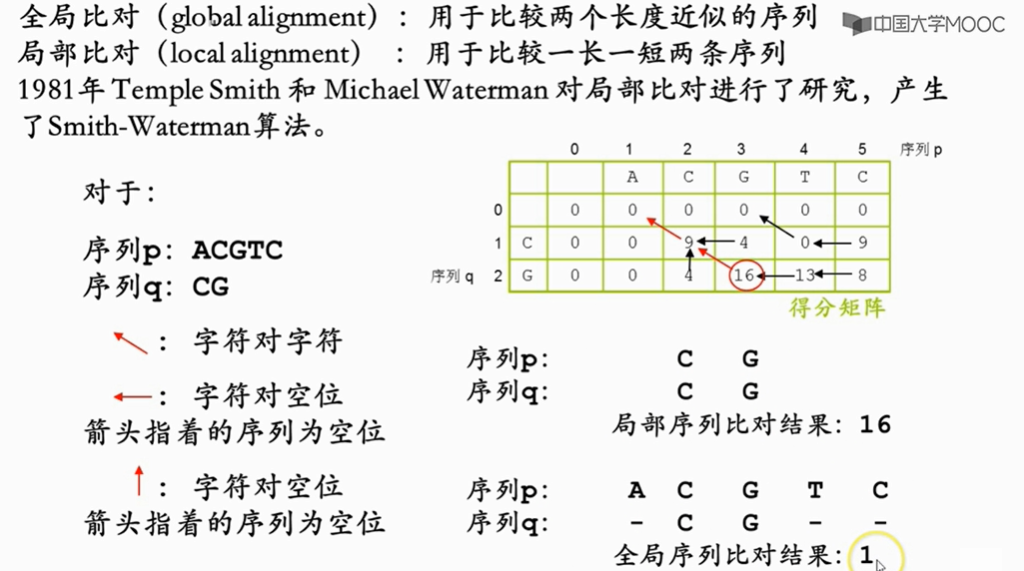
相对于两段序列的全局比对结果,局部比对,将他们的空位就基本全部忽略掉了。
3.5.4 一致度和相似度


全局比对的长度,不一定等于序列的长度。所以不管两条序列是否相同,都要对他们进行全局比。


第三章:序列比较 第二部分
3.7 在线双序列比对工具




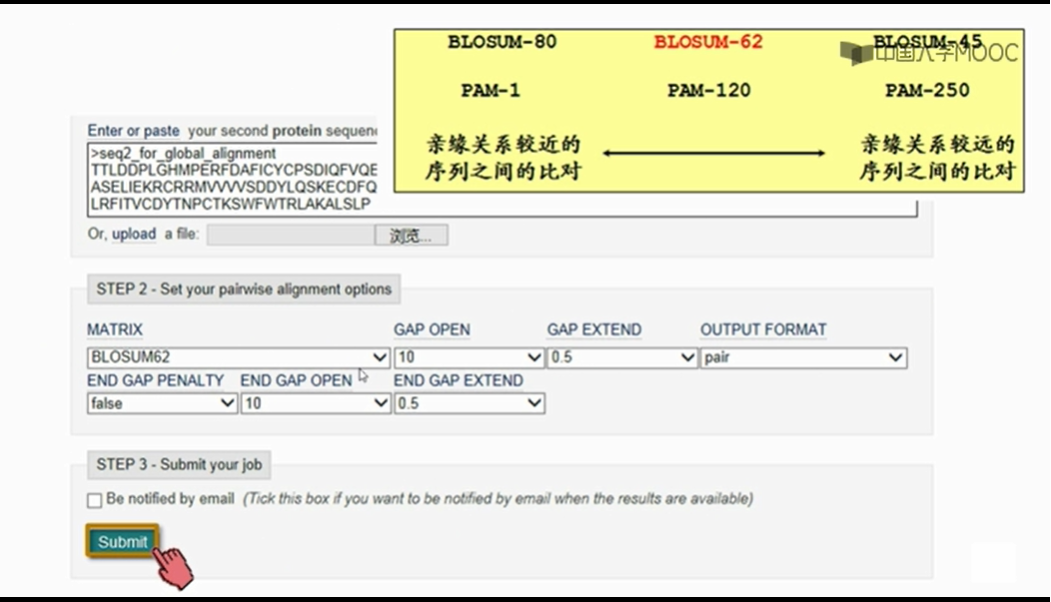
上节课说过怎么选替换计分矩阵,实在不知道怎么选,那就选BOLSUM-62。
GAP是空位罚分,显示10,代表计算的时候按照-10来处理。

序列必须长度
一致度
相似度
Gaps:空位比例
Score:比对
上下两个字母一样,用竖线表示
上下两个字母显示,用两个点表示,上下两个字母不相似用1个点表示。字母对空位的情况,用空格来表示。这样我们只要数一数比对结果里竖线的个数,再除以序列比对的长度就可以得到一致度。
竖线个数+两个点的个数除以比对的长度,就是相似度。
整个比对里一共插入了65个空位,占比对长度的33%左右。
序列两端的数,指的是序列的字母,在序列当中的位置数。
3.7.1 GAP的类型及分值设置

3.8 BLAST搜索
第三章:序列比较 第三部分
3.9 多序列比对介绍
3.10 在线多序列比对工具
3.11 多序列比对的编辑和发布
3.12 寻找保守区域
第四章:分子进化与系统发生
4.1 进化的故事
4.2基本概念
4.3系统发生树
4.4 系统发生树的构建
4.5 MEGA7构建NJ树
4.6课后甜品
第五章:蛋白质结构预测与分析(第一部分)
5.1蛋白质的结构
5.2 蛋白质的二级结构
5.3 蛋白质的三级结构
5.4 三级结构可视化软件VMD
第五章:蛋白质结构预测与分析(第二部分)
5.5计算方法预测三级结构
5.6 三级结构的比对
5.7 蛋白质分子表面性质
第五章:蛋白质结构预测与分析(第三部分)
5.8 获取蛋白质四级结构
5.9 蛋白质-蛋白质分子对接
5.10 蛋白质-小分子分子对接
5.11 虚拟筛选与反向对接
5.12 分子动力学模型
第六章:高通量测序技术及应用
6.1基因组学与预测技术
6.2 高通量测序技术在精准医学中的应用
6.3 生物信息学面临的挑战
6.4 从头测序
6.5 重测序
6.6 转录组测序
6.7 表观基因组学
6.8 猛犸象基因组测序计划
6.9 古基因组学面临的挑战
6.10 古基因组学研究中的生物信息技术
第七章:统计基础与序列算法
7.1贝叶斯公式机器生物学应用
7.2 二元预测的灵敏度和特异度
7.3 基本序列算法
第八章:数据挖掘
8.1 什么是数据挖掘
8.2 数据库系统
8.3机器学习
8.4weka
第九章:编程基础与网页制作(第一部分)
9.1 Linux操作系统
9.2 Linux基本命令
9.3Perl语言基础入门
9.4Perl语言基础高级
第九章:编程基础与网页制作(第二部分)
9.5 前端开发和HTML介绍
9.6 HTML常用标签
9.7 设计简单的网页
9.8 HTML与CGI简单交互
这篇关于生物信息学-山大精品课的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!