本文主要是介绍比较基因组学终将走向黄金时代,希望对大家解决编程问题提供一定的参考价值,需要的开发者们随着小编来一起学习吧!
- 背景介绍 -
始于1990年的人类基因组计划(The Human Genome Project)开启了基因组时代。随着测序技术的不断进步和测序成本的下降,越来越多的物种基因组被测序,从原核生物(流感嗜血杆菌,1995)到真核生物(酿酒酵母,1996),从单细胞生物到多细胞生物(秀丽线虫,1998;果蝇,2000),从陆生生物到水生生物(河豚鱼,2002),从动物到植物(拟南芥,2000),从小基因组生物到大基因组生物(人,2001;小鼠,2002;大鼠,2004)。大量基因组数据的深入研究,开阔了人类对物种进化及生命之树(地球上所有生命之间的进化关系网络,图1)认识的眼界,推动了物种间的比较基因组学的研究。
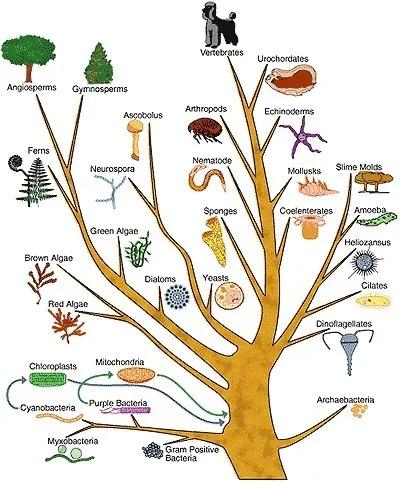
图1 | 生命之树(来源于网络图片)
比较基因组学是什么?
比较基因组,一般是指一个物种的多个个体基因组(群体)或多个物种基因组的结构和功能基因区域进行比较分析(Nobrega and Pennacchio 2004),本文主要指后者。具体来讲就是:比较多个物种的基因组结构特征的异同,研究物种间基因家族收缩与扩张,研究分化时间和演化关系,研究新基因的产生与进化等。其中,基因组的结构特征包括DNA序列、基因及基因家族、基因排序、调控序列和其它基因组结构标志等(图2)。
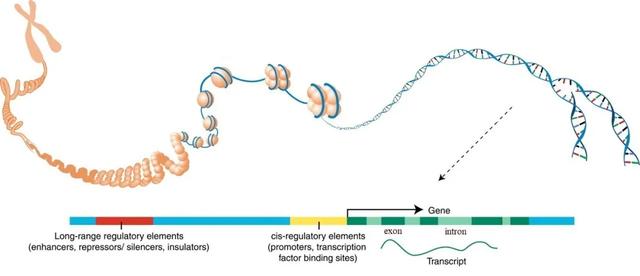
图2 | 基因组特征结构(修改网络图片)
比较基因组学研究基础是什么?
当物种A与物种B在从共同祖先分化的初期,两物种基因序列(内含子和外显子)都具有非常高的保守性(图3左)。但是随着两物种的分化时间越来越久远,由于碱基自发突变,两物种的基因序列会存在越来越多的变异。不同的是内含子序列由于不参与编码蛋白,其突变不改变蛋白序列,因此受选择压力较小,允许积累较多突变;而外显子区域由于负责编码蛋白,其突变导致蛋白序列和功能发生改变,特别是功能重要的看家基因,其外显子序列受到较强的负向选择,一般不容易积累突变,因而两物种的外显子区域仍然具有非常高的同源性(图3右)(Miller, Makova et al. 2004)。
比较基因组研究的基础正是基于物种基因序列的相似性,即物种间基因序列同源性越高,基因组共线性越好,说明物种的亲缘关系越近;相反,如果物种间基因序列变异越大,说明物种分化时间越久远,亲缘关系就越远。
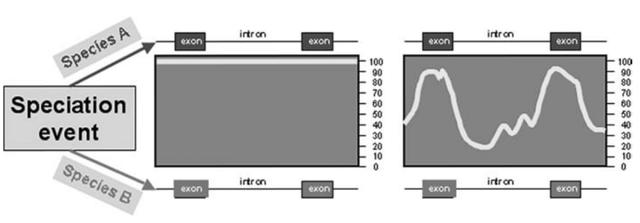
图3 | 基因编码和非编码序列在物种分化后因受到不同选择压力,具有不同保守性
研究比较基因组学的意义是什么?
通过比较在生命进化树中处于不同地位的物种的基因组结构特征,推测序列的共同祖先状态(common ancestral states),得到最近共同祖先序列(MRCA: most recent common ancestor)。通过研究物种演化过程中序列变异,了解物种分类谱系的动态变化过程,解析物种进化关系,为阐明物种进化历史提供依据(图4)(Kawashima,T. 2019)。同时,比较基因组对于解释基因功能,阐明其在适应性进化中的作用,鉴定基因型和表型之间的联系等方面也有重要意义(Alfoldi and Lindblad-Toh 2013)。
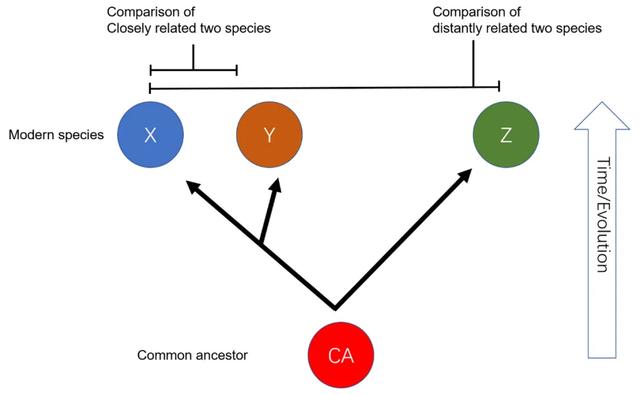
图4 | 比较基因组中物种生物学分类距离示意图
X、Y、Z为现存物种,都从共同祖先CA分化而来,其中X和Y的亲缘关系比X和Z的亲缘关系更近。当将X、Y和Z三个物种同时进行比较基因组分析时,可以推测其共同祖先CA的性状特征。
比较基因组分析主要包括哪些方面?
比较基因组学通过比较多物种蛋白序列或者基因位置与相对顺序,包括基因的丢失、复制、水平转移等,鉴定物种之间保守的基因(找物种相似之处),或者每个生物自身特征基因(找物种不同之处),旨在阐释物种多样性的分子遗传基础。其常见分析包括:基因家族聚类得到直系同源基因、系统进化分析、物种分歧时间估算、鉴定基因融合与基因簇、信号通路基因簇重构、基因家族收缩与扩张、全基因组共线性比较等(图5)(V. de Crécy-Lagard and Hanson, A. 2013)。

图5 | 比较基因组常见分析流程图
比较基因组物种的选择?
选择合适的物种是比较基因组分析的第一步,是成功挖掘生物学故事的关键,更是发高分文章的必要条件。那如何选择合适的物种呢?小编在这里列举些高分文章的实例,抛砖引玉,给老师们提供些思路。
案例1:比较基因组分析揭示黄麻纤维生物合成机制
研究人员首先获得两个高质量的锦葵科黄麻基因组,然后通过与其他13个物种,包括锦葵类(可可,棉花,拟南芥),豆类(亚麻,蓖麻,苜蓿,大豆,杨树),菊类(番茄和土豆),葡萄和单子叶水稻(外类群)等物种进行比较基因组分析,结合纤维特异表达的转录组数据,揭示了黄麻中纤维合成的分子机制(图6)(Islam, Saito et al. 2017)。
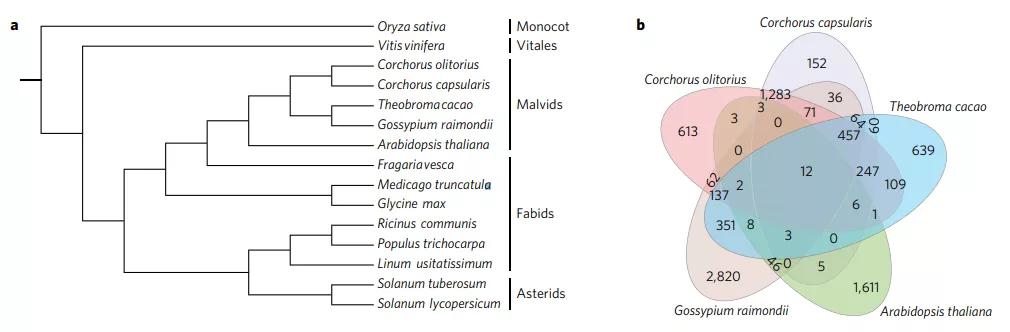
图6 | 黄麻系统进化树
案例2:比较基因组分析揭示大熊猫(Giant panda)和小浣熊(Red panda)都爱吃竹子的趋同进化机制
虽然大熊猫和小浣熊名字中都带“熊”字,但它们实际上是远亲。通过将小浣熊、大熊猫、白釉、北极熊、狗、老虎、人和老鼠等物种进行比较基因组分析发现,小浣熊与白釉亲缘关系相对较近,位于进化树的姐妹支;而大熊猫与北极熊亲缘关系相对较近(图7A)。虽然是远亲,小浣熊却表现出与大熊猫相同的食性,都爱吃竹子。这种两个亲缘关系较远的生物独立发展出相似特性的现象被称为趋同进化。通过比较基因组分析,研究人员发现两个基因PCNT 和DYNC2H,在两个物种的四肢发育中是重要的,允许手腕的骨头额外增长成拇指状附属物(伪拇指的第六指)来帮助它们进食,有助于把握竹竿(图7B)(Hu, Wu et al. 2017)。
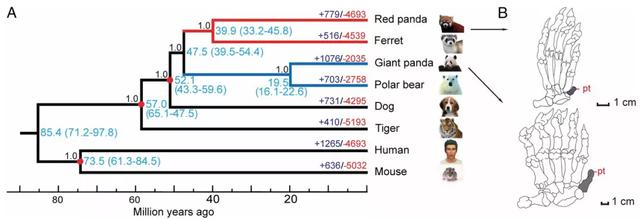
图7 | 小浣熊和大熊猫的系统进化树分析和伪拇指的趋同表型
案例3:芡实和金鱼藻基因组揭示被子植物早期进化
两种水生植物-芡实和金鱼藻通过与其它代表性陆生植物基因组包括真双子叶植物(拟南芥、桃子、葡萄和耧斗菜),木兰类植物(牛油果、牛樟和鹅掌楸),单子叶植物(水稻、小果野焦和蝴蝶兰),无油樟和裸子植物银杏,进行深入比较分析,揭示了被子植物在生命之树中关键进化地位:无油樟和睡莲是其他所有被子植物的姐妹群;而金鱼藻是双子叶植物的姐妹群(图8)(Yang, Sun et al. 2020)。
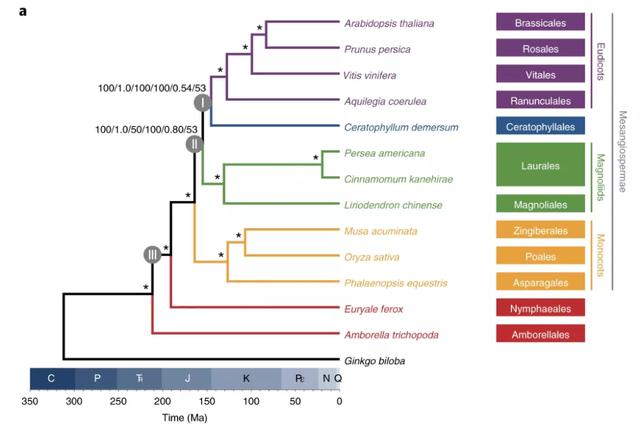
图8 | 芡实和金鱼藻系统进化树
从以上比较基因组研究论文中可以看出,物种的选取与阐述的生物学故事紧密相关。在此,小编依据对文献的理解和项目分析的经验,大胆提出针对不同的项目需求,采用不同的原则选取物种:
- 如阐释物种特异的生物学特性,即找物种之间差异或不同,比如案例1,要采取“相近相异”原则,即选取亲缘关系较近,但生物学特性不同的物种。
- 如阐释物种共有的生物学特性,即找物种间相似性,比如案例2,要采取“相远相同”原则,即选取物种亲缘关系相对较远,但具有相似生物学特性的物种。
- 如阐释物种的系统发育关系,则采取“跨度合适”的原则,即选取生命之树上跨度合适的物种。
当然,比较基因组分析还可以应用在其他很多方面,选取物种的原则也因具体研究而异。
- 结束语 -
高质量基因组的不断涌现,使得比较基因组的应用越来越广泛和深入,也是研究者挖掘生物学问题和解释生物学现象的有力工具。欧易生物拥有专业的动植物基因组研发团队,为客户提供从PacBio三代测序、de novo组装、基因注释、基础分析,到比较基因组、个性化分析及多组学联合分析等高质量的技术服务,多名博士级专业人员,经过多年经验沉淀积累,具备多角度分析问题能力,提供多元化项目技术服务,多组学技术平台联合支撑,极力打造一站式服务。欢迎登陆欧易生物官网或来电咨询。
参考文献
Alfoldi, J. and K. Lindblad-Toh (2013). "Comparative genomics as a tool to understand evolution and disease." Genome Res 23(7): 1063-1068.
Hu, Y., Q. Wu, S. Ma, T. Ma, L. Shan, X. Wang, Y. Nie, Z. Ning, L. Yan, Y. Xiu and F. Wei (2017). "Comparative genomics reveals convergent evolution between the bamboo-eating giant and red pandas." Proc Natl Acad Sci U S A 114(5): 1081-1086.
Islam, M. S., J. A. Saito, E. M. Emdad, B. Ahmed, M. M. Islam, A. Halim, Q. M. Hossen, M. Z. Hossain, R. Ahmed, M. S. Hossain, S. M. Kabir, M. S. Khan, M. M. Khan, R. Hasan, N. Aktar, U. Honi, R. Islam, M. M. Rashid, X. Wan, S. Hou, T. Haque, M. S. Azam, M. M. Moosa, S. M. Elias, A. M. Hasan, N. Mahmood, M. Shafiuddin, S. Shahid, N. S. Shommu, S. Jahan, S. Roy, A. Chowdhury, A. I. Akhand, G. M. Nisho, K. S. Uddin, T. Rabeya, S. M. Hoque, A. R. Snigdha, S. Mortoza, S. A. Matin, M. K. Islam, M. Z. Lashkar, M. Zaman, A. Yuryev, M. K. Uddin, M. S. Rahman, M. S. Haque, M. M. Alam, H. Khan and M. Alam (2017). "Comparative genomics of two jute species and insight into fibre biogenesis." Nat Plants 3: 16223.
Miller, W., K. D. Makova, A. Nekrutenko and R. C. Hardison (2004). "Comparative genomics." Annu Rev Genomics Hum Genet 5: 15-56.
Kawashima T, (2019). “Comparative and Evolutionary Genomics.” Encyclopedia of Bioinformatics and Computational Biology.
Nobrega, M. A. and L. A. Pennacchio (2004). "Comparative genomic analysis as a tool for biological discovery." J Physiol 554(Pt 1): 31-39.
V. de Crécy-Lagard, Hanson, A. (2013) “Comparative Genomics and Genome Evolution.” Brenner's Encyclopedia of Genetics (Second Edition).
Yang, Y., P. Sun, L. Lv, D. Wang, D. Ru, Y. Li, T. Ma, L. Zhang, X. Shen, F. Meng, B. Jiao, L. Shan, M. Liu, Q. Wang, Z. Qin, Z. Xi, X. Wang, C. C. Davis and J. Liu (2020). "Prickly waterlily and rigid hornwort genomes shed light on early angiosperm evolution." Nature Plants 6(3): 215-222
这篇关于比较基因组学终将走向黄金时代的文章就介绍到这儿,希望我们推荐的文章对编程师们有所帮助!







